CRISPR-Cas9実験を成功させるために、実験条件が最適化されていることを確認します。
Edit-R synthetic sgRNA(化学合成sgRNA)
-
ポジティブコントロールと検出プライマー
十分に特徴付けられた遺伝子を標的とする種特異的な化学合成sgRNA、およびミスマッチ検出アッセイプライマーを使用して、ゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。
-
non-targetingコントロール
標的遺伝子特異的sgRNAの非存在下でCRISPR-Cas9コンポーネントに対するベースラインの細胞応答を評価するために使用します。
-
Cutting controls
Cutting control (safe harbor) synthetic sgRNAs recommended for determination of baseline cellular responses in CRISPR-Cas9 experiments.
Edit-R synthetic crRNA(化学合成crRNA)
-
ポジティブコントロールと検出プライマー
十分に特徴付けられた遺伝子を標的とする種特異的な化学合成crRNA、およびミスマッチ検出アッセイプライマーを使用して、ゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。
-
non-targetingコントロール
標的遺伝子特異的crRNAの非存在下でCRISPR-Cas9コンポーネントに対するベースラインの細胞応答を評価するために使用します。
-
Cutting controls
Cutting control (safe harbor) crRNAs recommended for determination of baseline cellular responses in CRISPR-Cas9 experiments.
Edit-R all-in-one lentiviral sgRNA
-
ポジティブコントロールと検出プライマー
all-in-one lentiviral sgRNA用コントロールを使用して、DNA二本鎖切断とゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。
-
non-targetingコントロール
all-in-one lentiviral sgRNAコンストラクトは、ヒト、マウス、またはラットのゲノム内の遺伝子を標的としないようにバイオインフォマティクスによって設計および検証されています。
Edit-R lentiviral sgRNA
-
ポジティブコントロールと検出プライマー
十分に特徴付けられた遺伝子を標的とする種特異的な化学合成sgRNA、およびミスマッチ検出アッセイプライマーを使用して、ゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。
-
non-targetingコントロール
標的遺伝子特異的sgRNAの非存在下でCRISPR-Cas9コンポーネントに対するベースラインの細胞応答を評価するために使用します。
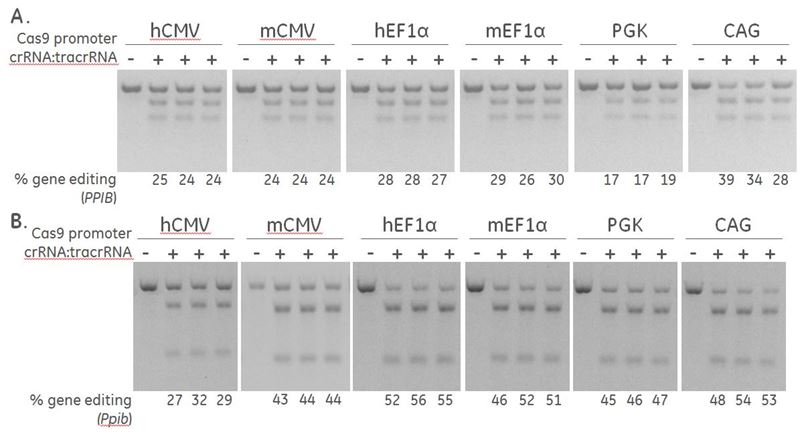
CRISPR-Cas9ゲノム編集実験における効果的なコントロールの重要性
CRISPR-Cas9ゲノム編集システムでは、効果的な遺伝子ノックアウトのために複数のコンポーネント(ガイドRNAとCas9ヌクレアーゼ)を細胞に導入する必要があります。いずれかのコンポーネントの量が不十分な場合、ゲノム編集が不十分になります。
DNAミスマッチ検出アッセイにおいて検証済みの検出プライマーを使用した生物種特異的ポジティブコントロールを使用することで、CRISPR-Cas9ゲノム編集成功させるための実験条件の妥当性を検証し、また継続して行われる実験における優れたトランスフェクションのインジケーターとして機能します。
数種類のnon-targetingコントロールをラインアップしており、潜在的な非特異的効果の評価を可能にし、細胞タイプやアッセイに最適なネガティブコントロールを選択することができます。
Horizonの検出プライマーを使用したDNAミスマッチ検出アッセイ(SURVEYOR®またはT7EI)は、クローン細胞またはクローン化前のプール細胞における遺伝子ノックアウトの結果を評価するための比較的高速でハイスループットな方法です。それらは挿入と欠失の相対的なパーセンテージしか評価できませんが(機能的なタンパク質ノックアウトではありません)、次の場合に非常に役立ちます。
- crRNA:tracrRNAトランスフェクション条件の最適化
- Cas9ヌクレアーゼ発現を駆動するための最適なプロモーターの決定