Add any of our CRISPR portfolio reagents (CRISPRko, CRISPRa, CRISPRi) to your cart to see new pricing (up to 56% off) now including All-in-one lentiviral sgRNA formats in 2026
遺伝子発現調節ツールであるDharmacon™ CRISPRmodプラットフォームは、DNA を切断することなく、標的遺伝子の転写を活性化(CRISPRa)または抑制(CRISPRi)します。
CRISPRmodとは?
CRISPRmod (CRISPR modulation)は、標的とする内在性遺伝子の発現調節に使用される、CRISPR activationおよびCRISPR interference試薬から成るDharmaconファミリーです。
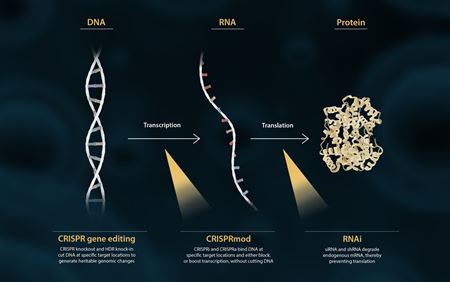
分子生物学のセントラルドグマでは、DNAはRNAに転写され、次にタンパク質に翻訳されます(下図)。研究者は、遺伝子の機能をよりよく理解するために、機能喪失または機能獲得実験を通じて、この経路を複数のポイントで調節することができます。
既存のCRISPRノックアウトおよびHDRノックイン技術は、研究者に細胞内のDNAを編集(継承可能な変更)することを可能にしました。一方、従来のRNAi技術ではmRNAが分解され、翻訳が妨げられます。
CRISPRmodは、転写レベルで遺伝子発現を調節する新しい方法を提供します。Dharmacon CRISPRmodポートフォリオには、ターゲット遺伝子発現をそれぞれアップレギュレートまたはダウンレギュレートするために、CRISPRaシステムとCRISPRiシステムの両方があります。
CRISPRmodはどのように機能しますか?
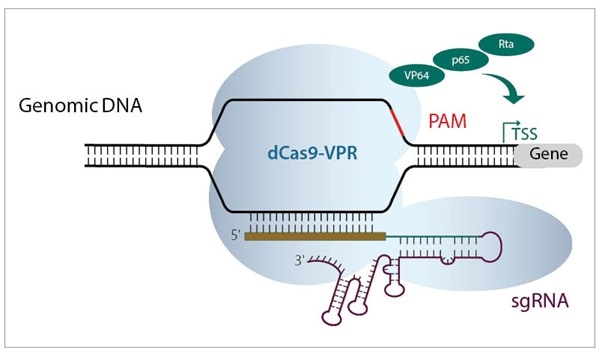
2つのCRISPRmodシステム(CRISPRaおよびCRISPRi)は、活性化因子または抑制因子のいずれかに融合した非活性化Cas9コンストラクトを使用して、転写調節のためのPAMアンカー型ターゲティングの力を活用します(figure 2)。 CRISPRmodは、遺伝子発現調節にCRISPRの精度をもたらします。
ネイティブのCas9は、RuvCIおよびHNHヌクレアーゼドメインの点突然変異(W. Cheng et al.)によってDNA切断機能が無くなります。これをdCas9(deactivated Cas9または dead Cas9)と呼びます。このdCas9タンパク質の C末端に、アクチベーターまたはリプレッサードメインを融合させることにより、標的遺伝子の転写調節を行うように設計しました。
CRISPRa システム |
CRISPRi システム |
 |
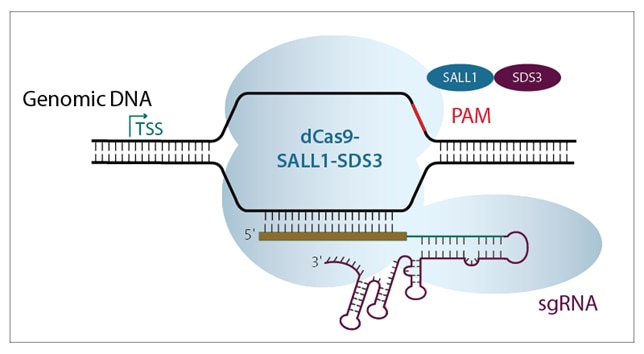 |
Figure 2. 転写を活性化するためにTSSの上流に結合するCRISPRa機構(左)、および転写をブロックするために標的遺伝子TSSの下流に結合するCRISPRi機構(右)を示す概略図
CRISPRmodを使用する理由は?
CRISPRmodは、内在性遺伝子調節のために、新世代のCRISPRベースの実験を可能にします。CRISPRmodは、他の遺伝子調節法から観察された表現型を確認するための直交検証としても使用できます。
- より生物学的に関連性のあるモデルのために、ネイティブコンテキスト内で遺伝子を活性化または抑制します。
- 複数の遺伝子を同時に調節して、パスウェイまたはネットワークをよりよく理解できます。
- より堅牢なデータセットのために、CRISPRノックアウトまたはsiRNAの結果を直交的に検証できます。
マルチプレックス遺伝子発現調節
CRISPRmodは、複数の遺伝子の同時発現調節のためのユニークな方法です。各ガイドRNAは、その特定のDNA標的に独立して結合し、外から供給されるdCas9融合タンパク質に依存します。そのため、内在性経路の競合を最小限に抑えます。ここでは、iPSC細胞内でのマルチプレックスCRISPRiの有効性を示します(figure 3)。
化学合成sgRNAは、複数遺伝子の同時抑制の簡便なマルチプレックスを可能にする
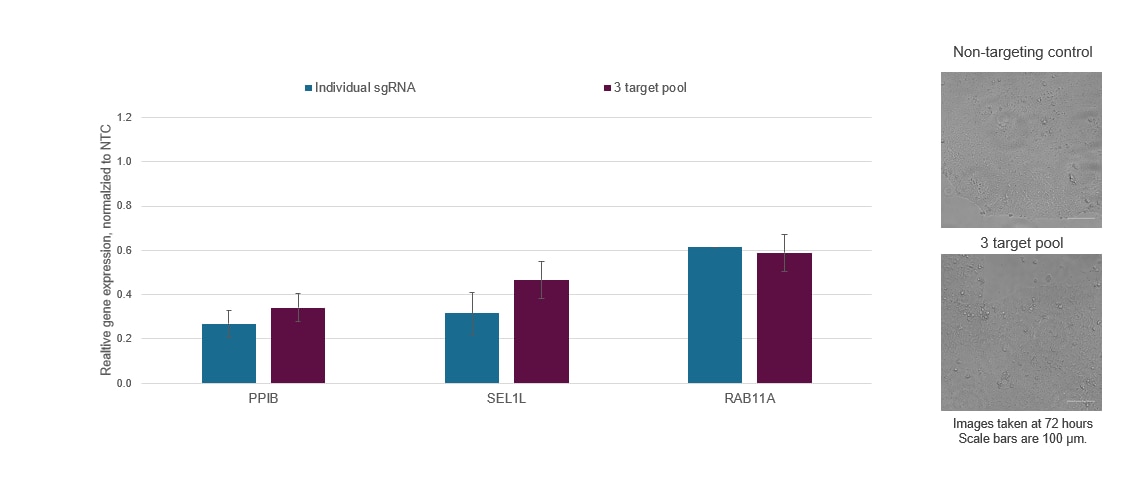
Figure 3. 個々のCRISPRi sgRNAを単一の試薬にプールして、標的遺伝子の抑制を強化したり、マルチプレックス化して複数の遺伝子の同時抑制を可能にしたりすることができます。dCas9-SALL1-SDS3を安定して発現するWTC-11ヒトiPS細胞に、Lonza 96ウェルシャトルシステムを介して、PPIB、SEL1L、およびRAB11Aを標的とする化学合成sgRNAを導入しました。各遺伝子ターゲットに対して最も有効なデザイン済みsgRNAが選択され、個別に、またはガイドあたり3 µMの濃度でマルチターゲットプールの一部として使用されました。ヌクレオフェクションの72時間後に細胞を回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各標的遺伝子の相対的発現は、ACTBをハウスキーピング遺伝子として使用するΔΔCq法で計算し、non-targeting control(NTC)に対して正規化しました。3つの遺伝子は、発現抑制の程度を大幅に減少したり、細胞の生存率と形態を著しく変化したりすることなく、同時に発現抑制されました。
直交検証
検証された研究は良い研究であると言えます。Dharmacon試薬は、細胞のパスウェイを調べる幅広いツールを含み、DNAレベルでの遺伝子の不活性化、RNA転写の調節、または成熟mRNA転写物の分解を可能にします。Horizonは研究者の皆様のアプリケーションにおいて、遺伝子の活性化または不活性化の最良の方法の選択をお手伝いいたします。さらには、堅牢でエレガントな検証済みの研究のために、2つの方法をご提案します。
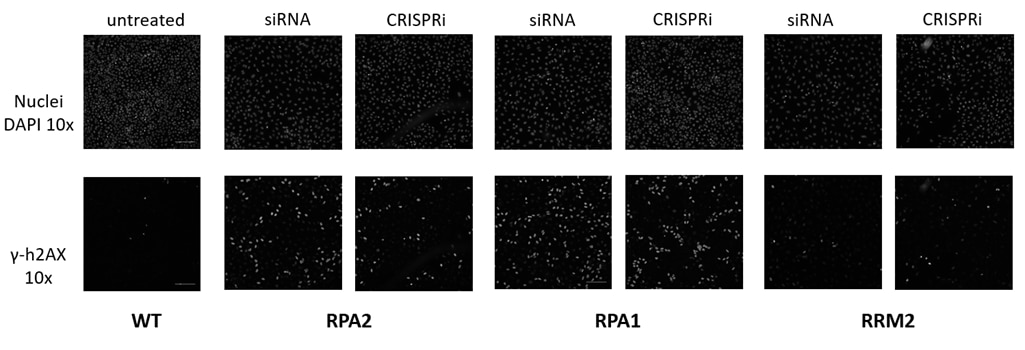
CRISPRmodは、RNAiまたはCRISPRノックアウトスクリーニング後のフォローアップ研究に理想的です(figure 4)。ここでは、CRISPRiを使用して、アレイ化siRNAスクリーニングの実験結果を更に調査しました。
化学合成CRISPRi試薬を用いて、siRNAスクリーニングからのヒットを直交的に検証する
Figure 4. 損傷応答アッセイ。二本鎖切断を取り巻くh4AX(コアヒストン複合体の構成要素)は急速にリン酸化され、DNA損傷シグナル伝達ネットワークを介して一連の応答を生成します。DNA損傷経路に重要なタンパク質をノックダウンすると、修復されていない二本鎖DNA切断が蓄積し、リン酸化h4AX(γ-h4AX)が増加します。以前に実施したRNAiスクリーニングからのヒットを、CRISPRi試薬を使用して直交的に検証しました。hEF1αプロモーター下でdCas9-SALL1-SDS3を構成的に発現するU2OS細胞に、DharmaFECT 4トランスフェクション試薬を使用して、RPA2、RPA1、またはRRM2をターゲットとするプール化sgRNA(50nM)またはON-TARGETplus SMARTpool(50nM)をトランスフェクトしました。トランスフェクションの72時間後、細胞を固定し、抗リン酸化h4AX抗体で染色し、ヘキスト染色を使用して核を同定しました。複製プレートを回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各標的遺伝子の相対的発現は、ハウスキーピング遺伝子としてGAPDHを使用するΔΔCq法で計算され、non-targetingコントロール(NTC)に対して正規化しました。
CRISPRmod製品プラットフォーム
遺伝子転写抑制(CRISPRi)と遺伝子活性化(CRISPRa)の両方に対応するCRISPRmod試薬システムを提供しています。これには、エレクトロポレーションまたはトランスフェクションを容易にし、迅速な結果を得るための化学合成ガイドとmRNAオプション、およびトランスフェクションが困難な細胞や長期経過実験のためのレンチウイルスオプションが含まれます。リンクよりそれぞれの製品ページを参照して、研究に最適な製品フォーマットをご確認ください。ご不明な場合はお問い合わせください。
CRISPRi |
CRISPRa |
|
|---|---|---|
用途 |
内在性遺伝子発現の転写抑制 | 内在性遺伝子発現の転写活性化 |
dCas9タンパク質 |
dCas9-SALL1-SDS3 | dCas9-VPR |
ガイド |
CRISPRi ガイドRNA | CRISPRa ガイドRNA |
| CRISPRi products | CRISPRa products |
Order Products
CRISPR activation試薬
CRISPRa実験条件には多くのオプションがあります。こちらから試薬をご検討ください。
CRISPR interference試薬
CRISPRi実験条件には多くのオプションがあります。こちらから試薬をご検討ください。
CRISPRmodコントロール
機能喪失実験用のCRISPRiコントロール
Helpful Resources
CRISPRaおよびCRISPRiとは?
遺伝子の転写活性化と転写抑制のためのCRISPR-Cas9ベース技術
CRISPRmod Reading List
Horizon CRISPRaまたはCRISPRiを使用した文献リスト