CRISPRaシステム
Dharmacon™ CRISPRmod CRISPRaシステムには、2つのコンポーネント(アクティベータータンパク質(VPR)に融合した特殊なdCas9と、遺伝子の転写開始部位(TSS)のすぐ上流の領域をターゲットにするように特別に設計されたガイドRNA)が必要です。ガイドRNAはdCas9-VPRと複合体を形成し、アクティベーター複合体をDNAターゲットサイトに誘導します(figure 1)。
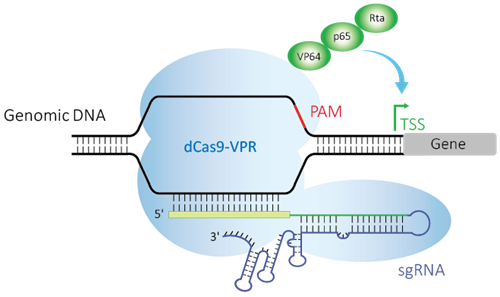
Figure 1. Diagram of dCas9-VPR with sgRNA targeting a gene’s promoter region
dCas9-VPR
CRISPR-Cas9ベースのCRISPRaシステムでは、ネイティブのCas9は、RuvCIおよびHNHヌクレアーゼドメインの点突然変異によってDNA切断機能が消失しています。これらの変異は、Cas9タンパク質をdCas9(deactivated Cas9または dead Cas9)に変化させます。このdCas9タンパク質は、さまざまな転写活性化因子を融合することによってさらに改変されます。
HorizonのCRISPRaシステムは、汎用されているdCas9-VPRアクティベーションシステムに基づいています。SAMなどの他の転写活性化戦略は、こちらでご確認ください。dCas9-VPRシステムは、dCas9がC末端で3つの転写活性化因子(VP64、p65、およびRta)(参考文献1〜7)に融合していることにより機能します。
CRISPRaガイドRNA
CRISPRaガイドRNAは、標的遺伝子のプロモーター領域または転写開始部位(TSS)の上流にある配列を標的にして転写活性化をもたらします(参考文献3, 5)。機能的なガイドRNAの設計は困難な場合があります。なぜなら、TSSは常に十分な注釈が付けられているとは限らず、また、他のタンパク質因子のためにアクセスできないこともあるためです。HorizonのCRISPRaガイドRNAのデザインは、機械ラーニング技術を介して開発された公開済みのCRISPRa v2アルゴリズムを使用しています(8)。アルゴリズムは、FANTOMおよびEnsemblデータベースを使用してTSSを予測し、クロマチン、位置、および配列データを組み込んで、非常に効果的なガイドRNA設計を予測しました。遺伝子に代替TSS(遺伝子の6.8%)がある場合、少数の遺伝子(0.1%)に3つ以上のTSSがある場合は追加の課題が発生します。Horizonは、TSSごとに10個のガイドRNAデザインを、予測される機能の順にランク付けしてリストしています。
HorizonのCRISPRaガイドRNAは、化学合成crRNAまたは発現sgRNAフォーマットのいずれかとしてご提供しています。
混合物(プール)によるCRISPRa効率の向上
複数のCRISPRaガイドRNAデザインの混合物(プール)を作成すると、遺伝子転写活性化の増加(Figure 2a)、または最も機能的な個別のindividualフォーマットのcrRNAと同等の遺伝子転写活性化(Figure 2b)のいずれかが生じることがわかっています。
公開済みのアルゴリズムで設計されたガイドRNA間のオーバーラップを調べたところ、多くの遺伝子(> 70%)で、ガイドRNAのターゲットサイトのオーバーラップが、ほとんどまたは全くないことがわかりました。これらの遺伝子の場合、通常、gRNAをプールすると転写活性化レベルが上昇します。TSSでは、遺伝子の一部のサブセット(〜12%)のみが4つのデザインすべてと重複しています。これらの遺伝子について、プールフォーマットであれば、一般的に、最も効果的なgRNAと同様に機能することがわかりました。概して、プールフォーマットは最大の遺伝子転写活性化を促進するための優れた戦略と言えます。プールフォーマットは、たとえば、プレートを用いて複数の遺伝子の分析を実行する場合など、実験の規模を縮小するのにも役立ちます。
プールフォーマットのcrRNAでは遺伝子転写活性化が促進される
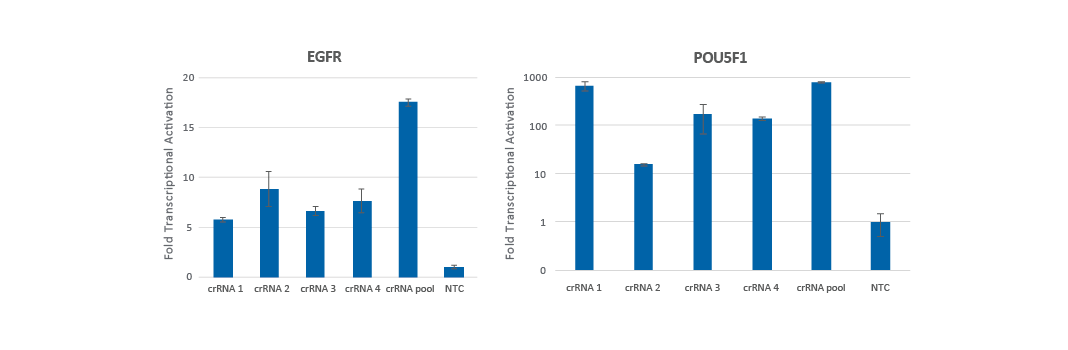
Figure 2. dCas9-VPRを安定して発現するU2OS細胞を10,000細胞/ウェルでプレーティングし、EGFR(A)またはPOU5F1(B)を標的とするCRISPRa化学合成crRNA:tracrRNA(25 nM)をDharmaFECT 4トランスフェクション試薬を使用してトランスフェクトしました。デザイン済みcrRNAは、個別のindividualフォーマット、またはプールフォーマット(合計濃度25 nMまで)で使用しました。トランスフェクションの72時間後に細胞を回収し、RT-qPCRを使用して相対的な遺伝子発現を計算しました。各遺伝子の相対的発現は、ハウスキーピング遺伝子としてGAPDHを使用してΔΔCq法で計算し、non-targetingコントロールに対して正規化しました。
sgRNA is as effective as crRNA:tracr for CRISPR activation
Robust CRISPRa activation can be achieved in multiple formats. Figure 4 shows that crRNA:tracrRNAs work similarly to sgRNA pools for activation of multiple gene targets.
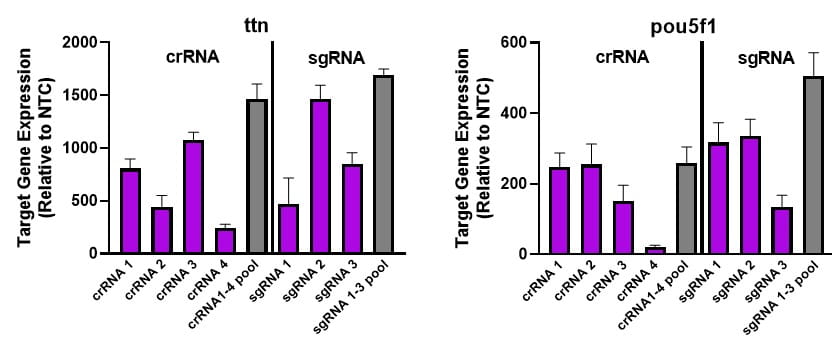
Figure 3.NIH-3T3 cells stably expressing integrated CRISPRa dCas9-VPR (driven by mCMV promoter) were plated at 10,000 cells/well and transfected using DharmaFECT 1 Transfection Reagent (0.2 uL/well) with CRISPRa synthetic single guide RNA (sgRNA) targeting ttn or pou5f1 genes. CRISPRa sgRNAs were used either individually or pooled (to an individual or pooled concentration of 25 nM). Gene expression was assessed using RT-qPCR as described in Figure 2.
遺伝子転写活性化の確認
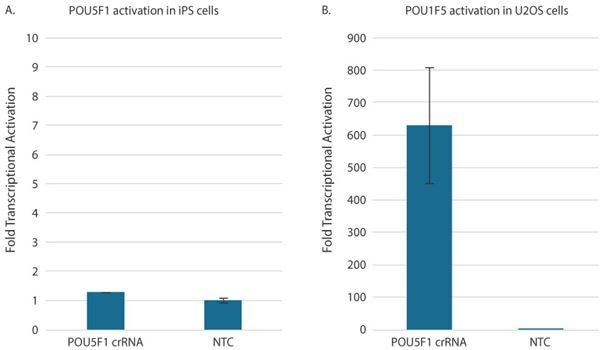
RT-qPCR、ウエスタンブロット、または免疫蛍光分析など、遺伝子の転写活性化を確認する方法はたくさんあります。通常、non-targetingコントロールとCRISPRaガイドRNAで処理されたサンプル間のターゲット遺伝子発現レベルの相対的変化を測定する最も速くて簡単な方法はRT-qPCRです。RT-qPCR分析は、SYBRグリーン法またはプローブベースの遺伝子発現アッセイのいずれかで機能します。遺伝子転写活性化のためにRT-qPCRを実行する際に注意すべきことの1つは、CRISPRaの前に遺伝子発現レベルが検出されない可能性があることです。この場合、ΔΔCq分析法を使用する場合、計算を実行するにはゼロ以外の値が必要であるため、qPCR機器の検出限界を表す任意の値が「検出不能」のプレースホルダーとして使用されます。ほとんどの場合、この値は、プログラムされたサイクル数と機器のCq決定方法に応じて、35〜40になります。多くのプロトコルでは、標準のqPCRサイクリング条件に追加のサイクル(合計で最大45)を追加することを推奨しています。CRISPRa実験を設計する際の重要な考慮事項は、遺伝子転写活性化のレベルがそれぞれの細胞タイプの基本的なターゲット発現レベルと相関することです。基礎発現の低い遺伝子ではより高い活性化(100〜10,000倍)が観察される可能性がありますが、中程度から高度に発現する遺伝子では活性化の倍数が低くなります(通常は100倍未満)。CRISPRaを実行する前に、目的の細胞株の基礎発現レベルを決定して、達成できる転写活性化に対する期待値を設定することは価値があります。例えば、遺伝子POU5F1は、iPSCのような多能性細胞で高度に発現する転写因子(OCT4)をコードしますが、細胞が分化すると通常は低発現します。POU5F1は、ポジティブコントロールとして推奨される遺伝子の1つですが、幹細胞を扱う場合、これは論理的な選択ではありません。ここでは、iPSC(Figure 3A)およびU2OS-dCas9-VPR導入細胞集団(骨肉腫細胞株)(Figure 3B)におけるPOU5F1の相対的な遺伝子転写活性化を示します。予想通り、iPS細胞ではPOU5F1の転写活性化は低い(〜1.2倍)が、骨肉腫細胞では高い転写活性化(〜600倍)が見られます。
CRISPRa実験のための考慮事項
CRISPRaの実験条件には多くのオプションと考慮事項があります。一般に、ユーザーは、dCas9-VPR安定発現細胞集団で作業するときに、最も強力な遺伝子転写活性化を得ることができます。長時間のアッセイ後に測定する場合(96時間以上)では、多くの研究者がレンチウイルスsgRNAを選択します。短時間のアッセイ(96時間未満)の場合、化学合成crRNA:tracrRNAまたはプラスミドsgRNAは同等の遺伝子転写活性化をもたらします。
| dCas9-VPR source | Guide RNA format | Delivery method | Recommendations for use & benefits |
|---|---|---|---|
|
CRISPRa dCas9-VPR stable cells or CRISPRa dCas9-VPR lentiviral particle transduction |
CRISPRa synthetic sgRNA | Transfection or electroporation |
|
| CRISPRa synthetic crRNA + tracrRNA | |||
| CRISPRa lentiviral sgRNA particles | Transduction |
|
|
| CRISPRa dCas9-VPR mRNA | CRISPRa synthetic sgRNA | Co-transfection or electroporation |
|
| CRISPRa synthetic crRNA + tracrRNA | |||
| CRISPRa dCas9-VPR lentiviral plasmid | CRISPRa lentiviral sgRNA plasmid | Co-transfection or electroporation with dCas9-VPR plasmid |
|
| CRISPRa all-in-one dCas9-VPR + sgRNA | Transduction |
|
|
CRISPRa製品プラットフォーム
Horizonは、遺伝子転写活性化実験にCRISPRaを使用するための複数の製品をご用意しています。
CRISPRa dCas9-VPR:
- dCas9-VPR安定発現細胞株から始めることができます。
- 安定発現細胞株を作製するための、プロモーターを選択できる発現ベクターは、精製プラスミドまたはレンチウイルス粒子をご用意しています。
- DNA-freeの実験の場合は、dCas9-VPR mRNAを使用します(濃縮のためのEGFPまたはピューロマイシンマーカーを選択できます)。
CRISPRaガイドRNA:
すべてのヒトおよびマウス遺伝子に対するCRISPRa用デザイン済み化学合成crRNAまたはlentiviral sgRNAを提供しています。
CRISPRa化学合成crRNAは、個別のindividual、set-of-4、またはプール(上位4つのデザインの等モル混合物)フォーマットのcrRNA試薬が用意されています。これらは、Cherry-pickカスタムライブラリー デザインツールを使用してチューブまたはマルチウェルプレートでお届けすることもできます。このガイドフォーマットは、化学合成 tracrRNAとともに使用する必要があります。
CRISPRaレンチウイルスsgRNAは、精製レンチウイルス粒子またはグリセロールストックのいずれかで、個別のcrRNA試薬またはset-of-4フォーマットが用意されています。
デザイン済みCRISPRaガイドRNA製品は、Horlbeck et al.の v2アルゴリズムによるスコアリングガイドRNAデザインの上位4つを表しています。該当する場合、他のTSS用の追加の4つのガイドRNAは、それぞれP1およびP2としてラベル付けされます。遺伝子のCRISPRaガイドRNAオプションにP1とP2の両方の指定がある場合は、実験のために両方をテストすることが有益な場合があります。遺伝子のCRISPRaガイドRNAにP1またはP2ラベルがない場合、単一の開始部位のみが定義されます。2つ以上のTSSが同定された少数の遺伝子については、カタログアイテムとしてP1とP2のみを提供しますが、カスタムリクエストとして公開されたアルゴリズム(8)の設計に基づいて任意のガイドRNAを製造できます。
References
- L. A. Gilbert et al., CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154, 442–451 (2013).
- W. Cheng et al., Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23, 1163–1171 (2013).
- L. A. Gilbert et al., Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159, 647–661 (2014).
- M. E. Tanenbaum, L. A. Gilbert, L. S. Qi, J. S. Weissman, R. D. Vale, A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159, 635–646 (2014).
- S. Konermann et al., Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517, 583–588 (2015).
- Chavez et al., Highly efficient Cas9-mediated transcriptional programming. Nat. Methods. 12, 326–328 (2015).
- J. G. Zalatan et al., Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160, 339–350 (2015).
- M. A. Horlbeck et al., Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760 (2016).
- Chavez et al., Comparison of Cas9 activators in multiple species. Nat Methods. 7, 563–567 (2016)
Order products
CRISPRaガイドRNA
あらゆるヒト遺伝子の特異性の高い転写活性化のための化学合成、およびレンチウイルス発現CRISPRa crRNA
'CRISPRa-ready'のdCas9-VPRを発現する安定細胞株
さまざまな汎用細胞株のバックグラウンドから選択してください。CRISPRaガイドRNAをトランスフェクトするだけで、CRISPRa遺伝子転写活性化実験を開始できます。
CRISPRmod Controls
Positive and non-targeting controls to ensure your CRISPRi or CRISPRa experimental conditions
dCas9-VPR試薬
あらゆるCRISPRaワークフローソリューションのための精製dCas9-VPR mRNA、またはレンチウイルス粒子をご用意しています。
