CRISPR-Casテクノロジーとは?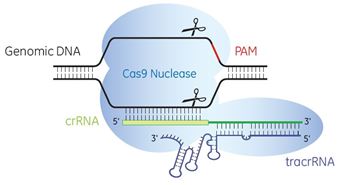
短いRNAが、真核生物の複雑なゲノム内のターゲットにヌクレアーゼ・タンパク質を高い特異性で誘導するために使用されます。一般にCRISPR-Cas(Clustered Regularly Interspersed Short Palindromic Repeats)技術として知られるこれらのシステムは、CRISPR関連(CRISPR-associated: Cas)タンパク質に依存しており、ゲノム編集機能や遺伝子発現を変化させる可能性があります。CRISPR-Cas9ゲノム編集システムの構成要素は、様々なゲノム編集アプリケーションのために複数の方法で組み合わせることができます。
CRISPR-Casシステムではどのような応用が可能か?
遺伝子ノックアウト
Cas9エンドヌクレアーゼは、真核生物系におけるゲノム編集のための汎用されるツールとなっています1-3)。標的特異的CRISPR RNA(crRNA)とトランス活性化crRNA(trans-activating crRNA: tracrRNA)、またはシングルガイドRNA(sgRNA)と呼ばれる融合形式を使用することで、複雑な哺乳動物ゲノム内の場所をCas9エンドヌクレアーゼが二本鎖切断の標的とすることができます1)。これらの切断は、非相同末端結合(non-homologous end-joining: NHEJ)と総称されるプロセスを通じて、内在性のDNA修復機構によって修復されます。NHEJはエラーを起こしやすいため、ゲノムの欠失や挿入(indel)が生じ、フレームシフトや未成熟終止コドンを生成し、標的遺伝子を永久に沈黙させます。NHEJの結果生じる挿入と欠失はランダムであり、細胞によって異なることを認識しておくことが重要です。結果として生じる正確なゲノムの変化は、クローン細胞株での追加実験によって決定することができます。crRNA、tracrRNA、およびsgRNAは、細胞内で転写されるか、in vitroで転写されるか、またはカスタムで化学合成され、トランスフェクションによって導入されます。Cas9エンドヌクレアーゼの細胞内発現は、構成性または誘導性プロモーターによって駆動されるプラスミドまたは統合型レンチウイルス発現ベクターによって実現することができます。有効なレベルの細胞内Cas9は、Cas9をコードするmRNAのトランスフェクションまたはCas9タンパク質のデリバリーを通じて、追加のゲノム改変の可能性が極めて低いという利点を有するDNA-freeのシステムによってデリバリーすることもできます。
DNA-freeのCRISPR-Cas9ゲノム編集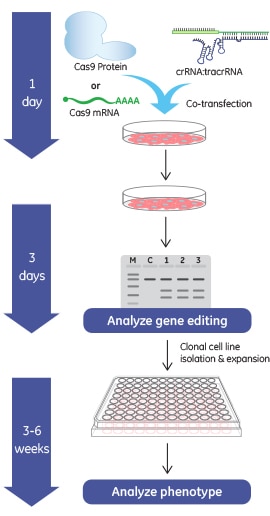
DNA-free CRISPR-Cas9ゲノム編集の本当の意味は?これは、システムがDNAベクターの形でCRISPR-Cas9コンポーネントを使用しないことを意味します。すなわち、各コンポーネントはRNAまたはタンパク質のいずれかです。ゲノム工学実験におけるCas9ヌクレアーゼ発現のソースとして、Cas9 mRNAまたは精製Cas9タンパク質から始めることは、アプリケーションによっては利点があります。それは、DNAベースのCas9またはガイドRNA発現システムの使用は、切断部位でのプラスミドDNAの統合またはランダムなレンチウイルスベクターの統合による望ましくない遺伝的変化の可能性を伴うからです。このため、DNAを含まないゲノム編集システムは、遺伝子改変細胞株の作製に適した選択肢となり得ます。実験が非クローン性細胞集団における表現型の観察を伴う場合、DNA-freeのオプションは必要ないかもしれません。しかし、実験の最終目標がCas9発現細胞の濃縮を必要とせず、潜在的な統合事象を回避したい場合は、Cas9 mRNAまたは精製Cas9タンパク質の使用をご検討ください。
相同組換え修復(Homology-directed repair: HDR)
CRISPR-Cas9による二本鎖切断は、標的遺伝子のノックアウトではなく、ノックインを作り出す機会としても利用できます。ドナーテンプレートを正確に挿入することで、遺伝子のコード領域を変化させ、変異を「固定」したり、タンパク質タグを導入したり、新しい制限部位を作ったりすることができます。我々は、Cas9ヌクレアーゼとともに化学合成crRNAとtracrRNAを用いて、一本鎖DNAで正確な挿入を行うことができることを実証しました。あるいは、Cas9の活性を変化させて、二本鎖切断ではなくニッキングを行うこともできます。Cas9ニッカーゼは、一対のcrRNA:tracrRNA複合体またはsgRNAと一緒に使用することができ、対向するDNA鎖上の間隔の狭い2つの領域を標的とし、短い二本鎖DNAと一緒に使用すると、相同組換え修復を行うことができます2,4)。
- RHDRの詳細については、Application Note Dharmacon™ Edit-R™ CRISPR-Cas9試薬と一本鎖DNAオリゴを用いた相同組換え修復ご参照ください。
- HDRのためのssDNAと合成ガイドRNAの使用に関するサイエンスポスターをダウンロードしてご覧ください。
一過性の遺伝子サイレンシングまたは遺伝子転写抑制(CRISPRi)
このアプリケーションでは、Cas9をDNAを切断できないように改変し、プロモーター領域を標的とするガイドRNAと組み合わせることで、その複合体が転写活性とそれに伴う遺伝子発現を低下させることができます5,6)。CRISPRiについての詳細はこちらからご覧ください。
一過性の内在性遺伝子の活性化(CRISPRaまたはCRISPRon)
DNAを切断できず、遺伝子転写活性化ドメインが融合されたCas9変異体を用いることで、Cas9融合タンパク質を内在性標的遺伝子のプロモーター領域にターゲティングすることにより、内在性遺伝子の発現を上昇させることができます。CRISPRaについての詳細はこちらからご覧ください。
胚性幹細胞とトランスジェニック哺乳動物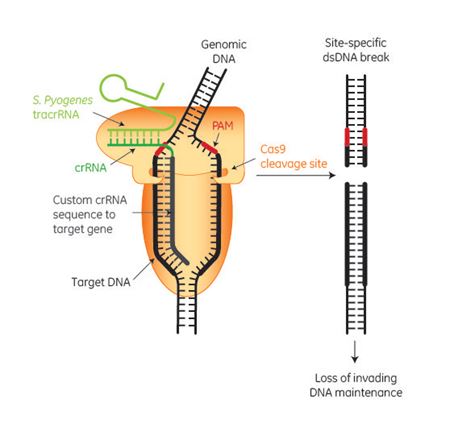
CRISPR-Casシステムは、遺伝子改変マウスを作製するために、マウス胚性幹細胞に1つまたは複数の遺伝子変化を迅速かつ効率的に導入するために使用することができます8)。同様のアプローチは、霊長類の単一細胞胚の遺伝子改変にも用いられています9)。
プール化ゲノムスケールノックアウトスクリーニング
プール化レンチウイルスライブラリーは、がん細胞の生存能力、多能性、薬剤耐性のゲノムスケールスクリーニングを行うために、低いMOIで使用されてきました10,11)。シングルコピー数で組み込むことにより、高い頻度でバイアレリック遺伝子ノックアウトを行うことができます。スクリーニングの表現型の判定基準(細胞死、増殖、薬剤耐性など)の発現に関与する遺伝子の相対的表現を、次世代シーケンサーによって同定することができます。
- CRISPR-Cas9 lentiviral sgRNAプール化スクリーニング用ライブラリーを用いた機能ゲノミクススクリーニングの概要をご覧ください。
- レンチウイルスCas9およびsgRNAを用いたプールスクリーニングのプロトコールをダウンロードしてご覧ください。
実験デザインにおけるCRISPR-Cas9コントロールの重要性
ポジティブコントロール
ほぼすべてのCRISPR-Cas9ゲノム編集アプリケーションにおいて、1つ以上のポジティブコントロールの使用は不可欠な最初のステップです。トランスフェクションの効率は細胞の種類によって異なり、高効率のゲノム編集を達成するためにはそれぞれに最適化する必要があります。高効率で編集できることが知られているガイドRNAや、明確な表現型が読み取れる遺伝子の感受性の高い領域を標的とするガイドRNAをポジティブコントロールとして用いるべきです。ポジティブコントロール実験の結果は、ミスマッチ検出アッセイによって評価し、インデルの存在を確認し、評価したトランスフェクション条件ごとに編集効率を定量します。最適な条件が確立されれば、ポジティブコントロールで決定したのと同じ高効率条件を用いて実験的ゲノム編集を開始することができます。
ネガティブコントロール
ネガティブコントロールは、表現型リードアウトで評価するようにデザインされたCRISPR-Cas9実験では特に重要です。ネガティブコントロールで処理した細胞における細胞の表現型、生存率、または遺伝子発現レベルの変化は、より正確な結果の解釈のために標的特異的crRNAで処理した細胞におけるレベルと比較することができるベースラインの細胞応答を反映している可能性が高いと言えます。
CRISPR-Cas9実験のための化学合成sgRNA
天然の化学合成の2つのRNA(crRNA:tracrRNA)を組み合わせたシステムは、ほとんどのアプリケーションにおいて非常に効率的でコスト効率が高いですが、in vivoやex vivoのモデルを扱う研究者は、sgRNAシステムを好む傾向があります。
- 化学合成 99-merシングルガイドRNAを用いたCRISPR-Cas9ゲノム編集の詳細はこちら
CRISPR-Cas9ガイドRNAアルゴリズム
最適な機能的ガイドRNAを選択する基準を特定するために、GFPリードアウトを用いたプロテアソーム機能の表現型アッセイを用いた機能的遺伝子ノックアウトに基づくアルゴリズムを開発しました。機能的タンパク質ノックアウトを引き起こす可能性の高いガイドRNAを選択するために、Edit-R アルゴリズムがどのように開発されたのか、その概要をご覧ください。
CRISPR-Cas9ガイドRNAの特異性
CRISPRの特異性には、潜在的なオフターゲット切断部位の同定が不可欠です。利用可能なアライメントツールとストラテジーの詳細をご覧ください。
誘導性レンチウイルスCas9ヌクレアーゼ
ゲノム編集実験のタイミングは、特にプール化スクリーニングの設定、代謝負荷増加のリスクを伴わない安定した細胞株の作製、または前駆体の状態でCas9を統合し、後に誘導された細胞の状態で実験を実施する場合など、非常に重要です。誘導可能なレンチウイルスCas9ヌクレアーゼは、編集が必要な時にのみ起こることを確実にするために必要な時間的制御を提供することができます。
References:
- Jinek, M., et al., RNA-programmed genome editing in human cells. Elife, 2013. 2: p. e00471.
- Cong, L., et al., Multiplex genome engineering using CRISPR/Cas systems. Science, 2013. 339(6121): p. 819-23.
- Mali, P., et al., RNA-guided human genome engineering via Cas9. Science, 2013. 339(6121): p. 823-6.
- Ran, F.A., et al., Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013. 154(6): p. 1380-9.
- Qi, L.S., et al., Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013. 152(5): p. 1173-83.
- Gilbert, L.A., et al., CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013. 154(2): p. 442-51.
- Cheng, A.W., et al., Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res, 2013. 23(10): p. 1163-71.
- Wang, H., et al., One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013. 153(4): p. 910-8.
- Niu, Y., et al., Generation of Gene-Modified Cynomolgus Monkey via Cas9/RNA-Mediated Gene Targeting in One-Cell Embryos. Cell, 2014. 156(4): p. 836-43.
- Wang, T., et al., Genetic screens in human cells using the CRISPR-Cas9 system. Science, 2014. 343(6166): p. 80-4.
- Shalem, O., et al., Genome-scale CRISPR-Cas9 knockout scre
Order products
Cas9ヌクレアーゼ
強固なCas9発現を確実にするために、細胞タイプに最適なプロモーターを設定する、またはDNA-freeのオプションを探索する
CRISPRコントロールと検出プライマー
CRISPR-Cas9ゲノム編集実験の評価には適切なコントロールが不可欠です。
CRISPRガイドRNA
Cas9切断を誘導する高品質ですぐに使用可能なレンチウイルスおよび化学合成試薬
CRISPR-Cas9ゲノム編集
信頼性の高いゲノム工学のための最適化されたツール
CRISPR-Cas9スクリーニングライブラリー
ハイスループットゲノム編集研究のためのプール化sgRNAまたはアレイ化crRNA
Helpful resources
CRISPR-Cas9遺伝子工学ワークフロー - App Note
CRISPR-Cas9システムは、多くの異なる生物学的用途におけるゲノム工学に広く使用されている
Edit-R CRISPR-Cas9 Cas9ヌクレアーゼ発現プラスミドと化学合成RNAによる遺伝子工学 - Tech Manual
Dharmacon™ Edit-R™ Cas9ヌクレアーゼ発現プラスミドと化学合成ガイドRNAを用いたCRISPR-Cas9遺伝子工学
ビデオ:Edit-R CRISPR-Cas9 遺伝子工学プラットフォーム
CRISPR遺伝子ノックアウトのワークフロー、細胞タイプ、実験目的に適した製品の選択方法をご紹介します。このビデオでは、実験に最適なコンポーネントを選択するためのいくつかのハイレベルな質問について説明します。
CRISPR-Cas9システムは、様々な生物学的アプリケーションにおけるゲノム工学に広く使用されています。
