Add any of our CRISPR portfolio reagents (CRISPRko, CRISPRa, CRISPRi) to your cart to see new pricing (up to 56% off) now including All-in-one lentiviral sgRNA formats in 2026
CRISPR interference(CRISPRi)は、特異性の高い遺伝子ノックダウンの新しい手法です。
CRISPRiシステムは、独自のSALL1-SDS3リプレッサーコンストラクトに融合した不活化Cas9ヌクレアーゼ(dCas9)を使用して、DNAを切断せずに標的遺伝子の転写をブロックします。
CRISPRiのメリット: |
理想的なアプリケーション: |
|
|
|
|
|
|
|
CRISPRiシステム
Dharmacon™のCRISPRmod CRISPRiシステムには、リプレッサータンパク質(SALL1およびSDS3)に融合した特殊なdCas9と、遺伝子の転写開始部位(transcriptional start site: TSS)のすぐ下流の領域をターゲットとするように設計されたガイドRNAの2つのコンポーネントが必要です。ガイドRNAはdCas9-SALL1-SDS3と複合体を形成し、リプレッサー複合体をDNAの標的部位に誘導します(figure 1)。
私共は、dCas9-リプレッサータンパク質とガイドRNAの細胞への導入の双方に、レンチウイルスによる長期発現のアプローチおよび一過性発現のアプローチを提供します。
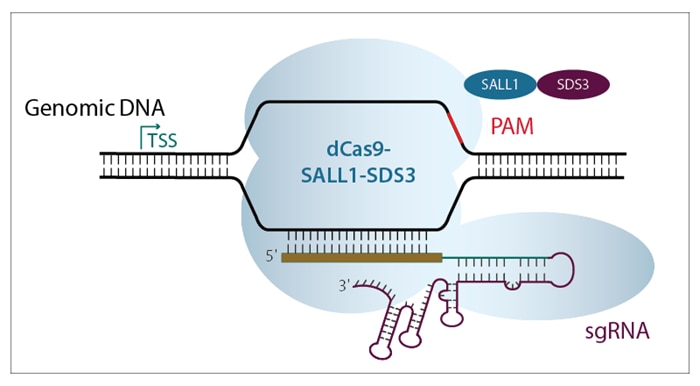
Figure 1. 遺伝子の転写開始部位(TSS)のすぐ下流の領域を標的とするsgRNAと複合体を形成したdCas9-SALL1-SDS3の図解。CRISPRiシステムでは、dCas9-SALL1-SDS3タンパク質とCRISPRiガイドRNAの2つのコンポーネントを目的の細胞に導入する必要があります。
CRISPRi dCas9-SALL1-SDS3リプレッサーコンストラクト
CRISPRiシステムでは、ネイティブのCas9のDNA切断機能を点突然変異によって消滅させ、その不活化されたCas9(dCas9)タンパク質にHorizon独自のSALL1およびSDS3リプレッサードメインを融合することにより、遺伝子抑制に用いることができるようになりました。
私共は、dCas9-SALL1-SDS3コンストラクトを作製する前に、多くのリプレッサーコンストラクトをテストしました。現在の多くのCRISPRiシステムはdCas9-KRABバリアント(Gilbert et al., 2013,Yeo et al., 2018, Moghadam et al., 2020, Alerasool et al., 2020)に依存していますが、dCas9タンパク質に共有結合したHorizonの新しいリプレッサーは、クロマチンリモデリングと遺伝子サイレンシングに関与するタンパク質を動員することにより、標的遺伝子の転写をさらに阻害します(Alland et al.2002)。その結果、広く機能するリプレッサーが得られ、遺伝子発現は低下しますが、完全消滅にはなりません(figure 2)。
私共はまた、全トランスクリプトームRNAシーケンシング(RNA-seq)を使用してdCas9-SALL1-SDS3とdCas9-KRABの特異性を比較し、標的遺伝子全体で一貫した傾向を観察しました。標的遺伝子の抑制(figure 3)。Horizonのリプレッサーシステムは、追加のノイズや不確実性を導入することなく、より明確な実験結果を研究者に提供するものです。
CRISPRiガイドRNA
すべてのCRISPRmodガイドRNAは、特異性とパフォーマンスを最大化するようにアルゴリズムが最適化されており、化学合成およびレンチウイルスパッケージフォーマットの両方で利用できます。
CRISPRiは、転写抑制を引き起こすために、標的遺伝子の転写開始部位(transcriptional start site: TSS)の下流の0〜300塩基対の領域を標的とするガイドRNAのデザインを必要とします。TSSは、いつも十分にアノテーションされているわけではなく、また、他のタンパク質因子のためにアクセスできないことがあるため、機能的なガイドRNAの設計が困難な場合があります。HorizonのCRISPRiガイドRNA製品は、機械学習技術を用いて開発されたCRISPRi v2.1アルゴリズム(MA Horlbeck et al., 2016)を使用しています。このアルゴリズムは、FANTOMおよびEnsemblデータベースを使用してTSSを予測し、クロマチン、位置、および配列データを組み込んで、非常に効果的なガイドRNAの設計を予測しました。別個のTSSを持つ遺伝子については、それぞれのTSSを標的とするガイドRNAが提供されています。Horizonは、TSSごとに3つのガイドRNAデザインを、予測される機能の順にランク付けしてリストしています。
CRISPRi化学合成sgRNA
私共は、CRISPRi化学合成シングルガイドRNA(sgRNA)を最初に提供したことを誇らしく思います。このフォーマットは、CRISPRi dCas9-SALL1-SDS3 mRNAとのコトランスフェクションまたはエレクトロポレーション、または安定したdCas9-SALL1-SDS3発現細胞への導入を可能にし、いずれかの方法によりCRISPRi結果を取得するための最速の方法を選べます。この方法では、遺伝子発現抑制はトランスフェクションの24時間後には認められ、96時間まで持続します(figure 4)。
CRISPRi遺伝子抑制はトランスフェクションの24時間後に観察され、トランスフェクションの48〜72時間後に最大になる
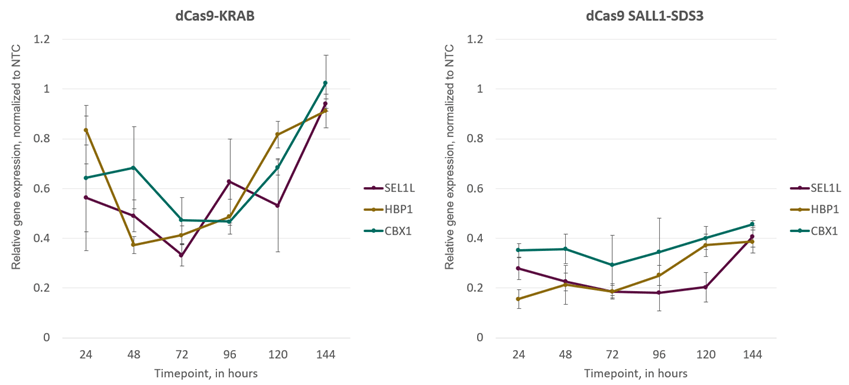
Figure 4. CRISPRi化学合成sgRNAは、dCas9-KRABおよびdCas9-SALL1-SDS3発現細胞の両方でトランスフェクション後48〜72時間で最大の抑制を達成します。dCas9-KRABまたはdCas9-SALL1-SDS3を安定して発現するU2OS細胞を、10,000細胞/ウェルでプレーティングし、DharmaFECT 4トランスフェクション試薬を使用して、CBX1、HBP1、またはSEL1Lをターゲットとするデザイン済み化学合成sgRNA混合物(25nM)でトランスフェクトしました。トランスフェクションの24、48、72、96、120、および144時間後に細胞を回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各標的遺伝子の相対的発現は、ハウスキーピング遺伝子としてGAPDHを用いたΔΔCq法で計算し、non-targeting コントロール(NTC)に対して正規化しました。
化学合成sgRNAの混合物(プール)によるCRISPRiの効率の向上
化学合成sgRNAを混合したプールフォーマットのsgRNAは、最も機能的な個々のガイドRNAと同等またはそれ以上の遺伝子ノックダウンを実現します(figure 5)。
プール化は、最大の遺伝子転写抑制を実現するための優れた戦略であり、実験の規模を縮小するのにも役立ちます(例えば、アレイ化プレート形式で複数の遺伝子の分析を実行する場合)。
sgRNAをプール化すると遺伝子ノックダウンが促進される
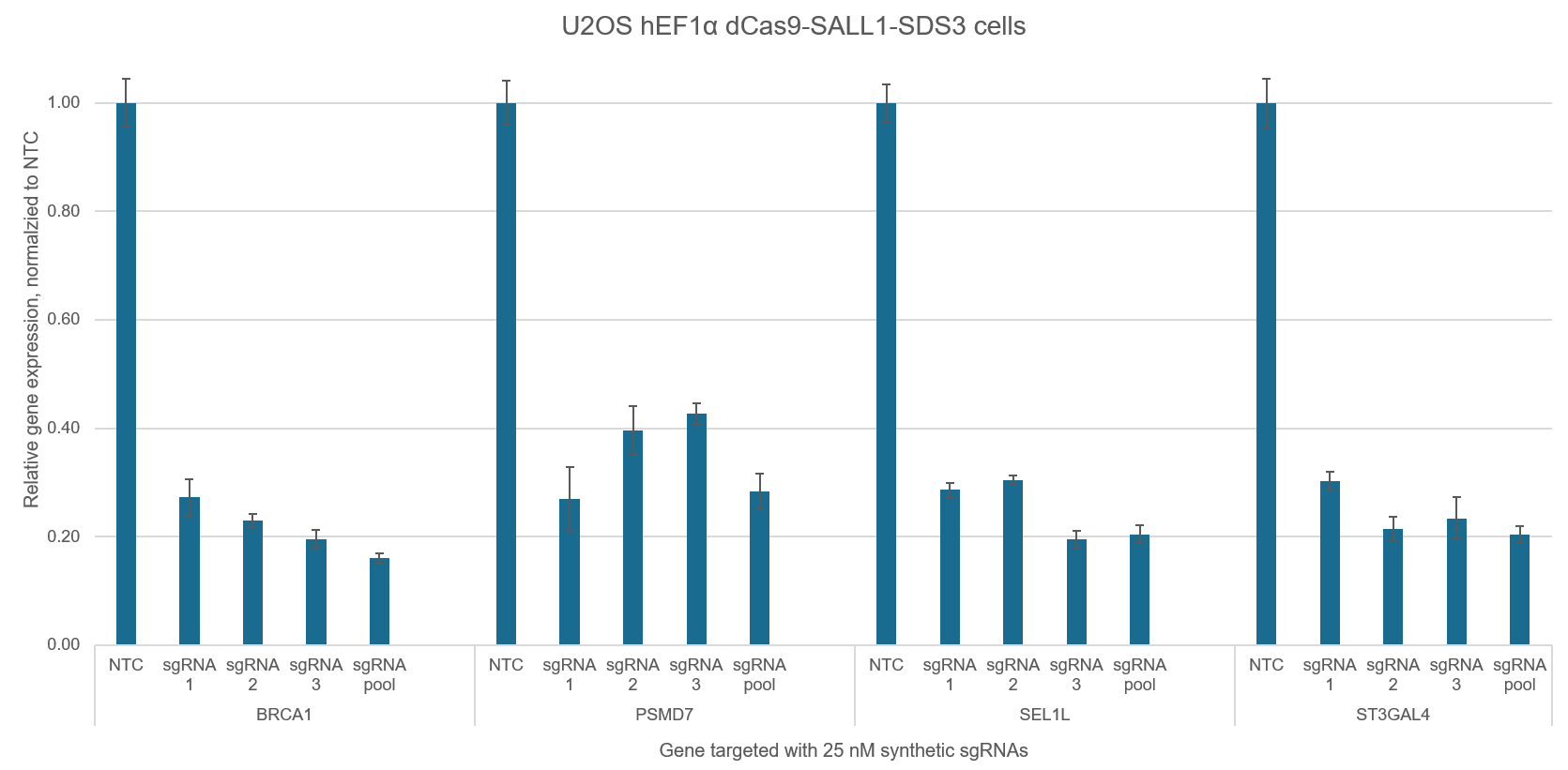
Figure 5. 個々のCRISPRi sgRNAは、独立して強力な標的遺伝子抑制を達成しますが、一緒に混合(プール)して単一の試薬にすると、抑制レベルが向上します。dCas9-SALL1-SDS3を安定して発現するU2OS細胞を10,000細胞/ウェルでプレーティングし、DharmaFECT 4トランスフェクション試薬を使用して、BRCA1、PSMD7、SEL1L、またはST3GAL4を標的とするCRISPRi化学合成sgRNAをトランスフェクトしました。CRISPRi sgRNAは、個別に使用、またはプールして使用しました(合計濃度は25 nM)。トランスフェクションの72時間後に細胞を回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各遺伝子の相対的発現は、ハウスキーピング遺伝子としてGAPDHを用いたΔΔCq法で計算し、non-targeting コントロール(NTC)に対して正規化しました。
複数の遺伝子の同時ノックダウン
CRISPRi化学合成sgRNAは、複数の遺伝子を同時にノックダウンするためのユニークな方法です(figure 6)。各ガイドRNAは、その特定のDNAターゲットに独立して結合し、外から供給されるdCas9融合タンパク質に依存するため、内在性経路の競合を最小限に抑えます。Horizonの科学者は、iPSC細胞におけるマルチプレックスCRISPRiの有効性を検証しました。
化学合成sgRNAは、複数の遺伝子の同時抑制のための簡単な多重化を可能にする
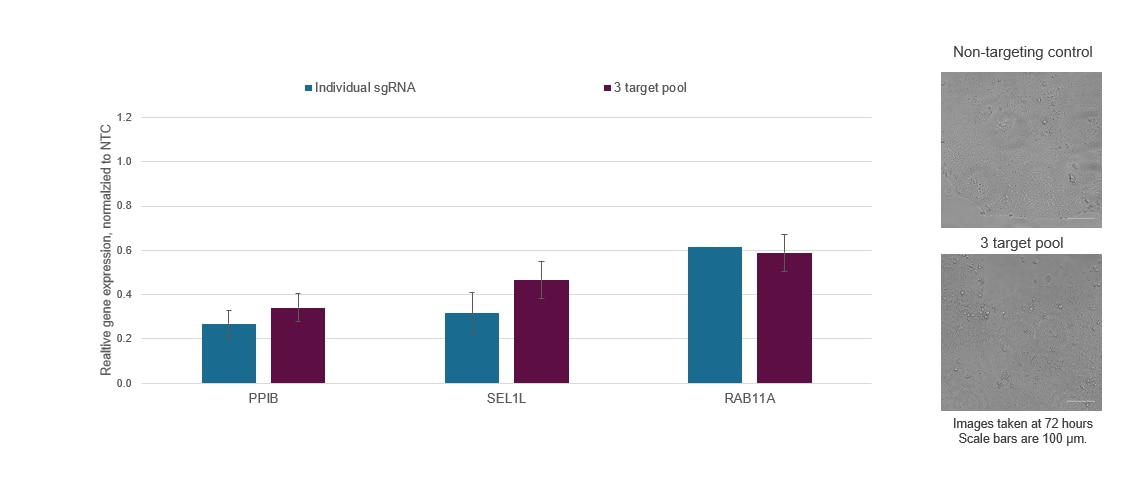
Figure 6. 個々のCRISPRi sgRNAを混合して単一の試薬にしたプールを用いて、標的遺伝子の転写抑制を強化したり、多重化して複数の遺伝子の同時抑制を可能にしたりすることができます。dCas9-SALL1-SDS3を安定して発現するWTC-11ヒトiPS細胞に、Lonza 96ウェルシャトルシステムを用いて、PPIB、SEL1L、およびRAB11Aを標的とするCRISPRi化学合成sgRNAを導入しました。各遺伝子ターゲットに対して最も有効なCRISPRi sgRNAを選択し、個別に、またはガイドあたり3 µMの濃度でマルチターゲットプール(3つの異なる遺伝子をターゲットとするsgRNAの混合物)の一部として使用しました。ヌクレオフェクションの72時間後に細胞を回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各標的遺伝子の相対的発現は、ACTBをハウスキーピング遺伝子として用いたΔΔCq法で計算し、non-targeting コントロール(NTC)に対して正規化しました。
遺伝子発現抑制の確認
RT-qPCR、ウエスタンブロット、または免疫蛍光分析など、遺伝子発現抑制を確認する方法はたくさんあります。通常、non-targeting コントロールとCRISPRiガイドRNAで処理されたサンプル間の標的遺伝子発現レベルの相対的変化を測定する最も速くて簡単な方法はRT-qPCRです。RT-qPCR分析は、SYBRグリーン法またはプローブベースの遺伝子発現アッセイのいずれかで行います。RT-qPCRを実行する際に注意すべきことの1つは、遺伝子発現レベルがCRISPRi処理後に検出できないレベルに低下する可能性があることです。この場合、ΔΔCq分析法を使用する場合、計算を実行するにはゼロ以外の値が必要であるため、qPCR機器の検出限界を表す任意の値が「検出不能」のプレースホルダーとして使用されます。ほとんどの場合、この値は35〜40になりますが、これはプログラムされたサイクル数と機器のCq決定方法によって異なります。多くのプロトコルでは、標準のqPCRサイクリング条件に追加のサイクル(合計で最大45)を追加することを推奨しています。
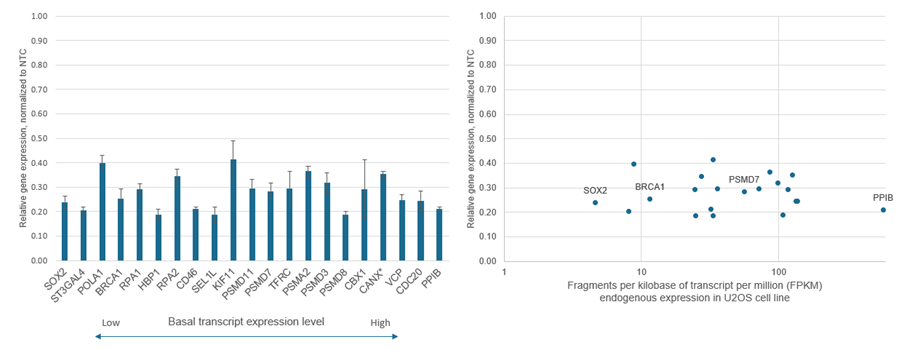
私たちの研究室では、遺伝子ノックダウンのレベルは、それぞれの遺伝子の基本的な発現レベルとの相関関係がないことが示されています(図7)。たとえば、遺伝子PPIBは、peptidyl-prolyl cis-trans isomerase(PPIase)ファミリーのメンバーです。PPIBは多くの細胞型で高く発現しており、ポジティブコントロールとして推奨される遺伝子の1つです。ここでは、広範囲の遺伝子の相対的な遺伝子ノックダウンを示します。興味深いことに、基礎発現レベルが高いまたは低い遺伝子間で同程度の転写抑制が見られます。
CRISPRiは内在性の発現レベルに依存しない
Figure 7. CRISPRiの転写抑制は遺伝子によって異なりますが、内在性遺伝子の発現レベルに依存しているようには見えません。データは、dCas9-SALL1-SDS3を安定して発現するU2OS細胞において、以下のマッチング条件で実行された複数のトランスフェクションから編集されました。細胞を10,000細胞/ウェルでプレーティングし、DharmaFECT 4トランスフェクション試薬を使用して、異なる基礎発現レベルの21個の遺伝子を標的とするCRISPRi化学合成sgRNAプール(25 nM)をトランスフェクトしました。トランスフェクションの72時間後に細胞を回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各標的遺伝子の相対的発現は、ハウスキーピング遺伝子としてGAPDHを用いたΔΔCq法で計算し、non-targeting コントロール(NTC)に対して正規化しました。CRISPRiを介した標的遺伝子の抑制は、左グラフに示されています。ここでは、遺伝子が低レベルから高レベルの基本転写産物発現の順に並べられています。。右グラフでは、標的遺伝子の発現が、100万あたりの転写産物のキロベースあたりのフラグメント(FPKM)に対してプロットされています。これは、親U2OS細胞株からのRNA-Seqデータに基づく基本的な遺伝子発現の表現です。U2OS細胞では、PPIBはSOX2の約100倍の基礎レベルで発現しますが、両方の遺伝子はCRISPRmod CRISPRi試薬を使用して基礎的な発現の約20〜25%に抑制できます。
直交検証
検証されたサイエンスは良い研究であると言えます。Dharmacon試薬は、細胞経路を調べる幅広いツールを含み、DNAレベルでの遺伝子の不活性化、RNA転写の調節、または成熟したmRNA転写物の分解を可能にします。私共は、アプリケーションに最適となる遺伝子の活性化または不活性化の方法を選択するお手伝いをします。堅牢でエレガントな検証済みのサイエンスには2つの方法を選択してください。
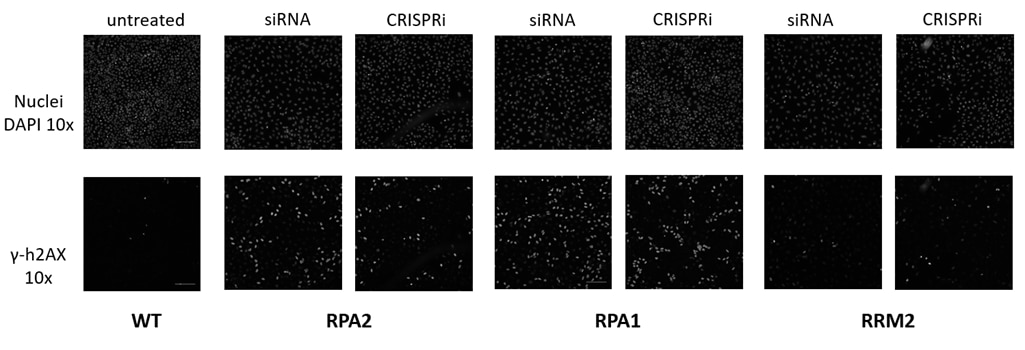
CRISPRiは、RNAiまたはCRISPRノックアウトスクリーニング後の後続研究に理想的な機会を提供します(figure 8)。ここでは、CRISPRiを使用して、以前に実施したアレイ化siRNAスクリーニングをフォローアップしました。
化学合成CRISPRi試薬は、siRNAスクリーンからのヒットを直交的に検証可能
Figure 8. 損傷応答アッセイ。DNA二本鎖切断に伴ってh4AX(コアヒストン複合体の構成要素)は急速にリン酸化され、DNA損傷シグナル伝達ネットワークを介して一連の応答を引き起こします。DNA損傷経路に重要なタンパク質をノックダウンすると、修復されていない二本鎖DNA切断が蓄積し、リン酸化h4AX(γ-h4AX)が増加します。以前のRNAiスクリーニングからのヒットを、CRISPRi試薬を使用して直交的に検証しました。hEF1αプロモーター下でdCas9-SALL1-SDS3を構成的に発現するU2OS細胞に、DharmaFECT 4トランスフェクション試薬を使用してRPA2、RPA1、またはRRM2を標的とするプールフォーマットのsgRNA(50nM)またはON-TARGETplus SMART pools(50nM)をトランスフェクトしました。トランスフェクションの72時間後、細胞を固定し、抗リン酸化h4AX抗体で染色し、ヘキスト染色を使用して核を同定しました。複製プレートを回収し、全RNAを単離し、RT-qPCRを使用して相対的な遺伝子発現を測定しました。各標的遺伝子の相対的発現は、ハウスキーピング遺伝子としてGAPDHを用いたΔΔCq法で計算し、non-targeting コントロール(NTC)に対して正規化しました。
CRISPRiの実験的考察
CRISPRiの実験条件には多くのオプションと考慮事項があります。一般に、ユーザーは、dCas9-SALL1-SDS3を安定発現する細胞で作業するときに、最も堅牢なノックダウンを実現できます。長時間のある時点のアッセイ(120時間以上)では、多くの人がレンチウイルスsgRNAを選択します。短期間のアッセイ(120時間未満)の場合、化学合成sgRNAは、ガイドRNA発現ベクターよりも強力な遺伝子発現抑制を提供します。
| CRISPRi dCas9-SALL1-SDS3ソース | ガイドRNAフォーマット | デリバリー方法 | メリットと推奨される使い方 |
|---|---|---|---|
|
CRISPRi dCas9-SALL1-SDS3 lentiviral particles CRISPRi dCas9安定発現細胞のための形質導入と選択 |
CRISPRi synthetic sgRNA(化学合成sgRNA) | トランスフェクションまたはエレクトロポレーション |
|
| CRISPRi lentiviral sgRNA particles | 形質導入 |
|
|
| CRISPRi dCas9-SALL1-SDS3 mRNA | CRISPRi synthetic sgRNA(化学合成sgRNA) | コトランスフェクションまたはエレクトロポレーション |
|
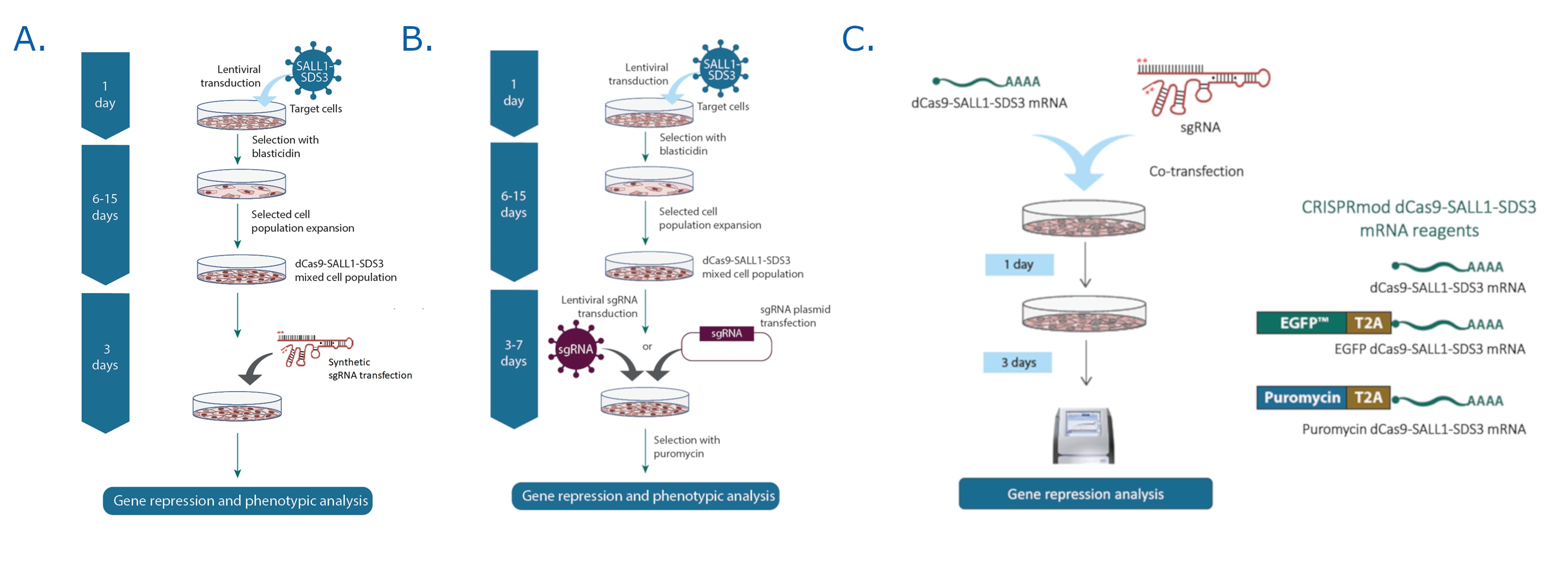
CRISPRiワークフロー
転写抑制にCRISPRiを使用する複数のワークフローオプションを提供しています。
- dCas9-SALL1-SDS3 lentiviral particlesで形質導入して、‘CRISPRi ready’のdCas9-SALL1-SDS3安定発現細胞株を作製します。次に、A)化学合成sgRNAまたはB)lentiviral sgRNA のいずれかを導入して、標的遺伝子をノックダウンします。このシステムは、スクリーニング、長時間のアッセイ、またはトランスフェクションが困難な細胞タイプでの作業に最適です。
- dCas9-SALL1-SDS3 mRNAおよび化学合成sgRNAを共導入し、蛍光またはピューロマイシン耐性オプションを使用して細胞集団を濃縮します。このシステムは、迅速で一過性の遺伝子転写抑制研究に最適です。
References
- L.A. Gilbert, et al., CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154(2): p. 442-51 (2013).
- NC. Yeo, et al., An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Methods. 15(8): p. 611-616 (2018).
- F. Moghadam, et al., Synthetic immunomodulation with a CRISPR super-repressor in vivo. Nat Cell Biol. 22(9): p. 1143-1154 (2020).
- N. Alerasool, et al., An efficient KRAB domain for CRISPRi applications in human cells. Nat Methods. 17(11): p. 1093-1096 (2020).
- L. Alland, et al., Identification of mammalian Sds3, an integral component of the Sin3? Histone deacetylase corepressor complex. Mol and cell bio. 22(8): p.2743-2750 (2002).
- M. A. Horlbeck et al., Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760 (2016).
Order products
CRISPRi dCas9-SALL1-SDS3
あらゆるCRISPRiワークフローソリューションのための精製dCas9-SALL1-SDS3 mRNA、またはレンチウイルス粒子をご用意しています。
CRISPRi sgRNA
あらゆるヒト遺伝子の特異性の高い転写抑制のための化学合成、およびレンチウイルス発現CRISPRi sgRNA
CRISPRmod Controls
Positive and non-targeting controls to ensure your CRISPRi or CRISPRa experimental conditions
Helpful resources
CRISPRi研究ポスター
新しいdCas9融合タンパク質と化学合成sgRNAによるCRISPRを介する遺伝子転写抑制
記事:CRISPRa、CRISPRiとは?
遺伝子の転写活性化と転写抑制のためのCRISPR-Cas9ベース技術