CRISPRガイドRNAはCas9ヌクレアーゼをプログラムし、特定の場所でゲノムDNAを切断します。二本鎖切断(double-strand break: DSB)が生じると、哺乳動物細胞は内在性のメカニズムを利用してDSBを修復します。ssDNAオリゴまたはプラスミドドナーのいずれかのドナーDNAの存在下で、DSBはHDR(相同組換え修復)を使用して正確に修復でき、目的のゲノムの変化(挿入、欠失、または置換)を引き起こすことができます。
Custom CRISPR guide design
-
CRISPR Design Tool
カスタムガイドRNAの注文、または独自の化学合成sgRNA、crRNA、またはlentiviral sgRNAの設計および注文を、使いやすいインターフェースを使用して行うことができます。
HDRドナーテンプレートのデザインと注文用ツール
-
Edit-R HDR Donor Designer - oligo
挿入、欠失、またはその他の変更のための一本鎖DNAドナーオリゴ(150nt以下)を設計し、注文することができます。
-
Edit-R HDR Donor Designer - plasmid
mKate2またはEGFP蛍光マーカー、またはカスタムインサート挿入用のプラスミドDNAドナーキットを設計し、注文することができます。
HDR Donor Template Kits
-
Edit-R HDRプラスミドドナーキット
HDR用のプラスミドドナーを迅速かつ簡単に組み立てることができます。
-
Edit-R HDRプラスミドドナーコンポーネント
Edit-Rプラスミドドナーキットのベクターバックボーンおよびインサートコンポーネントのみをご提供しています。
-
Edit-R HDRプラスミドドナープライマー
Edit-RプラスミドドナーキットのPCRコンポーネントのみをご提供しています。
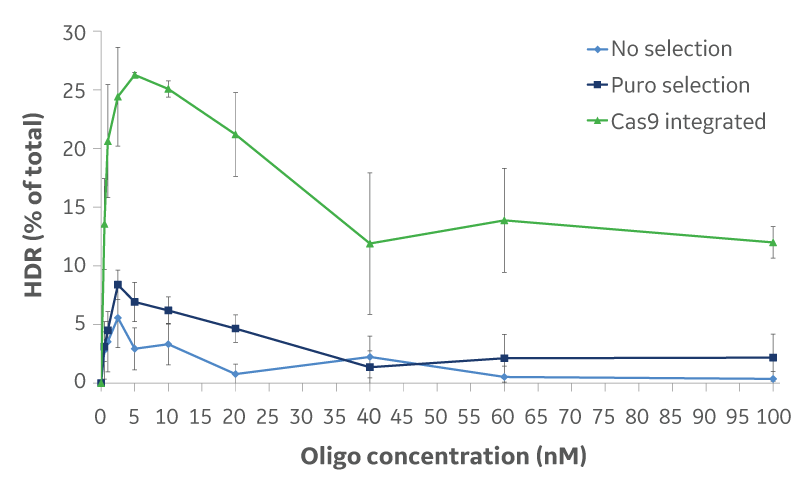
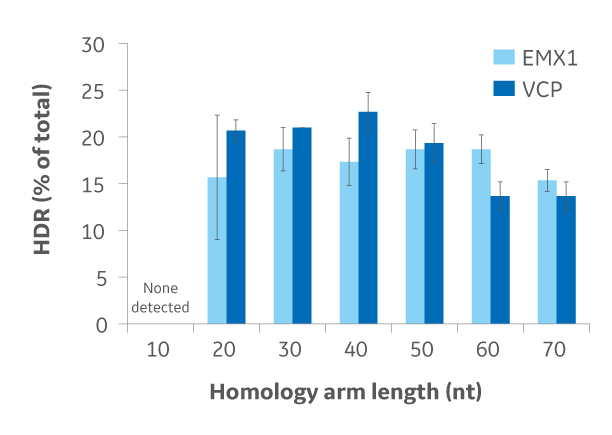
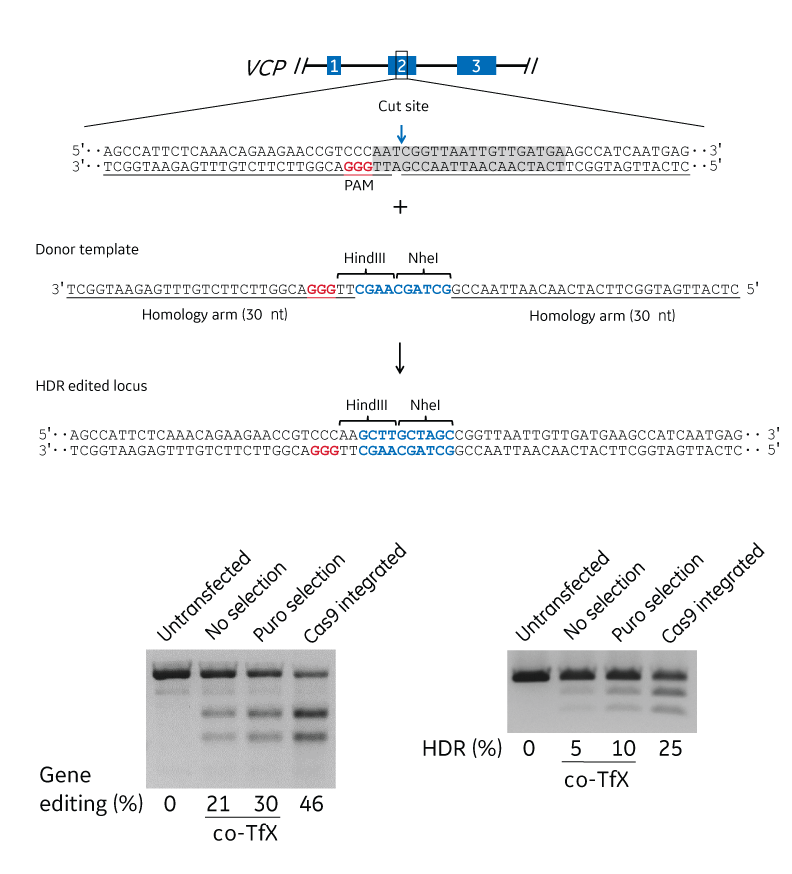
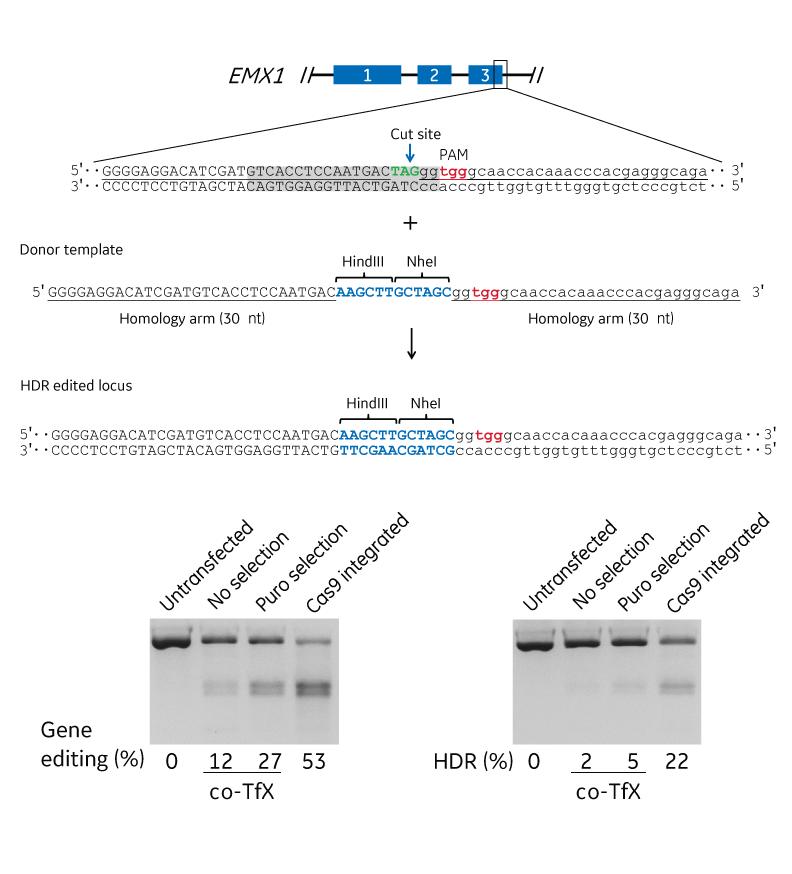
相同組換え修復(Homology-directed repair: HDR)は、切断部位に隣接する領域と十分な相同性を持つドナーテンプレートの存在に依存しています。相同性アームの長さとドナーのタイプ(ssDNAオリゴまたはプラスミド)は、正確な修飾のタイプとサイズに依存します。Figure 1は、タグや制限酵素部位などの短いDNA領域を挿入するためのドナーオリゴの使用を示しています。Edit-R HDR Donor Designerを使用すると、ドナーオリゴのカスタマイズを簡単に行うことができます。
短いDNA配列を正確に挿入するためのドナーオリゴ
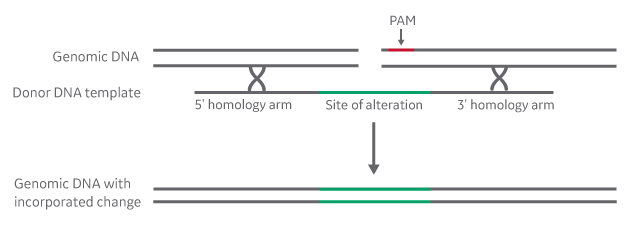
Figure 1. 蛍光マーカーなどのより大きな挿入の場合、効率的な組み換えをサポートするために適切な長さの相同性アームを備えたドナープラスミドテンプレートが必要です。Edit-R HDR DNA Plasmid donor kitとEdit-R HDR Donor Designerは、ドナープラスミドの設計と組み立てを迅速かつ簡単に行うことができます。
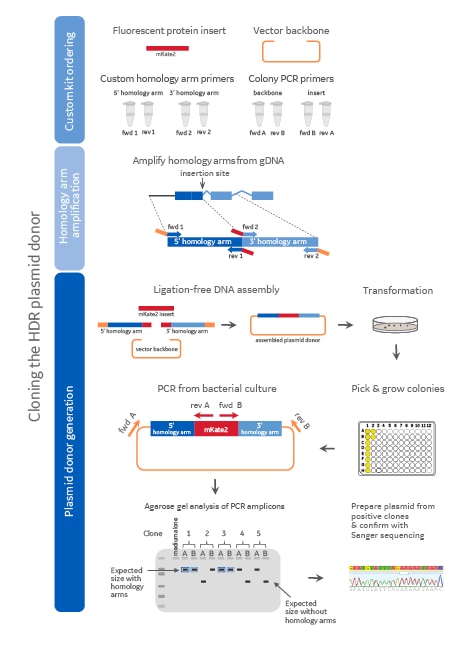
蛍光選択マーカー挿入のためのドナープラスミドアセンブリ
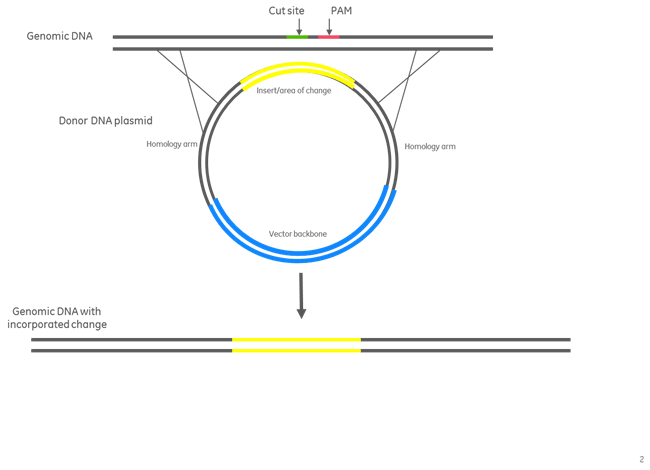
Figure 2. 蛍光選択マーカー挿入のためのドナープラスミドアセンブリ