Edit-R HDRプラスミドドナーキット
EGFP、mKate2、またはカスタム配列を持つHDRノックインプラスミドDNAドナーの組み立て用キット

CRISPR-Cas9を使用した遺伝子工学では、切断された標的DNAの修復に、非相同末端結合(non-homologous end joining: NHEJ)または相同組換え修復(homology-directed repair: HDR)の2つのいずれかの経路を用います。特定の変異を生じさせたり、目的の配列を挿入または削除したりするためのDNAの正確な修復には、HDR経路が必要です。このプロセスは、DNAの二本鎖切断修復に使用できるDNAドナーテンプレートが利用できるかどうかに依存します。最も一般的なアプリケーションの1つは、遺伝子の一端への蛍光レポーター配列の挿入です。Edit-R HDRプラスミドドナーキットを使用すると、EGPFまたはmKate2蛍光レポーターまたはユーザー独自のカスタム配列をほぼすべてのゲノム位置に挿入するためのプラスミドドナーを迅速かつ簡単に作製できます。
Highlights
- DNAドナープラスミドを迅速かつ効率的に組み立てます。
- ドナープラスミドの設計、組み立て、確認のための検証済みプロトコールをご用意しています。
- CRISPR Design Tool を利用して、切断部位へのcrRNAを設計できます。
- 挿入部位を挟む5'および3'相同性アームを作成するためのプライマー設計用のHDR Donor Designerとシームレスに統合されています。
HDRプラスミドドナーキットには、カスタムDNAドナーテンプレートプラスミドを構築および品質チェックするためのすべてのDNAコンポーネントが含まれており、Edit-R HDR DNAドナープラスミドホモロジーアーム・カスタムプライマーペアと組み合わせると、ほぼすべてのゲノム位置に蛍光レポーターを挿入できます。
HDR Donor Designerは、プラスミドの組み立てに必要な5'および3'相同性アームの増幅用PCRプライマーを設計および注文するために使用されます。
キット構成と容量
| Cat # | Description | Item# | Amount |
|---|---|---|---|
| UK-008100-01-10 | Edit-R HDR Plasmid Donor Kit - EGFP | ||
| Edit-R HDR Plasmid Donor Backbone | U-008300-01-1200 | 1200 ng | |
| Edit-R HDR insert - EGFP | U-008100-01-250 | 250 ng | |
| Edit-R Colony PCR Primer EGFP Forward | U-008100-FB-05 | 5 nmol | |
| Edit-R Colony PCR Primer EGFP Reverse | U-008100-RA-05 | 5 nmol | |
| Edit-R Colony PCR Primer Backbone Forward | U-008300-FW-05 | 5 nmol | |
| Edit-R Colony PCR Primer Backbone Reverse | U-008300-RV-05 | 5 nmol | |
| UK-008200-03-10 | Edit-R HDR Plasmid Donor Kit -mKate2 | ||
| Edit-R HDR Plasmid Donor Backbone | U-008300-01-1200 | 1200 ng | |
| Edit-R HDR insert - mKate2 | U-008200-01-250 | 250 ng | |
| Edit-R Colony PCR Primer mKate2 Forward B | U-008200-FB-05 | 5 nmol | |
| Edit-R Colony PCR Primer mKate2 Reverse A | U-008200-RA-05 | 5 nmol | |
| Edit-R Colony PCR Primer Backbone Forward | U-008300-FW-05 | 5 nmol | |
| Edit-R Colony PCR Primer Backbone Reverse | U-008300-RV-05 | 5 nmol | |
| UK-008300-01-10 | Edit-R HDR Plasmid Donor Kit - Universal | ||
| Edit-R HDR Plasmid Donor Backbone | U-008300-01-1200 | 1200 ng | |
| Edit-R Colony PCR Primer Backbone Forward | U-008300-FW-05 | 5 nmol | |
| Edit-R Colony PCR Primer Backbone Reverse | U-008300-RV-05 | 5 nmol |
Diagram of the plasmid donor cloning workflow
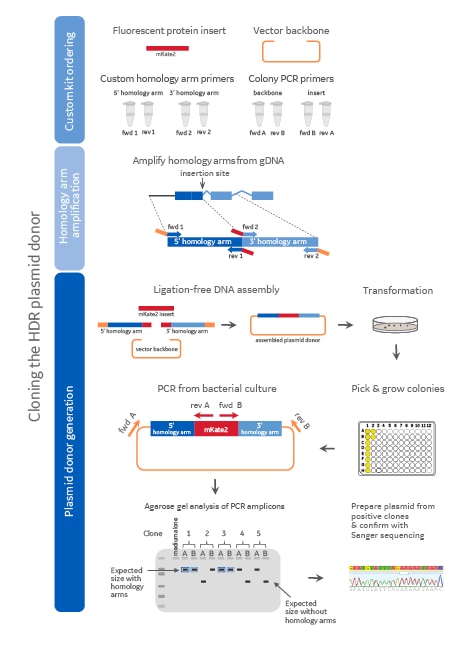
Diagram of the plasmid donor cloning workflow including component ordering, homology arm amplification, and plasmid donor generation. Colors on the diagram indicate the origin of the DNA (dark blue = 5' homology arm, light blue = 3' homology arm, red = mKate2 fluorescent reporter sequence, orange = plasmid backbone). medium alone = media-only negative PCR control.
U2OS細胞におけるEGFPでN末端にタグ付けされたLMNA
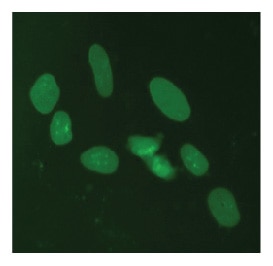
U2OS細胞に、Cas9 mRNA、LMNAをターゲットとする化学合成crRNA:tracrRNA、および相同性アームあたり約1000 bpの相同性を含むLMNAのN末端に特異的なEGFPドナープラスミドをDharmaFECT Duoを用いてトランスフェクトしました。必要に応じて細胞を継代し、トランスフェクションの7日後にInCell 2200ハイコンテント蛍光顕微鏡で画像化しました。
U2OS細胞におけるmKate2-SEC61B融合
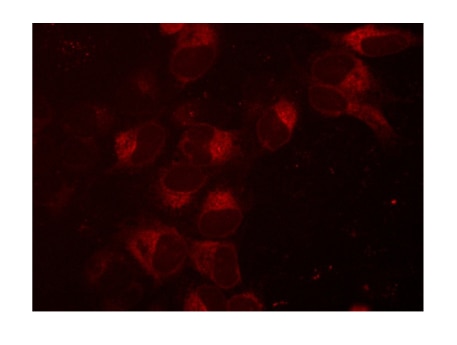
小胞体におけるタンパク質輸送の構成要素であるSEC61B遺伝子へのmKate2 N末端融合の作製。U2OS細胞に50 nM SEC61Bを標的とする化学合成crRNA:tracrRNA、25 nM Cas9タンパク質、2 ng/µL SEC61B mKate2 HDRドナープラスミド、0.3 µL/ウェルのDharmaFECT Duoをトランスフェクトし、トランスフェクションの7日後に蛍光顕微鏡で可視化しました。Note: Cas9タンパク質トランスフェクションのリボ核タンパク質(RNP)複合体比は2:1です。
マルチプレックスCRISPR-Cas9を介したノックインによって作製されたEGFP-LMNAおよびmKate2-SEC61B蛍光レポーターを備えたU2OS細胞株
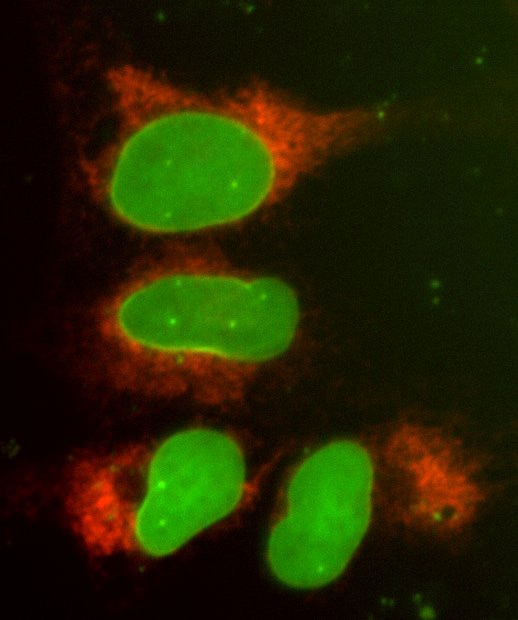
LMNAおよびSEC61Bを標的とする化学合成crRNAを、CRISPR RNA Configuratorを使用して各タンパク質のN末端付近に二本鎖切断を生じるように設計しました。Edit-R HDR Donor Designerを使用してLMNAおよびmKate2のN末端にそれぞれ特異的なEGFPおよびmKate2ドナープラスミドを生成する試薬を設計しました。両方のドナープラスミドは、相同性アームあたり約1000塩基対の相同性を含んでいます。U2OS細胞は、トランスフェクションの前日に96ウェルプレートにウェルあたり10,000細胞で播種しました。予備実験で決定された至適条件に従って、各コンポーネントを、0.3 µL /ウェルのDharmaFECT Duoトランスフェクション試薬を使用して細胞をトランスフェクトしました。トランスフェクトしたコンポーネントは、200 ngのEdit-R Cas9ヌクレアーゼmRNA、LMNAを標的とする25 nMの化学合成crRNA:tracrRNA、SEC61Bを標的とする25 nMの化学合成crRNA:tracrRNA、200 ngのEGFP-LMNAドナープラスミド、および200ng mKate2-SEC61Bドナープラスミドです。細胞を3回継代し、トランスフェクションの7日後にNikon倒立落射蛍光顕微鏡で画像化し、EGFP(緑)とmKate2(赤)の両方が挿入された単一細胞を同定しました。
U2OS細胞においてN末端がEGFPでタグ付けされたLMNAとN末端がmKate2でタグ付けされたSEC61B
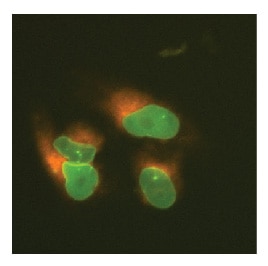
U2OS細胞に、Cas9 mRNA、LMNAを標的とする化学合成crRNA-tracrRNA、SEC61Bを標的とする化学合成crRNA:tracrRNA、相同性アームあたり約1000 bpの相同性を含むLMNAのN末端に特異的なEGFPドナープラスミド、および相同性アームあたり約1000 bpの相同性を含むSEC61BのN末端に特異的なmKate2ドナープラスミドを、DharmaFECT Duoを用いてトランスフェクトしました。必要に応じて細胞を継代し、トランスフェクションの7日後にInCell 2200ハイコンテント蛍光顕微鏡で画像化して、EGFPとmKate2の両方が挿入された単一細胞を同定しました。
Dharmacon Edit-R mKate2ノックインドナー
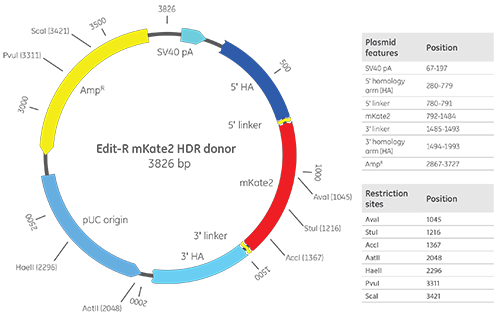
Edit-R mKate2プラスミドドナーマップと重要なベクター機能。この図では、例として500 bpの相同性アームが含まれています。
プラスミドドナークローニングワークフロー

コンポーネントの順序付け、相同性アームの増幅、およびプラスミドドナーの生成を含むプラスミドドナーのクローニングワークフロー。図の色は、DNAの由来を示しています(濃青 = 5' 相同性アーム、水色 = 3' 相同性アーム、赤 = mKate2蛍光レポーター配列、オレンジ = プラスミドバックボーン)。medium alone = 培地のみのネガティブPCRコントロール
コロニーPCRによる成功したクローニングの検出
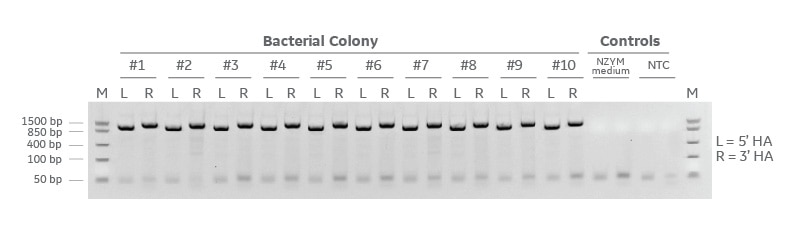
ユニバーサルバックボーン、EGFPインサート、および2つのPCRで生成された相同性アームを備えたEdit-Rドナープラスミドキットのギブソンクローニング結果。得られたクローニング反応をNEB 5-αケミカルコンピテントセルで形質転換し、37℃で一晩増殖させました。10個の細菌コロニーをランダムに選択し、コロニーPCRを実行して、各相同性アーム(L = 5' 相同性アームおよびR = 3' 相同性アーム)の存在を検出しました。コロニーPCRアンプリコンは、エチジウムブロマイドとFastRuler低分子量分子量マーカーを含む2%アガロースE-ゲルで実施しました。
mKate2ノックインクローン細胞株を作製するためのワークフロー
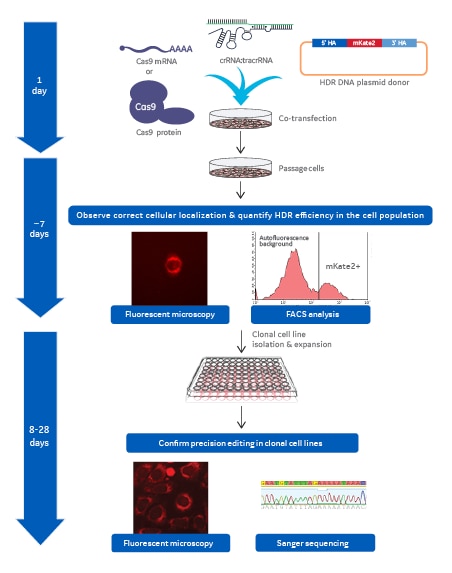
mKate2ノックインクローン細胞株を作製するために必要な主要なプロセスステップと一般的なタイミングを示すワークフロー
