- Dharmaconスクリーニングライブラリー
- CRISPRmod CRISPRa all-in-one lentiviral sgRNA 全ゲノムプール化ライブラリー
本ライブラリーフォーマットは、遺伝子導入が困難な細胞や初代細胞での遺伝子活性化に有効であり、効率的なゲノムスクリーニングを可能にすることで、遺伝子研究や治療法の発見を促進します。
プロモーター(mCMVおよびhEF1α)の選択により、目的の細胞における最適なdCas9-VPR発現が可能になります。
CCRISPRmod CRISPRaシステムは、CRISPRa sgRNAと転写活性化因子(VPR)が融合した切断活性が不活化されたCas9(dCas9)の2つのコンポーネントが必要です。当社のCRISPRa all-in-one ライブラリーは、ネイティブコンテキストでの過剰発現によって遺伝子の機能を研究するための簡便で効率的なツールです。
CRISPRmod CRISPRa ガイドRNAデザインは、公開されているCRISPRa v2アルゴリズム(Horlbeck et al 2016)を使用して設計されています。CRISPRa ガイドRNAは、標的遺伝子のプロモーター領域または転写開始部位(TSS: transcriptional start site)の上流の遺伝子配列を標的とし、転写活性化を促進します。
| CRISPRmod CRISPRa all-in-one lentiviralベクターマップ |
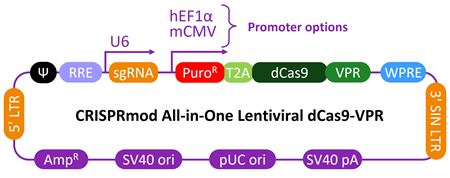 |
Highlights:
- dCas9-VPRおよび遺伝子特異的sgRNA発現の両方に単一のレンチウイルスベクターを使用する効率的な all-in-oneベクターシステム
- 効率的かつ特異的な遺伝子活性化をもたらす合理的に設計されたレンチウイルスsgRNA
- ヒトゲノム全体にわたり、1遺伝子あたり4または8個のsgRNAで深く広いカバレッジを実現し、ヒットの信頼性を向上
- 生物学的に関連性のある細胞タイプでロバストな活性化を行うための複数のプロモーターオプション
- 最小限の細胞毒性で直接導入するために濃縮、精製された高品質の ≥ 2 × 107 TU/mL(± 20%)レンチウイルス粒子
各CRISPRmod CRISPRa all-in-one lentiviral pooled screening libraryには、次のものが含まれています:
- ≥ 2 × 107 TU/mL(± 20%)レンチウイルス粒子、10 × 100 uL アリコート、mCMVまたはhEF1αプロモーターオプション付き
- ヒトゲノムのどの遺伝子ともアライメントしない(標的化しない)ことがバイオインフォマティックに確認された最大100個のnon-targeting gRNA ネガティブコントロール
- 遺伝子アノテーション、sgRNAターゲット配列、完全なコントロールリスト、数百万マップリードあたりのカウントを含む完全なライブラリー情報を含むデータファイル
当社の検証済みプロトコールで推奨されている製品:
- トランスダクションの最適化とプロモーター選択のためのベクター対応CRISPRa EGFPデリバリーコントロール
- NGS Hit Identification primers (mCMVまたはhEF1α ベクター用):
- バイアスを最小限に抑えたゲノムDNAの効率的なPCR増幅
- NGSランに多様性をもたらすスタッガードフォワードプライマー
- 部分的なIllumina®互換TruSeq®アダプターの追加
Edit-R Lentiviral sgRNA プール化ライブラリーのカバレッジに関する情報
遺伝子数およびコンストラクト数は予告なく変更される場合があります。クローン数および遺伝子数はご要望に応じてご提供いたします。Scientific Supporへお問い合わせください。
全ゲノムスクリーニングに必要なレンチウイルス粒子の量
| Fold representation | Replicates | sgRNAs per gene | Volume of lentiviral particles |
|---|---|---|---|
| 200 | 2 | 4 | 1.7 mL |
| 200 | 2 | 8 | 3.3 mL |
| 400 | 2 | 4 | 3.3 mL |
| 400 | 2 | 8 | 6.6 mL |
| NGS Library Prep Kit Contents | Small: 12 Whole Genome Screens | Large: 24 Whole Genome Screens |
|---|---|---|
| NEXTFLEX® PCR Master Mix (green cap) | 3600 μL | 7200 μL |
| Dharmacon HIT Identification Primers (orange cap) | 300 μL | 600 μL |
| NEXTFLEX® PCR II Barcoded Primer Mix | 4 μL | 4 μL |
| Resuspension Buffer | 12 mL | 24 mL |
| Nuclease-free water | 8 mL | 16 mL |
| Cleanup Beads | 7 mL | 15 mL |
CRISPRmod CRISPRa all-in-one lentiviral プール化ライブラリーを用いた遺伝子過剰発現スクリーニングワークフロー
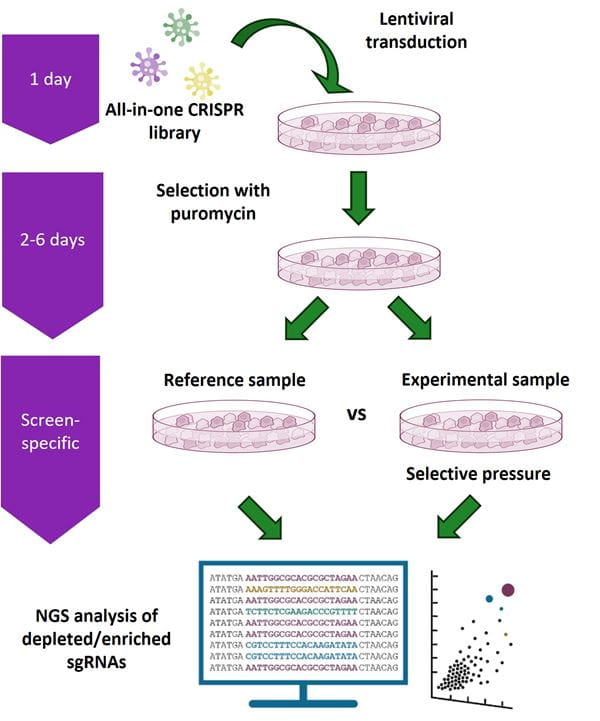
細胞にCRISPRmod CRISPRa all-in-one lentiviral プール化ライブラリーを低い多重感染度(MOI)で形質導入した後にピューロマイシンで選抜します。形質導入された細胞は、選択圧および/または表現型選択を適用するために対照集団と実験集団に分けます。次に、形質導入を行った実験細胞集団と対照細胞集団からゲノム DNA を単離して、ゲノムDNA 内の sgRNA コンストラクトをベクター特異的 NGS Hit Identification Primer を用いて増幅します。アンプリコンは、市販のIllumina TruSeq®プレップキットを用いて、Illuminaのプラットフォーム上でハイスループットシーケンス用にインデックス化および調製できます。対照サンプルと実験サンプルの両方に統合されたsgRNA配列を同定し、相対的な存在量を比較します。濃縮または枯渇したsgRNAコンストラクトがヒットとして同定され、個別のCRISPRmod CRISPRa all-in-one lentiviral sgRNAを用いて追加の表現型および/または生化学的アッセイでさらに確認および研究することができます。
高品質のプール化スクリーニングは、厳格なレンチウイルスプールライブラリーの作製から始まる
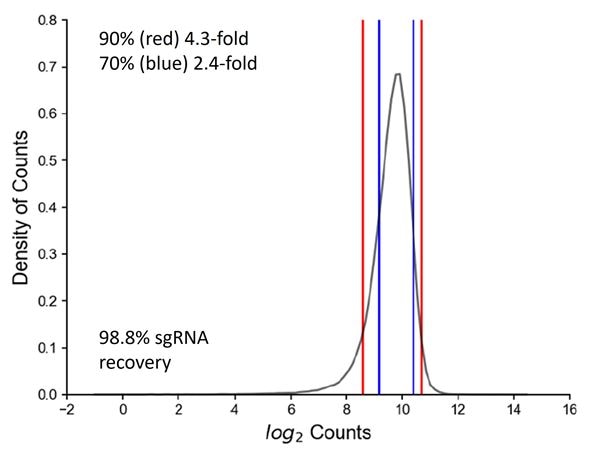
高品質のプールスクリーニングは、厳格なレンチウイルスプールライブラリーの作製から始まります。遺伝子あたり4つのレンチウイルスsgRNAからなるCRISPRmod CRISPRa Human All-in-one Lentiviral Whole Genome Pooled Libraryを作製し、次世代シーケンス(NGS)によりプラスミドDNAライブラリーの品質を検証しました。マップされた100万リードあたりのカウント数から、インプットsgRNAの回収率(99.8%)と、プール中の90%と70%のsgRNAの分布がそれぞれ4.3倍と2.4倍の範囲内であることがわかりました。
適合した蛍光デリバリーコントロールによりスクリーニング前の迅速な機能力価の確認と最適なプロモーターの選択が可能
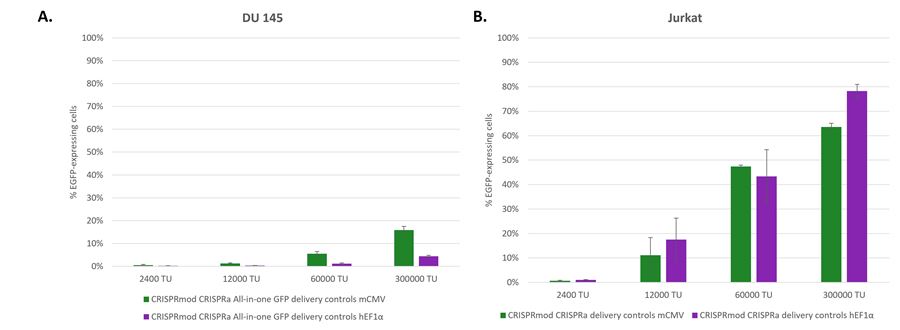
適合した蛍光デリバリーコントロールにより、スクリーニング前に迅速な機能的力価の確認と最適なプロモーターの選択が可能です。DU145細胞(A)およびJurkat細胞(B)に、HEK293細胞におけるの機能的力価単位(TU)を持つCRISPRmod CRISPRa all-in-one GFP delivery control mCMV(緑)およびhEF1α(紫)の5倍希釈系列を形質導入しました。導入後72時間にEGFP発現細胞の割合をフローサイトメトリーで定量しました。5~25%のEGFP発現細胞を示す希釈液は、目的の細胞株における相対的な機能力価および細胞株の相対的な導入効率を計算するために使用することができます。ある希釈度(5~25%のEGFP発現細胞を示す)におけるEGFP発現細胞の割合または平均蛍光強度を用いて、細胞株におけるプロモーター活性を比較することができます。どちらのプロモーターもJurkat細胞では同様の活性を示しますが、DU145細胞ではmCMVプロモーターの方がhEF1αプロモーターよりもかなり活性が高いことがわかります。
CRISPRmod all-in-one dCas9-VPRシステムおよびSynergistic Activation Mediator (SAM)システムを使用して実施された全ゲノムCRISPRaスクリーニング間で、高い一致率でヒットが検出された
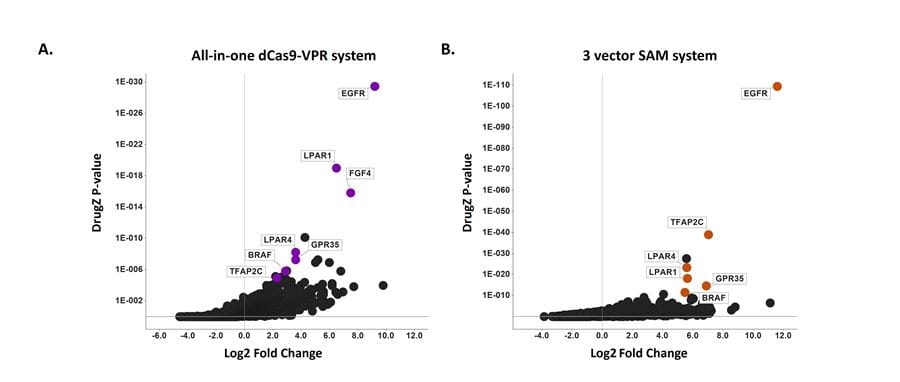
16日間ベムラフェニブ(PLX-4032)で処理したA375メラノーマ細胞において、CRISPRmod CRISPRa Human All-in-one lentiviral sgRNA全ゲノムプール化ライブラリー(A)、または3ベクターSynergistic Activation Mediatorシステム(B)を用いたスクリーニング間で、非常に一致性の高い耐性ヒットが検出されました。DrugZ スクリーニング分析による各遺伝子のlog2倍加濃縮と関連p値を示しています。有意に濃縮された遺伝子(正の Log2 倍加変化)は、薬剤治療に対する耐性を引き起こし、これらの遺伝子が薬剤の拮抗薬として作用する可能性を示しています。ハイライトされたヒットは、両方の技術で特定され、CRISPRa スクリーニングで以前に検証されています(Konermann et al., 2015)。
