- Dharmaconスクリーニングライブラリー
- Edit-R human all-in-one lentiviral sgRNA 全ゲノムプール化ライブラリー
Edit-R All-in-one sgRNA プール化レンチウイルスライブラリーを1回形質導入するだけで、数千の遺伝子をDNAレベルで簡単にノックアウト可能
このライブラリーフォーマットは、トランスフェクションが困難な細胞や初代細胞でノックアウトを行うのに有効であり、効率的なゲノムスクリーニングを可能にし、遺伝子研究や治療法の発見を推進します。
プロモーター(mCMVとhEF1α)の選択により、目的の細胞での最適なCas9ヌクレアーゼ発現が可能になります。
CRISPRガイドRNAが標的DNAに二本鎖切断を生じさせ、機能的タンパク質の破壊を引き起こす能力は、ガイドRNA(gRNA)の配列と標的遺伝子内の位置によって異なります。この問題に対処するため、Dharmacon Edit-Rアルゴリズムは、ヒトゲノム全体にわたって非常に特異的で機能的なRNAガイドを提供します。
| Edit-R all-in-one lentiviral sgRNAのベクターマップ |
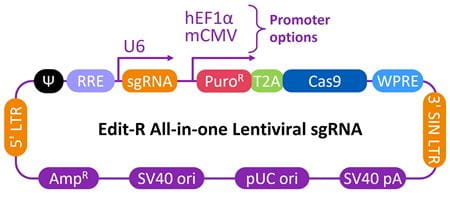 |
Edit-R CRISPR RNAアルゴリズムは、ガイドRNA配列を生成します:
- 複数のミスマッチやギャップのあるアラインメントの検出を含む、迅速かつ徹底的な特異性分析を備えた最適化されたアラインメントプログラム
- 単なるindelの生成ではなく、遺伝子の機能的破壊に関連する特性を特定するための体系的な評価研究(1100を超えるガイドRNAを使用)により、標的遺伝子の機能的ノックアウトが増加
- New! Edit-RヒトsgRNAデザインは2025年最新版RefSeqに更新されました。これにより、効率的なタンパク質ノックアウトを実現するための、最も特異的でゲノム関連性の高いガイドが提供されます。この更新により、Edit-Rアルゴリズムは最新のゲノムアノテーションをより正確かつ効率的に標的とすることが可能となり、ご研究ニーズに最適なソリューションを提供します。ご質問がございましたら、Scientific Supportまでお問い合わせください。
Edit-R CRISPR RNA アルゴリズムを用いて、プール化レンチウイルススクリーニングに必要なシングルコピーの組込みレベルで高効率な遺伝子ノックアウトを示すレンチウイルス sgRNA を設計しました。
Highlights:
- Cas9と遺伝子特異的sgRNA発現の両方に単一のレンチウイルスベクターを使用する効率的なall-in-oneベクターシステム
- 機能的に検証された独自のアルゴリズムを用いて合理的に設計されたEdit-RレンチウイルスsgRNAが、比類のない特異性で効率的な遺伝子ノックアウトを実現
- ヒトゲノム全体にわたり、1遺伝子あたり4または8個のsgRNAで深く広いカバレッジを実現し、ヒットの信頼性を向上
- 生物学的に関連性のある細胞タイプで強固な編集を行うための複数のプロモーターオプション
- 最小限の細胞毒性で直接導入するために濃縮、精製された高品質レンチウイルス粒子。力価2 x 107 TU/mL以上。
各Edit-R All-in-one lentiviral sgRNA プールドスクリーニングライブラリーには、次のものが含まれています:
- ≥ 2×107TU/mL(±20%)レンチウイルス粒子、10×100µLアリコート、mCMVまたはhEF1αプロモーターオプション付き
- NCBI参照配列データベースのコーディング遺伝子をターゲットとする遺伝子あたり4または8個のsgRNA
- ヒトゲノムのどの遺伝子ともアライメントしない(標的化しない)ことがバイオインフォマティックに確認された100個の非標的sgRNAネガティブコントロール
- 遺伝子アノテーション、sgRNAターゲット配列、完全なコントロールリスト、数百万マップリードあたりのカウントを含む完全なライブラリー情報を含むデータファイル
当社の検証済みプロトコールで推奨されている製品:
- トランスダクションの最適化とプロモーター選択のためのベクター対応Edit-R EGFPデリバリーコントロール
- 新製品:NGSライブラリー調製キット(推奨)
- hEF1αおよびmCMVプロモーター用のプライマーセットオプションが利用可能。12または24の全ゲノムスクリーニングに対応する2種類のサイズから選択可能
- gDNAからsgRNAコンストラクトを偏りなく増幅およびシーケンスするようにデザイン
- 追加試薬を必要とせず、Illumina®プラットフォームでのシーケンシングに対応する完全なソリューション
Edit-R Lentiviral sgRNA プール化ライブラリーのカバレッジに関する情報
遺伝子数およびコンストラクト数は予告なく変更される場合があります。クローン数および遺伝子数はご要望に応じてご提供いたします。Contact Scientific Support までお問い合わせください。
全ゲノムスクリーニングに必要なレンチウイルス粒子の量
| Fold representation | Replicates | sgRNAs per gene | Volume of lentiviral particles |
|---|---|---|---|
| 200 | 2 | 4 | 1.6 mL |
| 200 | 2 | 8 | 3.1 mL |
| 400 | 2 | 4 | 3.1 mL |
| 400 | 2 | 8 | 6.1 mL |
| NGS Library Prep Kit Contents | Small: 12 Whole Genome Screens | Large: 24 Whole Genome Screens |
|---|---|---|
| NEXTFLEX® PCR Master Mix (green cap) | 3600 μL | 7200 μL |
| Dharmacon HIT Identification Primers (orange cap) | 300 μL | 600 μL |
| NEXTFLEX® PCR II Barcoded Primer Mix | 4 μL | 4 μL |
| Resuspension Buffer | 12 mL | 24 mL |
| Nuclease-free water | 8 mL | 16 mL |
| Cleanup Beads | 7 mL | 15 mL |
Edit-R All-in-one Lentiviral Pooled Libraryプラットフォームを用いた遺伝子ノックアウトスクリーニングワークフロー
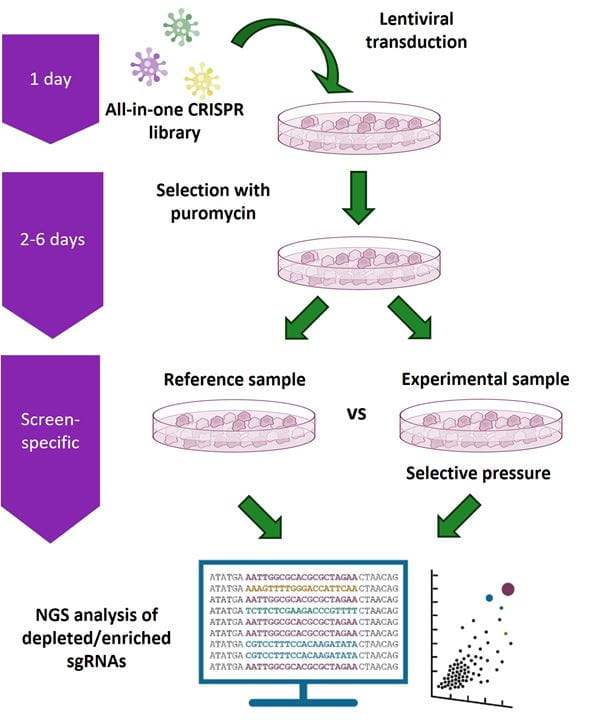
細胞にEdit-R All-in-one Lentiviral Pooled Libraryを低い多重感染度(MOI)で形質導入した後にピューロマイシンで選抜しました。形質導入された細胞は、選択圧および/または表現型選択を適用するために対照集団と実験集団に分けられます。次に、形質導入を行った実験細胞集団と対照細胞集団からゲノム DNA を単離して ゲノムDNA 内の sgRNA コンストラクトをベクター特異的なNGSライブラリー調製キットを用いて増幅します。その後、サンプルをマルチプレックス化し、Illumina®プラットフォーム上でシーケンシングが可能です。カスタムシーケンシングリードプライマーは不要です。コントロールサンプルと実験サンプルの両方に統合されたsgRNA配列を同定し、相対的な存在量を比較しました。濃縮または枯渇したsgRNAコンストラクトがヒットとして同定され、個別のEdit-R All-in-one Lentiviral sgRNAを用いて追加の表現型および/または生化学的アッセイでさらに確認および研究することができます。
高品質のプール化スクリーニングは、厳格なレンチウイルスプール化ライブラリーの製造から始まる
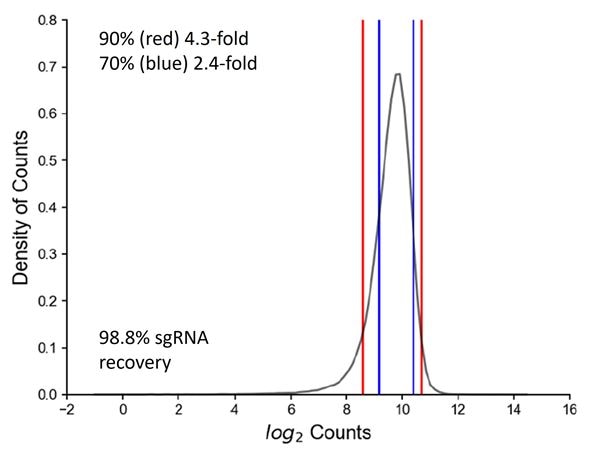
高品質のプールスクリーニングは、厳格なレンチウイルスプールライブラリーの作製から始まります。遺伝子あたり4つのレンチウイルスsgRNAからなるEdit-R Human All-in-one Lentiviral Whole Genome Pooled Libraryを作製し、次世代シーケンス(NGS)によりプラスミドDNAライブラリーの品質を検証しました。マップされた100万リードあたりのカウント数から、インプットsgRNAの回収率(99.8%)と、プール中の90%と70%のsgRNAの分布がそれぞれ6.5倍と3.2倍の範囲内であることがわかりました。
適合した蛍光デリバリーコントロールによりスクリーニング前の迅速な機能力価の確認と最適なプロモーターの選択が可能

適合した蛍光デリバリーコントロールにより、スクリーニング前に迅速な機能的力価の確認と最適なプロモーターの選択が可能。DU145細胞(A)およびJurkat細胞(B)に、HEK293細胞由来の機能的力価単位(TU)を持つEdit-R GFPデリバリーコントロールmCMV(緑)およびhEF1α(紫)の5倍希釈系列を形質導入しました。導入後72時間にEGFP発現細胞のパーセンテージをフローサイトメトリーで定量しました。5~25%のEGFP発現細胞を示す希釈液は、目的の細胞株における相対的な機能力価および細胞株の相対的な導入効率を計算するために使用することができます。ある希釈度(5~25%のEGFP発現細胞を示す)におけるEGFP発現細胞のパーセンテージまたは平均蛍光強度を用いて、細胞株におけるプロモーター活性を比較することができます。どちらのプロモーターもJurkat細胞では同様の活性を示しますが、DU145細胞ではmCMVプロモーターの方がhEF1αプロモーターよりもかなり活性が高いことがわかります。
ゲノム編集効率と相関するEdit-Rの機能スコア
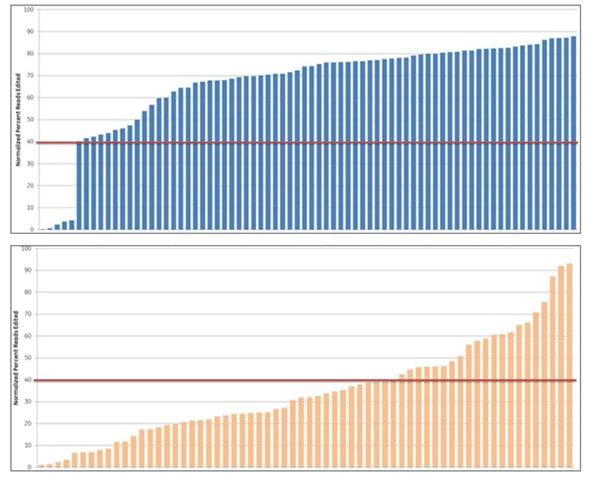
アルゴリズムスコアと編集効率との相関(indel形成のNGS解析)。Cas9を安定的に発現するHEK293T細胞に、10個の遺伝子を標的とするアルゴリズムスコアが最も高い10個のcrRNA(青色)とアルゴリズムスコアが最も低い10個のcrRNA(オレンジ色)をトランスフェクションし、次世代シーケンサーで編集を評価しました。Cas9-HEK293T細胞株を50 nM crRNA:tracrRNAでトランスフェクションしました。トランスフェクションから72時間後、細胞を溶解し、各 crRNA 部位にわたる Nextera のトランスポゾンアダプトアンプリコンを、各処理サンプルおよびトランスフェクションされていないサンプルからの対応するコントロールのアンプリコンに対して調製しました。。サンプルはNextera 96-well index kitを使用してインデックス化し、MiSeq装置でのシーケンス用にプールしました(ペアエンドリード、長さ2 x 300)。NGS品質フィルター基準をパスしたリードを参照ファイル(Bowtie2 v2.1.0)にアライメント。完全リードの割合を計算し、トランスフェクションしていないコントロールサンプルに対して正規化しました(Samtools v0.1.12a)。
高いEdit-R機能性スコアは機能的遺伝子ノックアウトの増加と相関
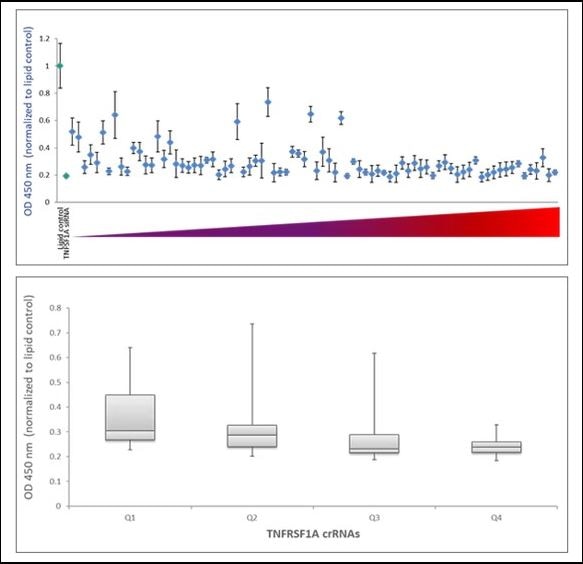
可溶性TNFRSF1AのELISAアッセイによるTNFRSF1A crRNAの機能性の検討
アルゴリズムによって設計されたgRNAが、タンパク質レベルで効率的な機能遺伝子ノックアウトを予測する能力を調べるために、特定の機能アッセイにおける標的遺伝子のタンパク質レベルまたは活性に対する効果とアルゴリズムスコアの相関性を検討しました。まず、TNFRSF1A (TNFRSF1A タンパク質の可溶性部分をコードする) を標的とする 79 個の gRNA について、TNFRSF1A ELISA アッセイで測定した TNFRSF1A タンパク質レベルの減少を引き起こす機能性を調べました。上段図は、細胞培地中の TNFRSF1A タンパク質レベルに対する、アルゴリズムが予測した機能性スコアで並べた全crRNAの効果を示しています。アルゴリズムスコアが高いcrRNAは、標的遺伝子タンパク質レベルの低下として測定される高い機能的遺伝子破壊を示します。低スコアのcrRNAでは、機能性のばらつきが大きく、多くのガイドが機能的遺伝子ノックアウトの能力が低いことを示しています(上段図)。これは、アルゴリズムスコアに基づき、低スコアから高スコアまで4つの四分位に分けられたcrRNAの機能性のボックスプロットによって明確に描かれています。中央値、下位四分位値と上位四分位値間のデータ分布、および最小値と最大値は、タンパク質ノックアウトアッセイにおいてアルゴリズムで設計された高スコアのcrRNAが機能性を高めていることを示しています(下段図)。
上段図:Cas9(CAGプロモーター下)を組み込んだU2OS-プロテアソーム細胞を96ウェルプレートに10,000細胞/ウェルでプレーティング。プレーティングから24時間後、50nM crRNA:tracrRNA を0.2 μg/ウェルのDermaFECT4を用いて細胞にトランスフェクションしました。TNFRSF1Aを標的とするsiRNAをポジティブコントロールとして用い、データをネガティブコントロール(脂質のみ)に対して正規化しました。トランスフェクションから72時間後に細胞培地を回収し、Quantikine Human sTNF RI/TNFRSF1A Quantikine ELISA Kit (R&D Systems)を用いて可溶性TNFRSF1Aをアッセイしました。
下段図:可溶性TNFRSF1AのELISAアッセイにおけるTNFRSF1A crRNAの機能性のボックスプロット。crRNAは、低から高までのアルゴリズムスコアに基づいて4つの四分位(Q1、Q2、Q3、Q4)に分けられています。
Edit-R Human All-in-one Lentiviral Pooled Libraryを用いた全ゲノムスクリーニングにより、Vemurafenibを投与したA375細胞において信頼性の高い耐性ヒットを同定
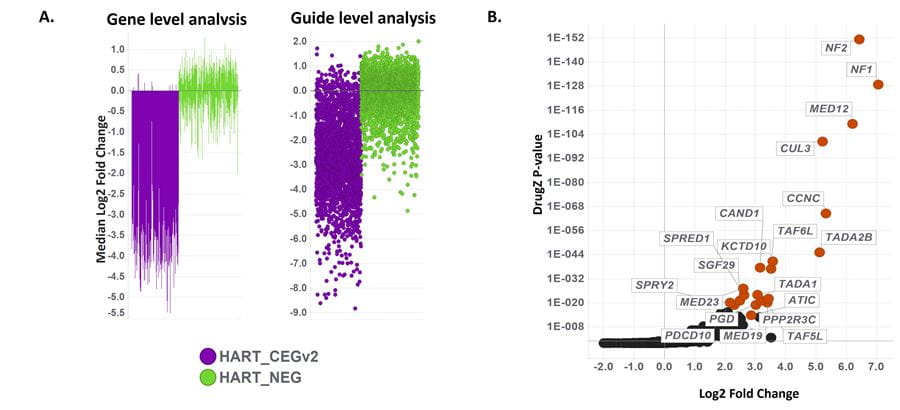
ベムラフェニブ(PLX-4032)で14日間処理したA375メラノーマ細胞におけるEdit-R Human All-in-one Lentiviral Pooled Libraryを用いた高信頼性耐性ヒットと明確な必須遺伝子の脱落。(A) T0と比較したスクリーニング終点における、公表されたコア必須遺伝子(HART_CEGv2、紫色)および非必須遺伝子(HART_NEG、緑色)の遺伝子およびガイドレベル解析(Hart et al., 2017)非必須遺伝子を標的とするガイドは中立的な挙動を示す一方、必須遺伝子を標的とするガイドは、これらのガイドを発現する細胞が生存できないはずであるため、スクリーニング中に脱落しました。(B)各遺伝子の濃縮(Log2 Fold Change)と関連するp値を示すDrugZスクリーン解析。有意に濃縮された遺伝子(正のLog2 Fold Change)は、薬物治療に対する耐性を促進しこれらの遺伝子が潜在的に薬物応答を増強することを示しています。Drug Z P値による上位20ヒットをラベル付けして強調表示しました。
Edit-R Human All-in-one Lentiviral Pooled Libraryを用いた全ゲノムスクリーニングにより、Vemurafenibを投与したA375細胞において信頼性の高い耐性ヒットを同定
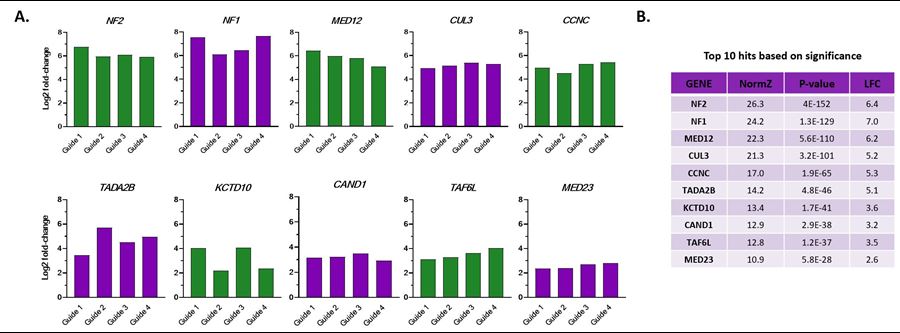
(A) CRISPR ノックアウト・ベムラフェニブ・スクリーニングからの DrugZ 解析で、上位 10 遺伝子レベルの各ヒットを標的とした各ガイドの対数 2 倍の濃縮。 (B) 関連する遺伝子レベルのNormZスコア、P値、Log2 Fold Change(LFC)の表。

