単一遺伝子の過剰発現実験は、個々のloss-of-function研究で得られた情報を完全に補完すると長い間考えられてきました。一般的な戦略の1つは、ORFを使用して、loss-of-function効果の影響を受けない目的の遺伝子のエクストラコピーを発現させることで、レスキュー実験として知られています。しかし、大規模なスクリーニングアプリケーションにレンチウイルスORF発現アプローチを活用することは論理的な考えのように思われますが、そのような計画には多くの予期しない問題があることがわかります。ここでは、ハイスループットORFスクリーニングを設定する際の最大のハードルと、アレイ化されたCRISPRaアプローチがより優れた戦略となる方法について説明します。
等しいrepresentationはライブラリー作製の鍵である
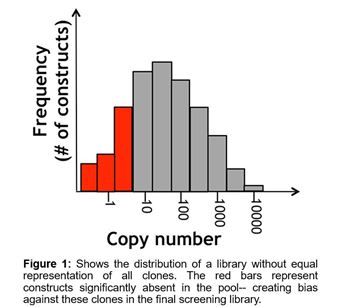
効果的なレンチウイルススクリーニングライブラリーは、すべてのコンストラクトが均一にrepresentationする方法で構築する必要があります。レンチウイルスORFライブラリーの場合、これは特に難しい場合があります。なぜなら、サイズの異なるORFインサートがさまざまな効率でパッケージ化されることにより、より短い配列に偏る傾向があるためです。これを試みる場合は、プールまたはウェル内の個々のコンストラクトをキャリブレーションして、特定のシーケンスに偏ったり離れたりしないようにする方法が必要になります(図1を参照)。これは、時間経過と凍結融解サイクルに依存して力価が予測できないほど低下するレンチウイルスの性質を考慮すると課題です。不均一な材料を使用する場合の最大の懸念は、偽ヒットの発生率が高くなり、スクリーニング間の再現性が低下する可能性があることです。どちらも、研究者にとって多大なフォローアップの無駄をもたらす可能性があります。
データのデコンボリューションのハードルに注意が必要
考慮すべきもう1つのポイントは、結果のデータをどのようにデコンボリューションするかです。スクリーニングアッセイがアレイフォーマットで実施される場合、堅牢な表現型の読み出しが開発されている限り、どのコンストラクトが個々のウェルに関連付けられているかを簡単に特定できます。ただし、レンチウイルスORFアレイプラットフォームの力価がウェルごとに異なる場合は、感染多重度(MOI:multiplicity of infection)の変動によって引き起こされる偽陽性または偽陰性を考慮する必要があります。この変動を制御するために使用された過去の戦略には、複雑なハイコンテント画像分析(参考文献2)が含まれていました。この分析方法は、全ての研究者がスクリーニング時のアウトプットに適用できるとは限りません。
プール化レンチウイルスフォーマットでスクリーニングを実行する場合、潜在的なヒットを特定するために次世代シーケンシングとバイオインフォマティクス分析が必要になります。これには、ライブラリー作製者が、参照FASTAファイルを設定するためにORFをバーコードベクターにサブクローニングする戦略、または配列のサイズと塩基配列の変更が可変であることが必要ですが、いずれの場合も集団内の特定のORFを特定することが難しいです。
解決策として:CRISPRa
CRISPRaライブラリーは、ハイスループットのgain-of-function実験に使用でき、上記の落とし穴を回避できる(レンチウイルスに代わる)代替ツールです。化学合成RNAテクノロジーにより、ウェル間で一貫したストック濃度を確実に得ることができます。細胞へのデリバリーは単純なトランスフェクションであるため、プレート内または複数のプレート間での均一なパフォーマンスが確実になります。CRISPRaライブラリーシステムはアレイ化フォーマット(ウェルごとに1つの遺伝子に対応するcrRNAを分注)で提供され、プール化フォーマットよりも簡単なスクリーニングデコンボリューションとより複雑な表現型アッセイを可能にします(参考文献3)。個々のレンチウイルスsgRNAコンストラクトは、このアプローチを必要とする細胞または実験にも利用できます。細胞のネイティブDNAから直接遺伝子を発現させることにより、アイソフォームが細胞生物学に関連しており、プロモーター機能、選択マーカーなどに関連する課題を打ち消すことを確信できます。CRISPRa crRNAライブラリーは、遺伝子ファミリーとパスウェイを幅広く選択することから全ゲノムまでで利用でき、カスタムのCherry-pickガイドRNAライブラリーも簡単に作製できます。したがって、クローニングや、プール化レンチウイルスライブラリーの検証などの、時間とコストのかかる手順を排除し、重要な結論と決定ポイントまで研究をスピードアップします。
Written by Jennifer Abarca PhD, Lead Scientific Support Specialist
Jennifer Abarcaは、10年以上にわたり、当社の製品ポートフォリオに関するお客様からの質問をサポートしてきました。彼女は、CRISPRaが、最近導入された最もエキサイティングなテクノロジーの1つであると信じています。「何年もの間、gain-of-functionスクリーニングを実行するためのより簡単な方法を要求する研究者がいました。このCRISPRaプラットフォームは、レンチウイルスおよびORFベースの方法に関係する問題を解消するソリューションを最終的に提供します。」Jenniferは、顧客に”実験の旅”のガイドをしていないときは、地元のコミュニティガーデンのディレクターを務めることで、近隣とのの友情を築くことを楽しんでいます。
Resources
- CRISPRa, cDNA, or ORF?- Featured Article
Featured Products
References
- Vermeulen, et al. Pooled shRNA Screening. Frontiers in RNAi, Vol. 1, 2014, 58-78
- Scaronkalamera D., Dahmer-Heath M., Stevenson A. J., Pinto C., Shah E. T., Daignault S. M., Said N. B.M., Davis M., Haass N. K., Williams E. D., Hollier B. G., Thompson E. W., Gabrielli B., et al Genome-wide gain-of-function screen for genes that induce epithelial-to-mesenchymal transition in breast cancer. Oncotarget. 2016; 7: 61000-61020. Retrieved from https://www.oncotarget.com/article/11314/text/
- Žaklina Strezoska, et al. CRISPR-mediated transcriptional activation with synthetic guide RNA. Journal of Biotechnology. Volume 319, 25-35. (2020). https://doi.org/10.1016/j.jbiotec.2020.05.005