Edit-R lentiviral sgRNAポジティブコントロールおよびキット
DNA二本鎖切断とゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用するコントロール
検証済みのEdit-R lentiviral sgRNAポジティブコントロールおにより、挿入および欠失(indel)生成を確認し、DNAミスマッチ検出アッセイを利用してゲノム編集効率を定量することができます。

ゲノム編集イベントの検証のための、検証済みのlentiviral sgRNAポジティブコントロールおよびキット
Edit-R lentiviral sgRNAポジティブコントロールおよびキットは、ヒトまたはマウスの遺伝子を標的として設計されており、簡易なDNAミスマッチ検出アッセイを使用して標的ゲノムDNAの挿入および欠失(indel)の生成を観察することにより、形質導入が適切に行われたことを確認し、ゲノム編集実験の検証をすることができます。
個々のEdit-R lentiviral sgRNAポジティブコントロールは、グリセロールストックおよび精製濃縮レンチウイルス粒子(50 µL、1x108 TU/mL)としてご用意しています。ヒトまたはマウスで使用可能で、以下に対して設計されています。
- Cyclophilin B (PPIB)
- DNA (cytosine-5)-methyltransferase 3B (DNMT3B)
Edit-R lentiviral sgRNAポジティブコントロールキットには以下が含まれます。
- レンチウイルス粒子またはグリセロールストック
- DNAミスマッチ検出用プライマー(フォワードおよびリバース)
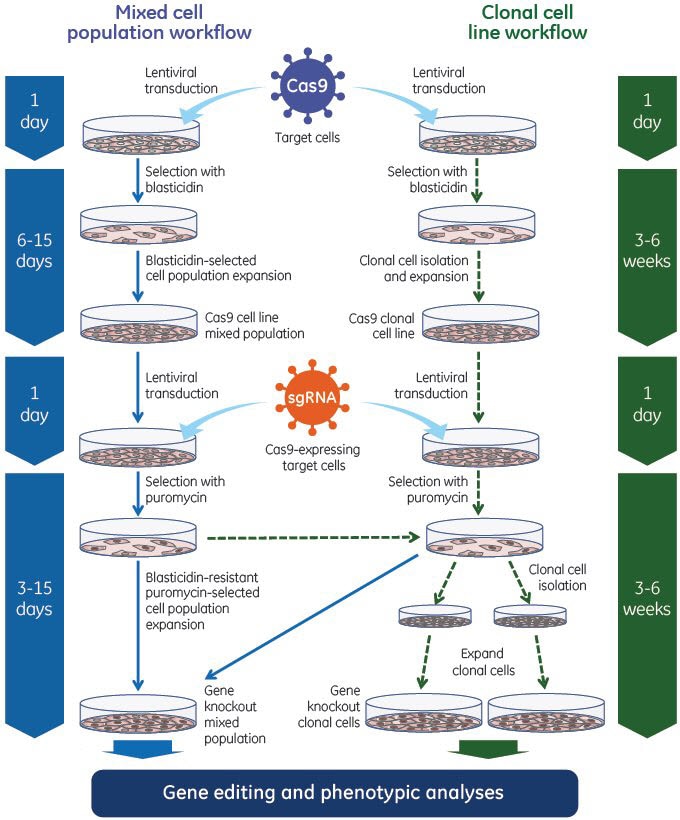
Edit-R lentiviral CRISPR-Cas9ゲノム編集プラットフォームには、天然のS. pyogenesシステムに基づく2つの重要なコンポーネント(Cas9発現用のベクター、目的の標的部位に合わせて設計されたsgRNA発現用の遺伝子特異的ベクター)が含まれています。Cas9ヌクレアーゼとsgRNAを構成的に発現する細胞株の迅速な作製を促進するために、Cas9およびsgRNAベクターは精製および濃縮されたレンチウイルス粒子にあらかじめパッケージ化されています。Cas9発現細胞株は、Edit-R lentiviral Cas9ヌクレアーゼ発現粒子を使用して簡単に作製でき、sgRNAを追加導入することでその後の遺伝子ノックアウトを得ることができます。
Edit-R lentiviral Cas9ヌクレアーゼ(A)とsgRNA(B)ベクターのベクターマップとベクターエレメント

Edit-R lentiviral CRISPR-Cas9プラットフォームは、Cas9発現を最大化するための複数のPol IIプロモーターオプションを持つレンチウイルスベクターと、目的の標的部位に設計されたsgRNA発現用の遺伝子特異的ベクターを利用する2ベクターシステムです。Edit-R lentiviral sgRNAベクターバックボーンでは、遺伝子特異的crRNAとtracrRNAがヒトU6プロモーターの制御下で発現され、一方、ピューロマイシン耐性マーカー(PuroR)の発現はマウスCMVプロモーターから駆動され、統合sgRNAを持つ細胞の迅速な選択を可能にします。
Edit-R lentiviral sgRNAベクターのプラスミドベクターマップ
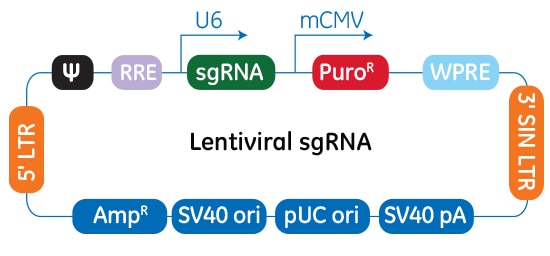
Edit-R lentiviral sgRNAベクターバックボーンでは、遺伝子特異的なcrRNAとtracrRNAがヒトU6プロモーターの制御下で発現します。一方、ピューロマイシン耐性マーカー(PuroR)の発現はマウスCMVプロモーターから駆動され、sgRNAが組み込まれた細胞の迅速な選択を可能にします。このプラスミドには、大腸菌での増殖と選択のためのAmpR耐性マーカーが含まれています。
Edit-R lentiviral sgRNAポジティブコントロールを使用した、異なるプロモーターの制御下におけるCas9の発現の違いによる、HEK293T細胞におけるさまざまなレベルのゲノム編集の検出
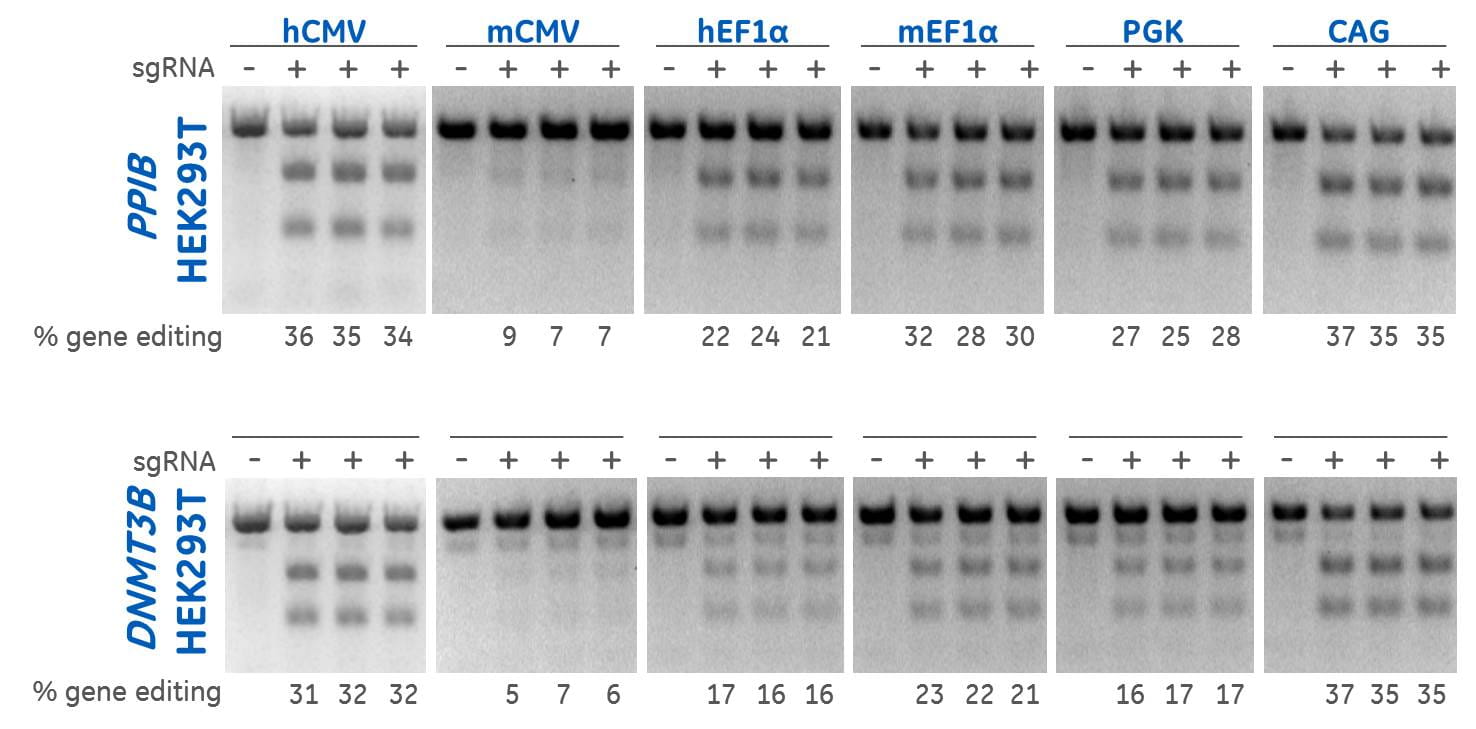
Edit-R lentiviral sgRNAポジティブコントロールとミスマッチ検出アッセイを使用して、HEK293T細胞におけるゲノム編集の効率をテストすると、hCMVおよびCAGプロモーターが最も高いレベルの編集をもたらすことが示されました。HEK293T細胞を、Cas9とブラステイジン耐性遺伝子を含むレンチウイルス粒子で安定的に形質導入しました。安定的に組み込まれた細胞集団は、sgRNAで形質導入する前に、最低10日間、blasticidinで選択しました。ポジティブコントロールsgRNAレンチウイルス粒子を低MOIで形質導入し、1つのインテグラントを持つ細胞を得、解析前に7日間ピューロマイシンで選択しました。T7エンドヌクレアーゼIを用いたDNAミスマッチ検出アッセイから、ピューロマイシン選択細胞におけるゲノム編集の相対頻度を算出しました。
Application notes
Protocols
Safety data sheets
Related Products
バイオインフォマティクスに基づきヒトまたはマウスのゲノムのどの遺伝子も標的にしないように設計および検証されたレンチウイルスsgRNAコンストラクト
