lentiviral Cas9ヌクレアーゼ試薬
生物学的に関連する細胞タイプを用いた確かなゲノム編集のための、レンチウイルスCRISPR-Cas9コンポーネント
Cas9ヌクレアーゼ安定発現細胞集団を作製するためのレンチウイルス粒子またはプラスミドDNAをご用意しています。大容量製品の価格についてはお問い合わせださい。
Try our new: Dharmacon™ Strict-R™ Inducible Cas9 Lentiviral System for dual controlled gene knockout in diverse cell types.

CRISPR-Cas9システムにおけるCas9ヌクレアーゼ
CRISPR関連酵素Cas9は、ゲノムDNAの標的認識とDNA二重鎖切断(double-strand breaks: DSB)のためにガイドRNAを必要とするRNA-guidedエンドヌクレアーゼです。
lentiviral Cas9試薬は、Cas9ヌクレアーゼを発現する細胞株の迅速な作製を容易にし、プール化lentiviral sgRNAスクリーニング、crRNAと共に使用するアレイ化スクリーニング、または単一遺伝子および/または複数の遺伝子を標的とする複数のガイドRNAの評価など、多くのゲノム編集アプリケーションに使用されています。
lentiviral Cas9ヌクレアーゼ発現ベクターには、S. pyogenes Cas9ヌクレアーゼのヒトコドン最適化バージョンを、さまざまな構成的または誘導的プロモーターの制御下が含まれています。全てのフォーマットは、精製されたレンチウイルス粒子またはプラスミドDNAとしてご提供しています。
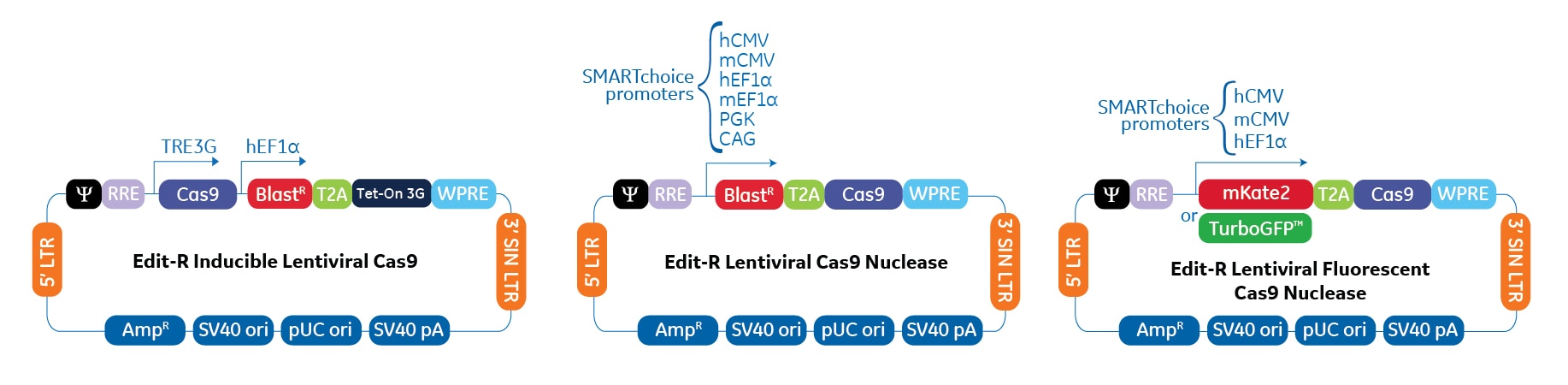
lentiviral Cas9 expression vector highlights
- ブラストサイジン耐性マーカー(BlastR)または蛍光マーカー(mKate2 または TurboGFP™)をを用いた選択が可能です。
- 即時形質導入が可能な濃縮精製レンチウイルス粒子であり、最小1x107 TU/mLの機能力価です。
- お客様独自のレンチウイルス粒子を生産するためのパッケージング細胞株への直接トランスフェクション用に、認証されたエンドトキシンフリープラスミドDNAとしてもご利用いただけます。
- 6 つのSMARTchoice構成的プロモーターのいずれかを使用してコンストラクトをカスタマイズし、目的の細胞株でのCas9の発現を最適化できます。
- Cas9の発現を一時的に制御する必要がある場合、またはバックグラウンド発現を最小限に抑えた安定した細胞株を作製する場合は、誘導型Cas9ベクターの厳密な制御をご利用いただけます。
すべてのRNA pol IIプロモーターが異なる細胞環境で同等にアクティブではない
Cas9ヌクレアーゼの転写を制御する任意のプロモーターの活性は、生物系によって大きく異なる可能性があり、Cas9の発現レベルが変化し、ひいてはDNA編集のレベルが変化します。したがって、細胞株または細胞タイプに最適なプロモーターを選択することは、実験におけるゲノム編集の程度に影響します。
Cas9ヌクレアーゼを発現するためのSMARTchoiceプロモーターオプション| Promoter | Description |
|---|---|
| hCMV | human cytomegalovirus immediate early promoter |
| mCMV | mouse cytomegalovirus immediate early promoter |
| hEF1α | human elongation factor 1 alpha promoter |
| mEF1α | mouse elongation factor 1 alpha promoter |
| PGK | mouse phosphoglycerate kinase promoter |
| CAG | chicken beta actin hybrid promoter |
| TRE3G | doxycycline-inducible promoter |
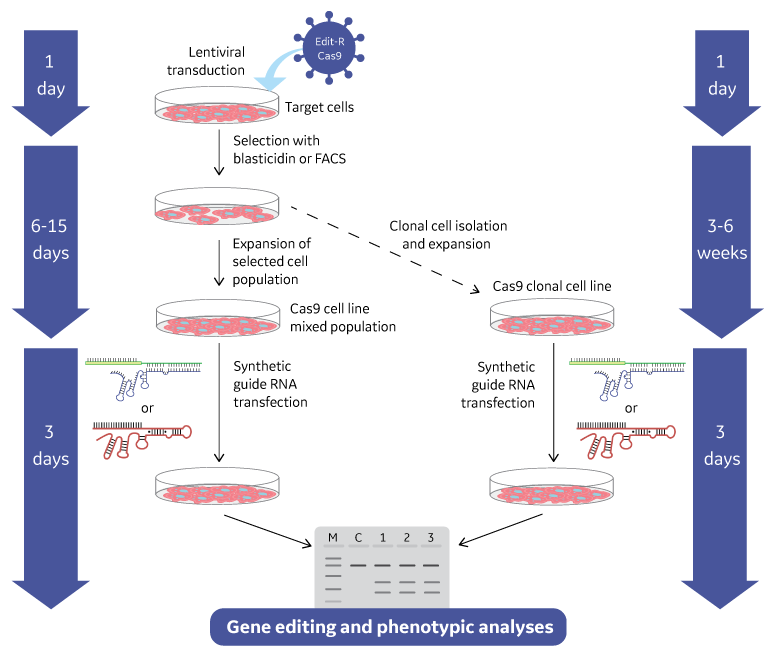
lentiviral Cas9ヌクレアーゼ試薬フォーマットのゲノム編集ワークフロー
lentiviral Cas9ヌクレアーゼとEdit-R Lentiviral sgRNAを使用した遺伝子ノックアウトのワークフロー
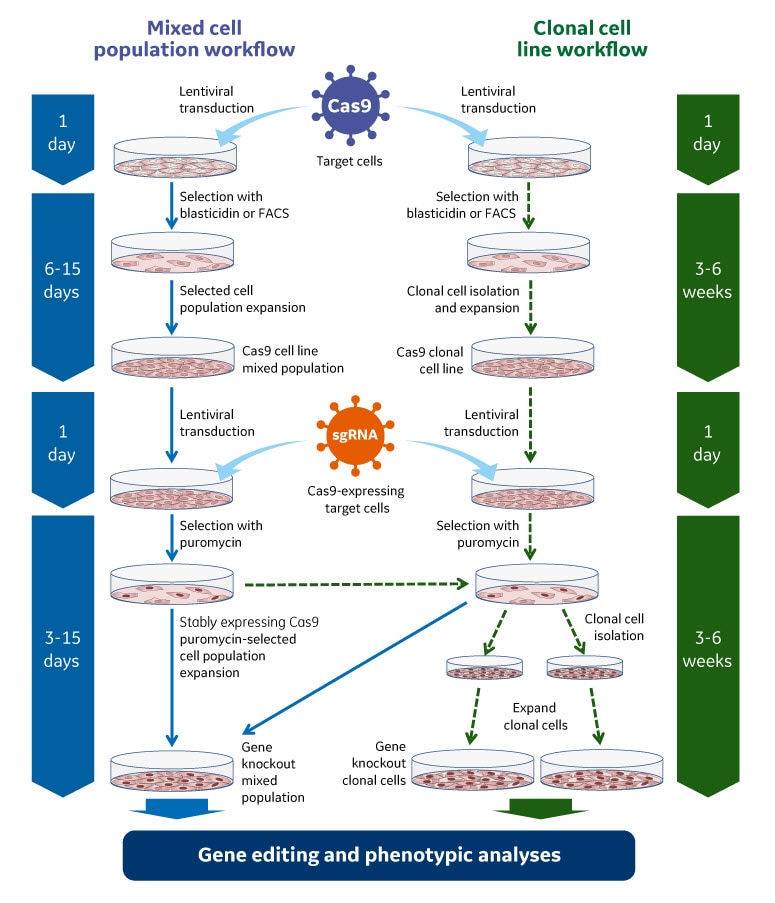
lentiviral Cas9ヌクレアーゼとsgRNAによる遺伝子ノックアウトワークフロー。ゲノム編集は、一般的に遺伝子ノックアウトスクリーニングのために混合細胞集団アプローチ(左側)、または表現型解析のためにより明確な細胞型が必要な場合には単離クローン細胞株アプローチ(右側)を用いて行うことができます。
誘導型lentiviral Cas9ヌクレアーゼとEdit-R Lentiviral sgRNAを使用した遺伝子ノックアウトのワークフロー
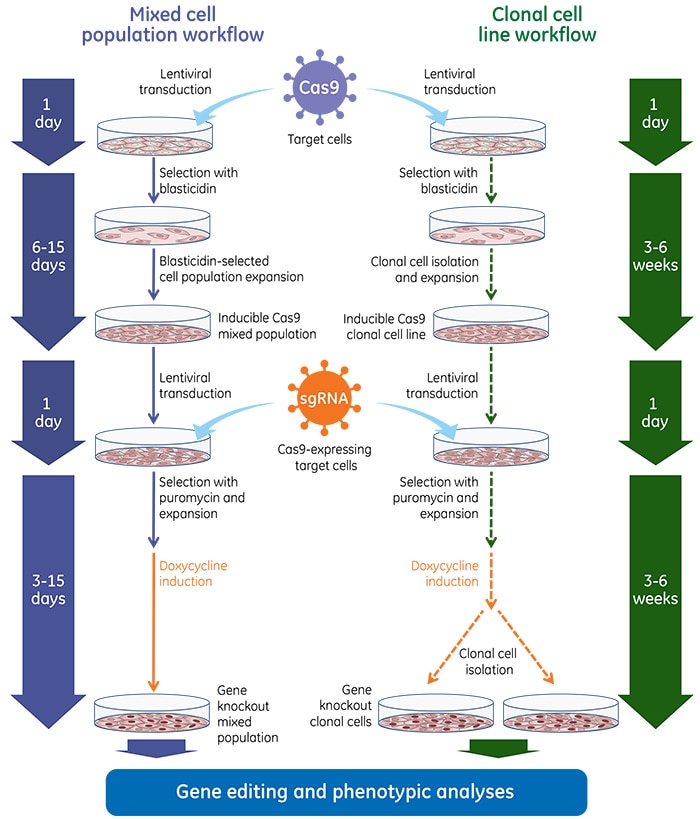
lentiviral Cas9ヌクレアーゼとEdit-R化学合成ガイドRNAを使用した遺伝子ノックアウトワークフロー
プール化lentiviral sgRNAライブラリーを用いたスクリーニングにおける、誘導型lentiviral Cas9ヌクレアーゼを使用した遺伝子ノックアウトワークフロー
lentiviral蛍光Cas9ヌクレアーゼデータ
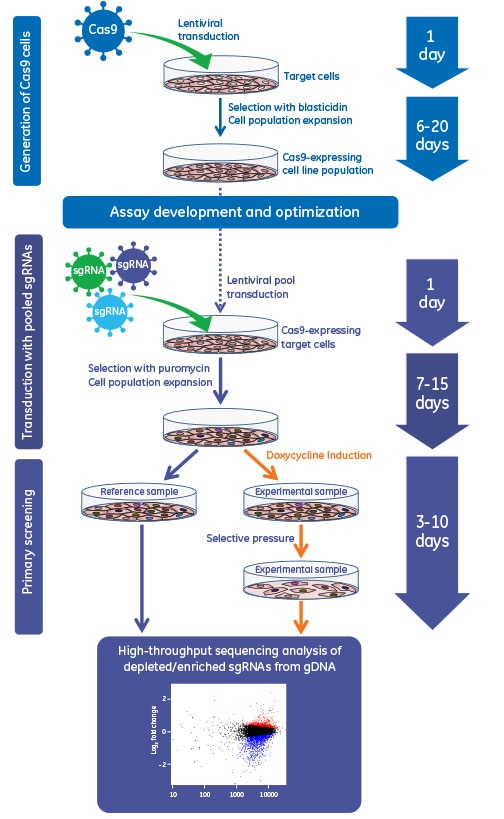
蛍光lentiviral Cas9ヌクレアーゼは、高発現細胞の濃縮とCRISPR-Cas9ゲノム編集効率の向上を可能にする
U2OS 細胞に、Edit-R lentiviral hCMV mKate2-Cas9ヌクレアーゼ粒子(カタログ番号:VCAS11869))を低感染多重度(MOI 0.3)で形質導入し、形質導入された細胞にCas9が1つだけ組み込まれるようにしました。細胞を増殖させ、その後FACSにより細胞集団をmKate2蛍光negative、low、medium、highに分けてソーティングしました。これらの亜集団を増殖し、96ウェルプレートに10,000細胞/ウェルで播種しました。1日後、DharmaFECT 1トランスフェクション試薬(0.3 µL/ウェル、カタログ番号:T-2001-01)を用いて、tracrRNA(25 nM、カタログ番号:U-002005-xx)、および Edit-R PPIB Synthetic crRNA Control(カタログ番号:U-007501-xx)または Edit-R MYC Predesign crRNA(カタログ番号:CM- 003282-01)を、細胞にトランスフェクトしました。 72時間後、In Cell Analyzer 2200(GE Healthcare; A)を使用して、細胞のmKate2の蛍光を画像化し、DNAミスマッチ検出アッセイのために収集して、ゲノム編集を評価しました (B)。mKate2の高発現は、PPIBおよびMYCを標的とするcrRNAのゲノム編集効率の高さと関連している可能性を示唆しています。
lentiviral誘導性Cas9ヌクレアーゼデータ
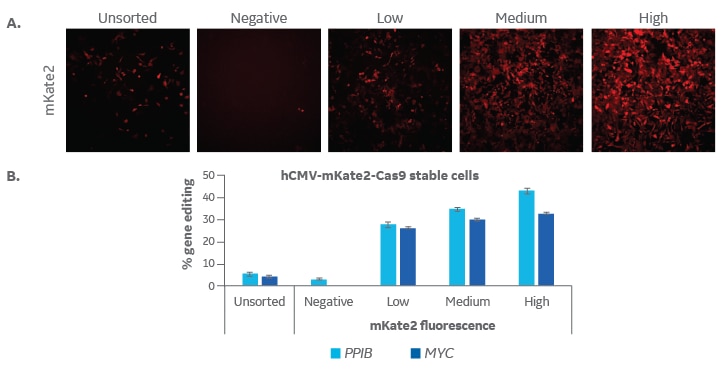
ドキシサイクリンで 7 日間誘導した後の誘導性 Cas9 ベクターのゲノム編集活性
構成型(CAG-Cas9)または誘導型(TRE3G-Cas9)Cas9発現レンチウイルス粒子を MOI 0.3で細胞に形質導入し、10 µg/mLのブラストサイジンを使用してテトラサイクリンを含まない培地で10日間選択しました。次に、Cas9安定細胞にDNMT3BまたPPIB-sgRNAレンチウイルス粒子をMOI 0.3で形質導入しました。テトラサイクリンを含まない培地で、細胞を2 µg /mLのピューロマイシンで2日間選択培養し、2つの集団(DOX-:非誘導、DOX+:誘導)に分割してドキシサイクリンでさらに 7 日間培養しました。細胞を溶解し、T7EIを用いたDNAミスマッチ検出アッセイを用いてIndelについて分析しました。
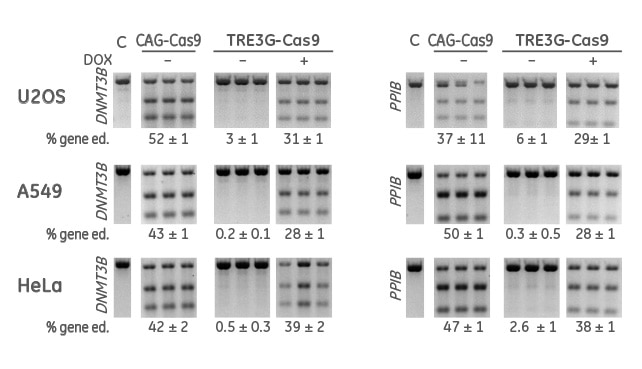
誘導型Cas9ヌクレアーゼベクターは、ドキシサイクリン誘導なしで21日後に最小限のリークを示す
構成型(CAG-Cas9)または誘導型(TRE3G-Cas9)Cas9発現レンチウイルス粒子を MOI 0.3で細胞に形質導入し、10 µg/mLのブラストサイジンを使用してテトラサイクリンを含まない培地で10日間選択しました。次に、Cas9安定細胞にDNMT3または PPIB-sgRNAレンチウイルス粒子をMOI 0.3で形質導入しました。テトラサイクリンを含まない培地で細胞を2 µg/mLピューロマイシンで選択し、21日間維持しました。細胞を溶解し、T7EIを用いたDNAミスマッチ検出アッセイを用いてIndelについて分析しました。
誘導型U2OS-Cas9細胞におけるドキシサイクリンの用量反応
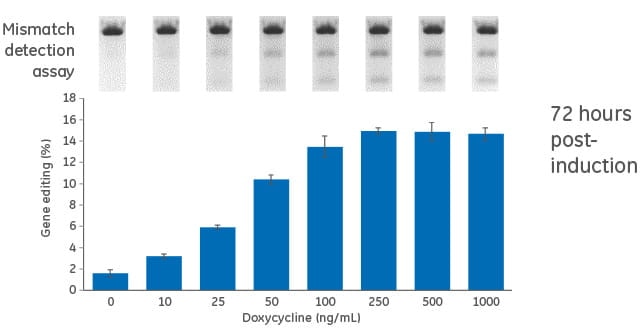
細胞に誘導型(TRE3G-Cas9)Cas9発現レンチウイルス粒子をMOI 0.3で形質導入し、テトラサイクリンを含まない培地中で10 µg/mLのブラストサイジンを使用して 10日間選択しました。次に、Cas9安定細胞にMOI 0.3でPPIB-sgRNAレンチウイルス粒子を形質導入しました。細胞を2 µg/mLのピューロマイシン・テトラサイクリンを含まない培地で4日間選択し、トリプシンで懸濁し、96ウェルプレートに様々な濃度のドキシサイクリン(0~1000 ng/mL)を含有した培地に播種しました。細胞を 72時間インキュベートした後、溶解し、T7EIによるDNAミスマッチ検出アッセイを用いてIndel形成について分析しました。
上段:PPIBターゲットアンプリコンのT7EIを使用したDNAミスマッチ検出アッセイの代表的なゲル画像
下段:独立に処理された3つのウェルからのゲノム編集の推定パーセンテージの平均±SD
lentiviral Cas9ヌクレアーゼ試薬データ
異なるプロモーターによる Cas9 の発現差により、さまざまなレベルのゲノム編集が誘導される
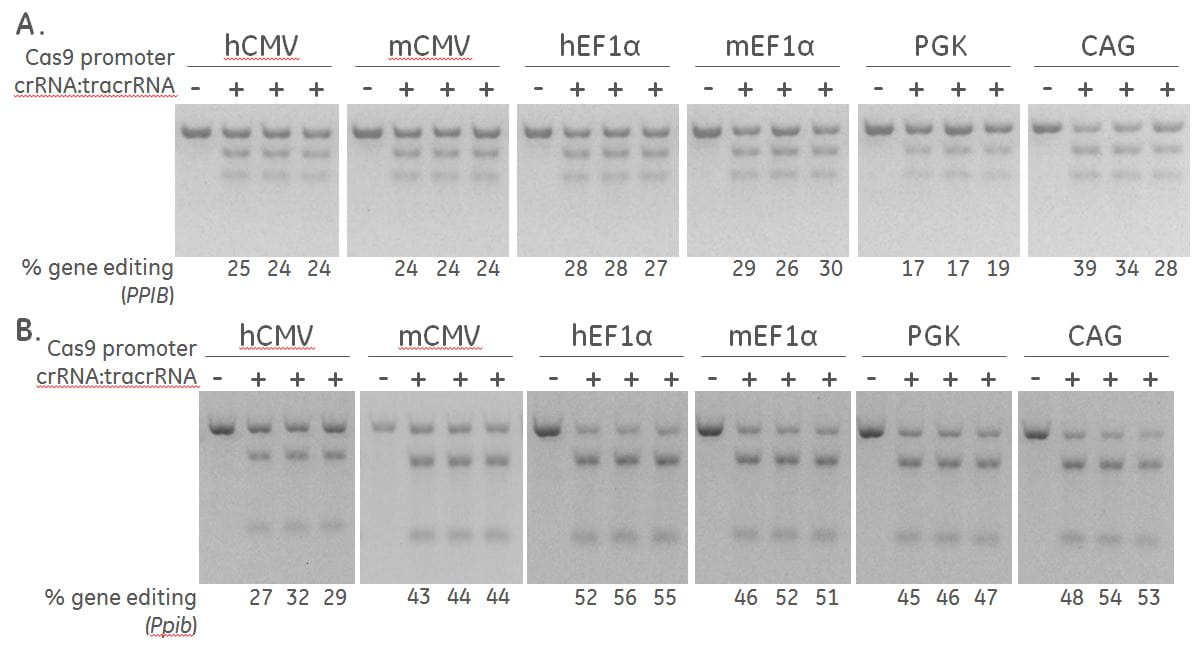
(A)ヒト組換えU2OSユビキチン-EGFPプロテアソーム細胞株(Ubi[G76V]-EGFP)と(B)マウス線維芽細胞(NIH/3T3)に、図示のプロモーターで駆動するCas9とブラストサイジン耐性遺伝子を含むレンチウイルス粒子を安定的に形質導入しました。Cas9-blastRが安定に組み込まれた細胞集団は、トランスフェクションの前に10日間以上ブラストサイジンで選択しました。DharmaFECT 1およびDharmaFECT 3トランスフェクション試薬をそれぞれ使用して、ヒトPPIB/マウスPpibを標的とする50 nM化学合成crRNA:tracrRNAを細胞にトランスフェクトしました。72 時間後、ゲノム編集の相対頻度は、トランスフェクトされた細胞から抽出されたゲノムDNAでT7EIを使用するDNAミスマッチ検出アッセイに基づいて計算しました。
- D. Bhaya, et al. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation. Annu. Rev. Genet.45, 273-297 (2011).
- M. Jinek, et al. A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science337, 816-821 (2012).
- E. Deltcheva, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor Nuclease III. Nature471, 602-607 (2011).
- P. Mali, et al. RNA-guided human genome engineering via Cas9. Science339, 823-826 (2013).
- Y. Fu, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol.31, 822-826 (2013).
- P.D. Hsu, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol.31, 827-832 (2013).
- T. Wang et al. Genetic screens in human cells using the CRISPR-Cas9 system. Science343, 80-84 (2014).
- D.Y. Guschin, et al. A rapid and general assay for monitoring endogenous gene modification. Methods Mol. Biol.649, 247-256 (2010).
- L.Cong, et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science339, 819-823 (2013).
- J.C. Kappes, X. Wu and J.K. Wakefield. Production of trans-lentiviral vector with predictable safety. Methods Mol. Med.76, 449-465 (2003).
- R.H. Kutner, X.-Y. Zhang and J. Reiser. Production, concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nat. Protoc.4, 495-505 (2009).
LentiBOOST Lentivirus Transduction Enhancer is a uniquely formulated transduction reagent that can be used with or without lentivirus spinfection in order to increase successful viral transduction events while preserving cell viability. Especially critical for preserving precious primary cells from patient cohorts, or, for engineering complex animal models; improving transduction efficiency can save time and costs by increasing the success of each editing/transduction step, or, even avoid the loss of irreplaceable samples. Additionally, LentiBOOST technology is already used in the manufacturing of a number of clinical stage therapies providing the opportunity to demonstrate improved workflow applicability to the clinic.
LentiBOOST can be purchased through the Dharmacon Reagents catalog.
To learn more about LentiBOOST technology visit the Revvity LentiBOOST webpage.
Supporting Data
Improved CD8+ T-cell SMARTvector™ shRNA lentiviral system transduction using LentiBOOST™ Lentivirus Transduction Enhancer
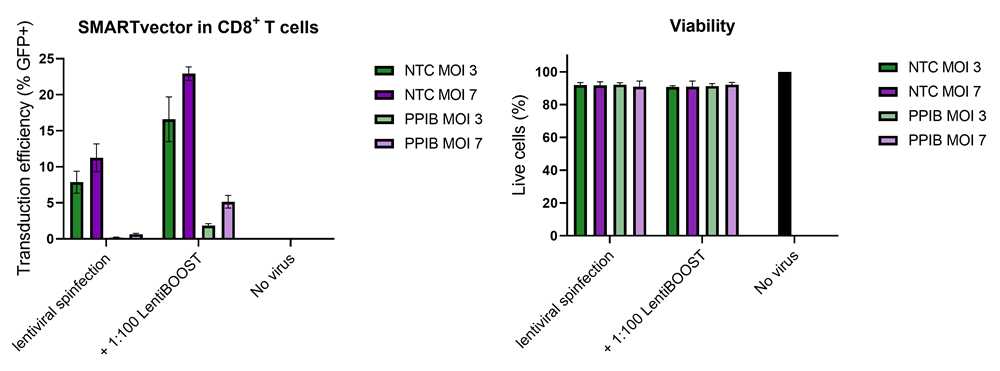
100,000 primary human CD8+ T cells were transduced with either 30,000 (MOI 3, green) or 70,000 (MOI 7, purple) TUs of SMARTVector™ mCMV tGFP Lentiviral Control Particles targeting either NTC or PPIB along with 1:100 LentiBOOST transduction enhancer. Cells were centrifuged at 800 x g for one hour at 32 °C followed by a four hour incubation prior to removal of lentiviral particles and transduction enhancer. Transduction efficiency (%GFP+ out of live cells) and viability were determined 5 days post-transduction by flow cytometry. The addition of LentiBOOST technology markedly improved transduction efficiencies without significantly impacting cell viability.
Improved CD4+ and CD8+ T-cell Edit-R™ All-in-one sgRNA/Cas9 lentiviral system transduction using LentiBOOST™ Lentivirus Transduction Enhancer
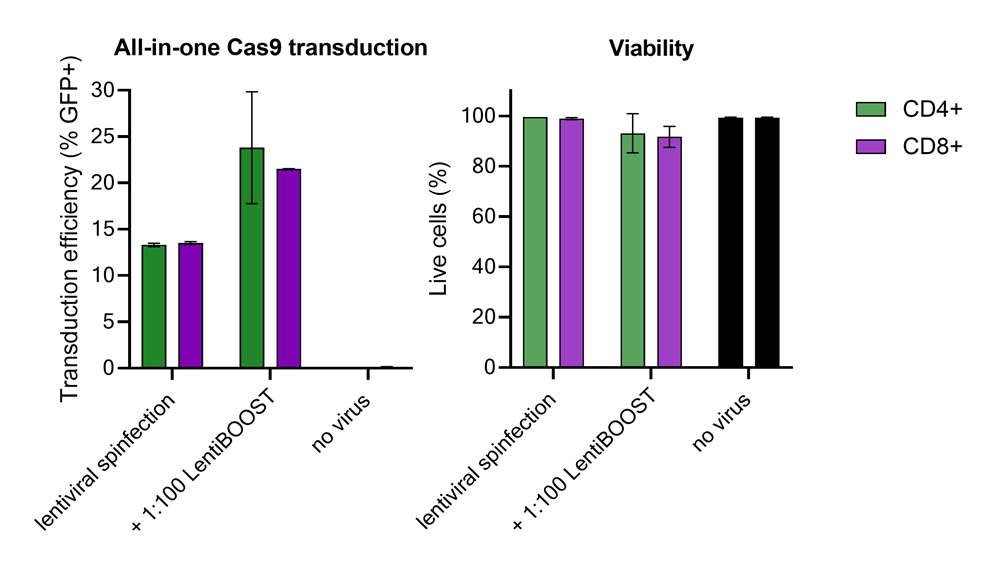
100,000 primary human CD4+ and CD8+ T cells from two donors were transduced with 250,000 TUs of Edit-R GFP Delivery controls mCMV along with 1:100 LentiBOOST transduction enhancer. Cells were centrifuged at 800 x g for one hour at 32 °C followed by an overnight incubation prior to removal of lentiviral particles and transduction enhancer. Transduction efficiency and viability were determined 72 hours post-transduction by flow cytometry. The addition of LentiBOOST technology markedly improved transduction efficiencies without significantly impacting cell viability.
Improved transduction of human induced pluripotent stem cells (hiPSCs) with the Strict-R™ Inducible CRISPRa lentiviral system transduction using LentiBOOST™ Lentivirus Transduction Enhancer

10,000 WTC hiPS cells were transduced with either 20,000 (MOI 2, green) or 40,000 (MOI 4, purple) TUs of Strict-R™ Inducible EGFP dCas9-VPR Lentiviral Particles along with 1:100 LentiBOOST transduction enhancer. Cells were centrifuged at 800 x g for one hour at 32 °C followed by an overnight incubation prior to removal of lentiviral particles and transduction enhancer. Transduction efficiency and viability were determined 72 hours post-transduction by flow cytometry. The addition of LentiBOOST markedly improved transduction efficiencies without significantly impacting cell viability.
Application notes
Posters
Protocols
Safety data sheets
Selection guides
Technical manuals
-
CRISPR-Cas9 genome engineering with inducible lentiviral Cas9 nuclease and Edit-R guide RNAs - Technical Manual
-
CRISPR-Cas9 genome engineering with lentiviral Cas9 particles and Edit-R synthetic guide RNA - Technical Manual
-
Edit-R CRISPR-Cas9 Gene Engineering with Lentiviral Cas9 and sgRNA - Technical Manual
Related Products
LentiBOOST transduction enhancer can increase successful viral transduction in challenging to transduce cells, or, complex cellular engineering work; while preserving cell viability and minimizing the amount of viral particles required for your experiment. LentiBOOST technology is actively used in the production of clinical stage lentivirally delivered therapies, including some approved therapies, providing a direct path to therapeutic applicability for your research studies. Tested with Dharmacon Lentiviral reagents.
Unlock precise, tunable gene knockout with the DharmaconTM Strict-RTM inducible Cas9 lentiviral system. This advanced platform enables stringent, control of gene disruption with exceptional specificity and minimal off-target activity. By integrating Tet-On 3G transcriptional regulation with FKBP12-derived degron–based destabilization, the system allows inducible expression and degradation of Cas9 in response to small-molecule regulators, providing tight temporal control over genome editing activity for dynamic and time-resolved functional studies.