Edit-R デザイン済みSynthetic CRISPR RNA(化学合成crRNA)
効率的な遺伝子ノックアウトと高い特異性を提供する化学合成crRNA
- 目的の標的遺伝子の編集(DNA切断)が保証されています。
- タンパク質の機能的ノックアウトの可能性を最大化し、オフターゲット編集を最小化するようにデザインされています。
- トランスフェクションに対応した化学合成RNAは、クローニングとin vitro転写ステップを必要としません。
- ヌクレアーゼ耐性の向上のために、化学修飾が導入されています。
- New! Edit-R human sgRNA designs have been updated to the latest RefSeq as of August 22, 2025.
Reminder:すべての化学合成crRNAは、化学合成tracrRNA(synthetic tracrRNA)と組み合わせて用い、Cas9編集を実現する完全な「ガイドRNA複合体」を形成する必要があります。

Edit-R デザイン済みSynthetic CRISPR RNA(化学合成crRNA)
1Start Here
2Choose
化学合成crRNAは効率的な遺伝子ノックアウトと高い特異性を提供します。

Edit-R crRNAは化学合成されたRNAであり、ゲノムDNAの標的部位を認識する20ヌクレオチドで構成され、その後に必要なS. pyogenesリピート配列が続きます。ゲノム編集に必要な〜100merのガイドRNA構造全体を完成させるには、crRNAとtracrRNAの複合化が必要です。したがって、化学合成crRNAをご使用の際には、必ずtracrRNAも一緒にご注文ください。
CRISPR-Cas9は遺伝子機能を調べるための非常に効果的なツールですが、すべてのガイドRNAがタンパク質の機能的なノックアウトを実現するのに十分効果的であるとは限りません。この問題に対処するために、Dharmaconは、挿入または欠失を起こすだけでなく、機能的な遺伝子ノックアウトを生じる可能性が最も高いガイドRNAを選択するように検証されたアルゴリズムを開発しました。
New! Edit-R human sgRNA designs have been updated to the latest RefSeq in 2025 providing the most specific and genomically relevant guides for producing efficient protein knockout. This allows the Edit-R algorithm to target the latest genome annotations more accurately and efficiently providing you with the best solution for your research needs. Please reach out to Scientific Support if you have any questions.
すべてのガイドRNAデザインは、各遺伝子についてアルゴリズムで選択した最上位のものです。機能性と特異性の定性的なランク付けにより、特定のアプリケーションに合わせて最適なヒトガイドRNAを選択できます。機能性スコアは、このガイドがどの程度機能的ノックアウトをもたらす可能性があるかを予測したものです。特異性スコアは、潜在的なオフターゲット部位での切断活性の予測リスクに基づいています。詳細については、Edit-RガイドRNAのアルゴリズムページをご覧ください。
化学修飾がすべてのデザイン済みcrRNAに導入されており、ヌクレアーゼによる分解が減少するため、DNA-freeのCas9との同時導入を行うアプリケーションにおいて、ゲノム編集性能が向上しています。詳細については、featured articleをご覧ください。
化学合成crRNAを使用したEdit-R CRISPR-Cas9ゲノム編集実験に必要なコンポーネント:
- Cas9ヌクレアーゼ(タンパク質、mRNA、発現用、発現用レンチウイルスベクター)
- 目的の遺伝子を標的としてデザインされEdit-R化学合成CRISPR RNA (crRNA)
- Edit-R trans-activating CRISPR RNA (tracrRNA)
Edit-RガイドRNA保証
すべてのデザイン済みEdit-RガイドRNAは、Edit-Rテクニカルマニュアルの記載に従い細胞導入した場合に、標的領域で編集(DNA切断)ができることを保証します。
Edit-RガイドRNA保証は、野生型S. pyogenes由来Cas9ヌクレアーゼ(タンパク質、mRNA、発現用プラスミド、または発現用レンチウイルスベクター)とともに使用した場合に適用されます。更にEdit-R 化学合成crRNAは、Edit-R tracrRNAとともに使用した場合に適用されます。
T7EIまたはSurveyorミスマッチ検出アッセイを使用して、試薬で処理された細胞集団の編集(DNA切断)の分析結果を提示する必要があります。同時並行で適切に実施されたEdit-R ポジティブコントロールが編集(DNA切断)に成功する一方で、Edit-Rデザイン済みガイドRNAによる編集(DNA切断)が成功しない場合、同じフォーマットで同じ容量の、異なるEdit-Rデザイン済みガイドRNAの交換製品が、1回限り無償で提供されます。
交換製品の提供は、テクニカルサポートチームとの話し合いでのみ承認されます。
DNAレベルでの編集(切断)の成功は、常に機能的な遺伝子ノックアウトにつながるとは限りません。複数のガイドRNAをテストして、標的遺伝子のノックアウトに最も効果的なガイドRNAを決定することをお薦めします。
この保証は付随する実験費用には適用されず、CRISPR Design Toolを介して注文されたガイドRNAには適用されず、また本保証により交換したガイドRNAには適用されません。
Gene knockout workflow using Cas9 nuclease protein and synthetic sgRNA or crRNA:tracrRNA
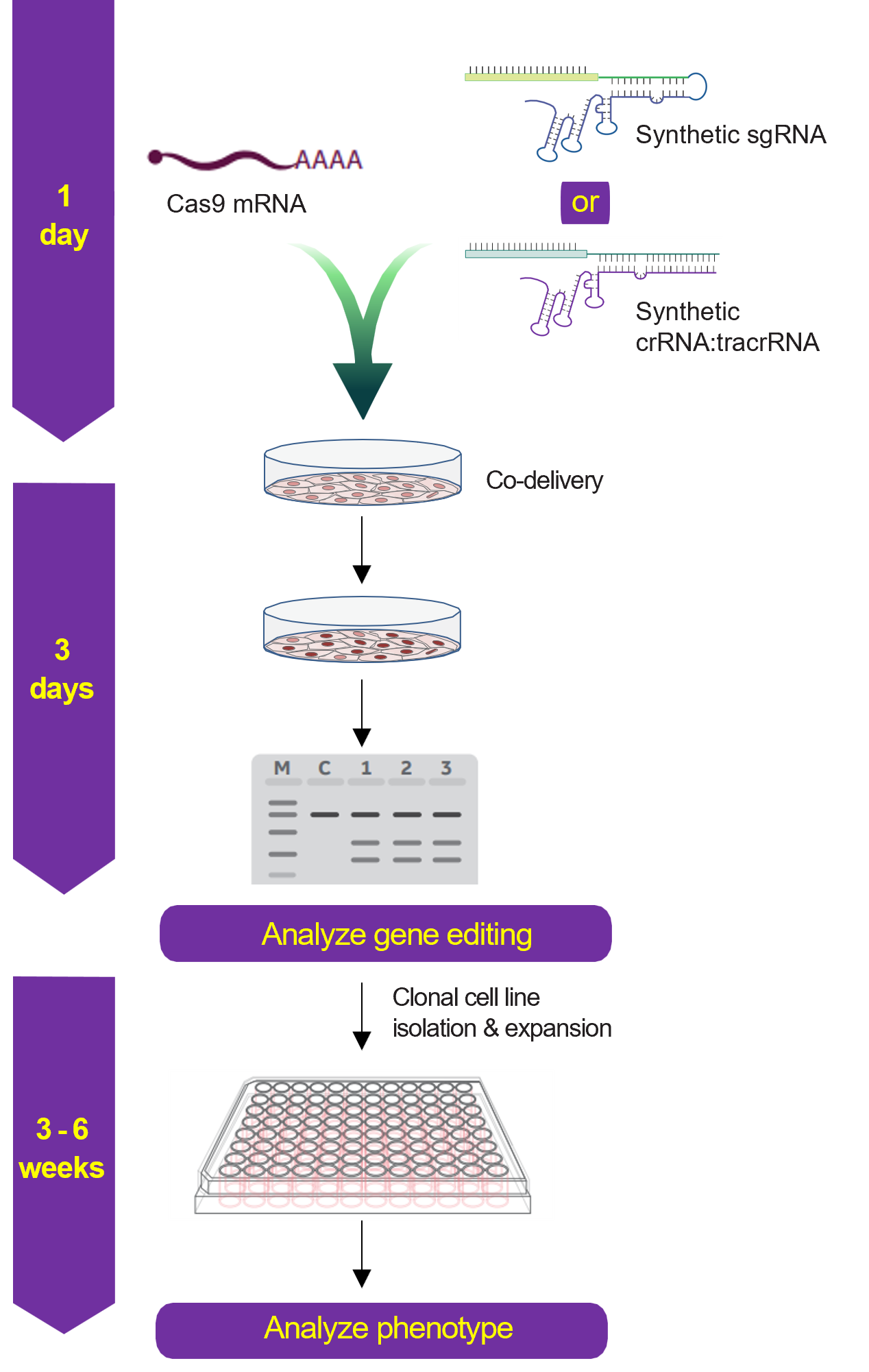
Gene knockout workflow using Cas9 nuclease protein and synthetic sgRNA or crRNA:tracrRNA

Gene knockout workflow using lentiviral Cas9 expression particles and synthetic sgRNA or crRNA:tracrRNA

Edit-R 化学合成crRNAコントロール
-
ポジティブコントロールと検出プライマー
十分に特徴付けられた遺伝子を標的とする種特異的な化学合成crRNAおよびミスマッチ検出アッセイプライマーを使用して、ゲノム編集効率を最大化する実験条件の検討、および最適化の確認のために使用します。
-
Non-targetingコントロール
標的遺伝子特異的crRNAの非存在下でCRISPR-Cas9コンポーネントに対するベースラインの細胞応答を評価するために使用します。
-
Cutting controls
Cutting control (safe harbor) crRNAs recommended for determination of baseline cellular responses in CRISPR-Cas9 experiments.
Synthetic guide RNA
-
Edit-R tracrRNA
Edit-R trans-activating CRISPR RNA (tracrRNA) is synthetic, HPLC-purified, long RNA required for use with Edit-R crRNA to form the complex that programs Cas9 nuclease. It is modified for nuclease resistance and can be used with modified or unmodified Edit-R crRNA.
-
Edit-R synthetic positive crRNA controls and detection primers
Species-specific crRNAs targeting well-characterized genes, as well as mismatch detection assay primers, to determine the effectiveness of your gene editing conditions for maximal efficiency.
-
Edit-R synthetic crRNA non-targeting controls
Non-targeting controls to evaluate cellular responses to CRISPR-Cas9 components in the absence of gene target-specific crRNA.
-
CRISPR design tool
Place a custom guide RNA order, or design and order your own synthetic sgRNA, crRNA, or lentiviral sgRNA with our easy-to-use interface.
-
Cherry-pick libraries
Have a favorite gene list? Customize and order plates of predesigned crRNA for knockout studies in your targets of interest
Cas9 nuclease
-
Cas9 nuclease protein NLS
Purified Cas9 protein ready to use for DNA-free nuclease expression.
-
Cas9 nuclease mRNA
Purified Cas9 mRNA for transient Cas9 Nuclease expression. Fluorescent options available for sorting, enrichment and visualization of delivery.
-
Lentiviral Cas9 nuclease expression reagents
Purified lentiviral particles or plasmid DNA for generation of stable Cas9 nuclease-expressing cell populations. Constitutive or inducible promoter options are available.
組み換えU2OS細胞株で安定的に発現したCas9、およびEdit-R 化学合成crRNA:tracrRNAを用いた細胞ベースのアッセイで観察されたVCPタンパク質の機能的ノックアウト
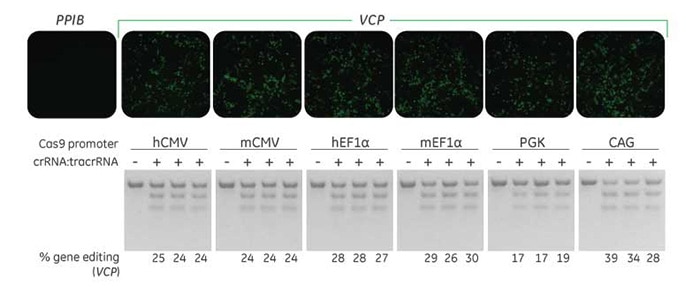
組み換えU2OSユビキチン-EGFPプロテアソーム細胞株(Ubi [G76V] -EGFP)に、特定の(図に示した)プロモーターの制御下でCas9を発現するレンチウイルス粒子を形質導入しました。Cas9が安定して組み込まれた細胞集団を、トランスフェクションの最低10日前からブラストサイジン処理で選択しました。DharmaFECT 3 Transfection Reagentを使用して、プロテアソーム機能に必要な遺伝子であるVCPを標的とするEdit-R 化学合成 crRNA:tracrRNA複合体を細胞に50 nMにてトランスフェクトしました。72時間後、トランスフェクトされた細胞のEGFP +細胞を調べ(上パネル)、T7エンドヌクレアーゼIを用いたDNAミスマッチ検出アッセイに基づいて、ゲノム編集の相対頻度を計算しました(下パネル)。
化学合成sgRNAおよびcrRNA:tracrRNAのノックアウトパフォーマンス
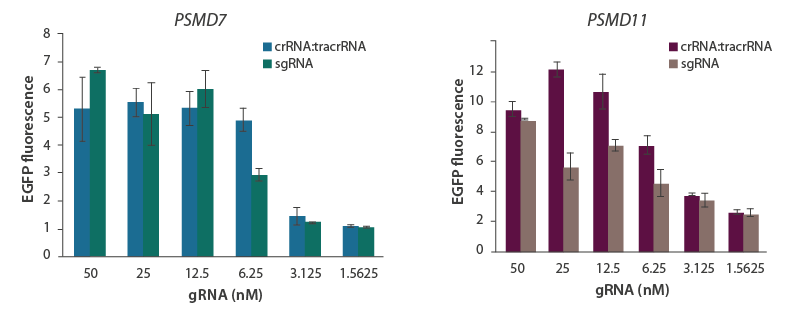
CAGプロモーター制御下でCas9を安定的に発現するU2OSユビキチン-EGFP細胞株に、プロテアソーム成分PSMD7(A)またはPSMD11(B)を標的とするガイドRNA(crRNA:tracrRNAまたはsgRNA)の濃度を変えてトランスフェクトしました。トランスフェクトされた細胞は、EGFPの蓄積/強度により機能的遺伝子ノックアウトについて評価しました。
化学合成crRNA:tracrRNAは、すべてのCas9 Nucleaseフォーマットと互換性がある
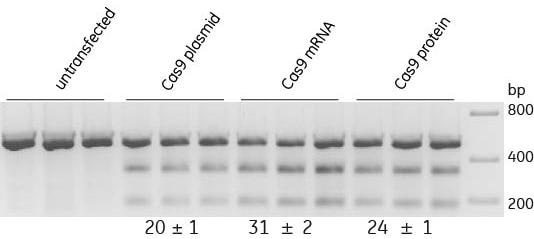
U2OS細胞をトランスフェクションの1日前に10,000細胞/ウェルで播種しました。DharmaFECT Duoトランスフェクション試薬(0.4 uL/ウェル)を使用して、Edit-R Cas9 Nucleaseプラスミド(200 ng)、Edit-R Cas9 Nuclease mRNA(200 ng)またはCas9ヌクレアーゼタンパク質(25 nM)のいずれかを、PPIBをターゲットとするcrRNA:tracrRNA(25 nM)と共にトランスフェクトしました(生物学的トリプリケート)。
Edit-R crRNA:tracrRNAのトランスフェクションによるCas9発現NIH/3T3細胞株での効率的なゲノム編集
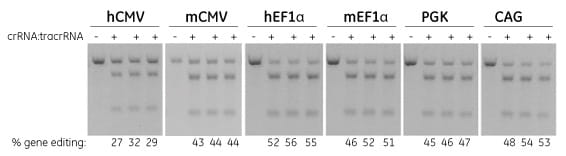
NIH/3T3細胞に、特定の(図に示した)プロモーターによって駆動されるCas9およびブラストサイジン耐性遺伝子を含むレンチウイルス粒子で安定的に形質導入しました。トランスフェクションの10日前からブラストサイジン選抜を行い、Cas9の安定的に導入された細胞集団を選択しました。DharmaFECT 1トランスフェクション試薬を使用して、PPIBをターゲットとする50 nM Edit-R 化学合成crRNA:tracrRNAを細胞にトランスフェクトしました。72時間後、T7エンドヌクレアーゼIを用いたDNAミスマッチ検出アッセイに基づいて、ゲノム編集の相対頻度を計算しました。
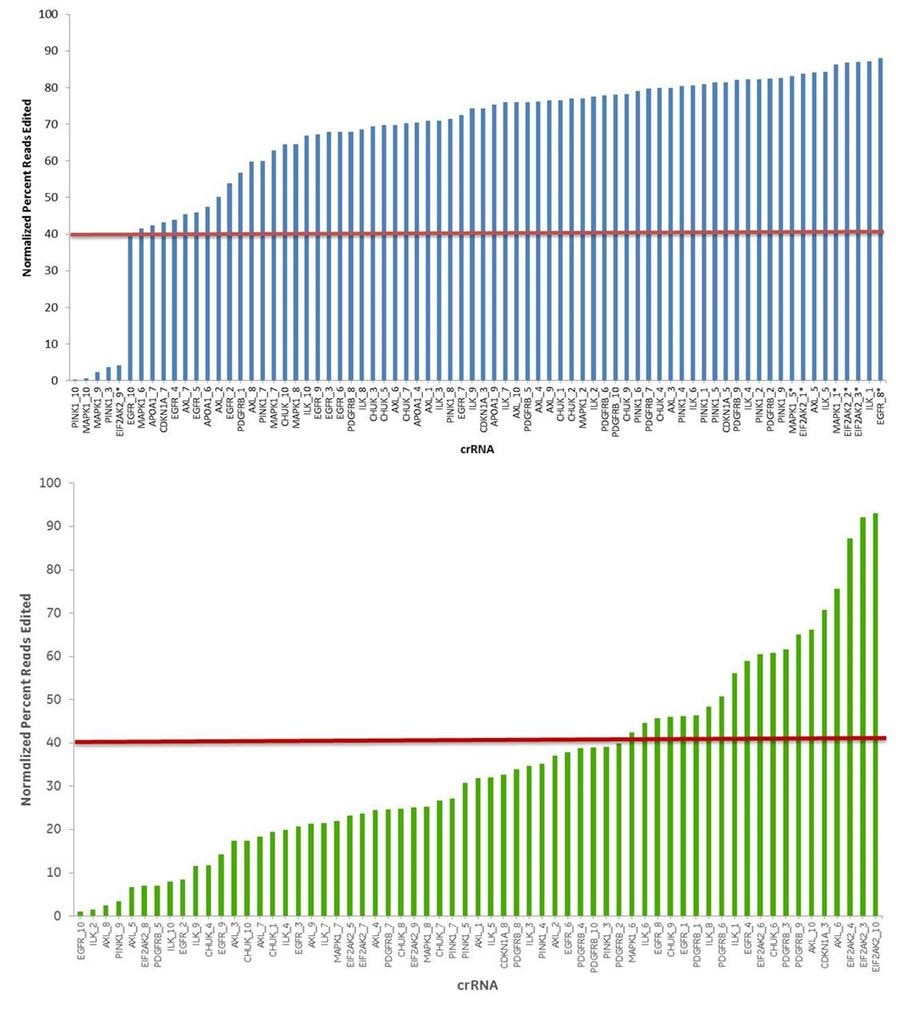
デザインアルゴリズムのパフォーマンス
Edit-R アルゴリズムのスコアが高いcrRNAは、スコアの低いデザインよりも切断効率が高い
10個の遺伝子について、機能スコアが高い10個のcrRNA(青いバー)と、同じ遺伝子について、機能スコアが低い10個のcrRNA(黄緑色のバー)を、次世代シーケンサーによってゲノム編集効率を見るためにテストしました。高スコアのcrRNAの93%について40%以上の編集(挿入または欠失)が確認できました。一方、低スコアのcrRNAの32%について40%以上の編集(挿入または欠失)が確認できました。0.25 µL/ウェルのDharmaFECT 1を使用して、Cas9-HEK293T細胞株に50 nM crRNA:tracrRNAをトランスフェクトしました。トランスフェクションの72時間後、細胞を溶解し、各crRNA部位にまたがるNexteraトランスポゾン適合アンプリコンを、処理したサンプルごとに生成しました。トランスフェクトされていないサンプルからのマッチしたコントロールアンプリコンも同時に生成しました。Nextera 96-well index kitを使用してサンプルのインデックスを作成し、MiSeqでのシーケンス用にプールしました。NGS品質フィルタリング基準に合格したリードは、参照ファイル(Bowtie2 v2.1.0)に配列されました。完全なリードの割合が計算され、対照であるトランスフェクトされていないサンプル(Samtools v0.1.12a)に対して正規化されました。 データは編集された割合を正規化して表示しています。
Edit-R アルゴリズムにおいて機能スコアが高いcrRNAのデザインは、低スコアよりもApoONEアッセイでより強い表現型を示す
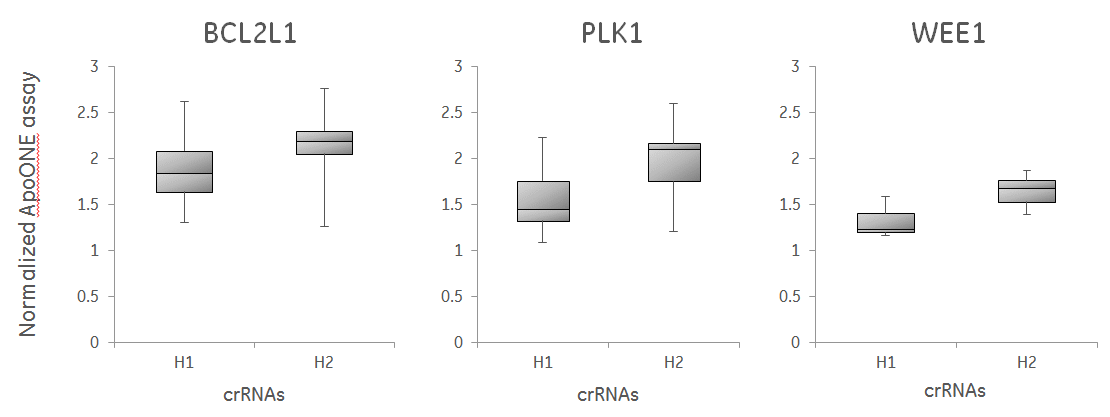
CAGプロモーターの下流にCas9が組み込まれたU2OS-プロテアソーム細胞を96ウェルプレートに1ウェルあたり10,000細胞で播種しました。プレーティングの24時間後、0.2 µg/ウェルのDharmaFECT 4を使用して、細胞に25 nM crRNA:tracrRNAをトランスフェクトしました。 ApoONEホモジニアスアッセイ(Promega)を使用して、トランスフェクションの48時間後にアポトーシスについて細胞を分析しました。ApoONEアッセイでBCL2L1、PLK1、またはWEE1をターゲットとするcrRNAの機能をボックスプロットで表しています。crRNAを機能スコアに基づいて下半分(H1)と上半分(H2)に分割して、ボックスプロットを作成しました。中央値、下位四分位と上位四分位間のデータの分布、および最小値と最大値は、スコアの高いcrRNAは機能が向上していることを示しています。
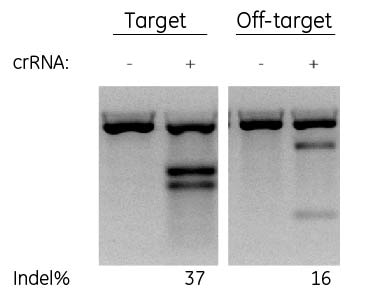
ギャップとミスマッチのあるデザインは、オフターゲット切断を引き起こす可能性がある
目的の標的部位を持つcrRNA(GGTCATCTGGGAGAAAAGCG)と、1つのギャップと1つのミスマッチを含みオフターゲットなcrRNA(GGT-ATCTGGGAGAAAAGCA)のT7EIミスマッチ分析です。オフターゲットなcrRNAについて、Edit-Rアライメントツールではギャップとミスマッチが特定されましたが、他のオンラインツールでは特定されませんでした。一般的に使用されるWebベースのcrRNA特異性ツールの多くは、アライメントを実行する際のギャップを完全には考慮していません。
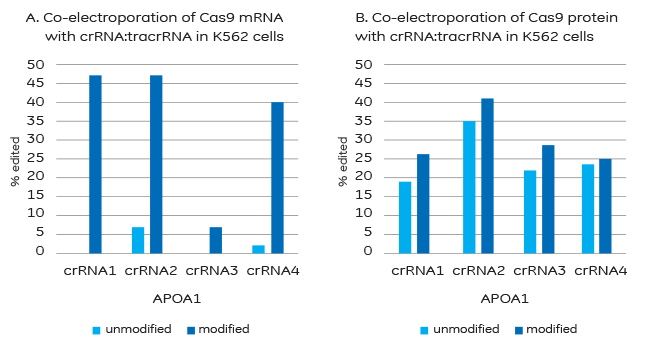
crRNA修飾デザイン
2x MS修飾または非修飾crRNA:tracrRNAによるゲノム編集の効率
A.ヌクレアーゼによる分解をブロックするための化学修飾は、Cas9 mRNAとcrRNA:tracrRNAの同時エレクトロポレーションを成功させるために必要です。
B. Cas9タンパク質とcrRNA:tracrRNAの同時エレクトロポレーションでは、安定化修飾は必要ありませんが、追加することで結果が改善される場合があります。
Edit-R 化学合成ガイドRNAは、in vitroで転写されたガイドRNAと比較して、本質的に自然免疫応答や毒性を引き起こさない
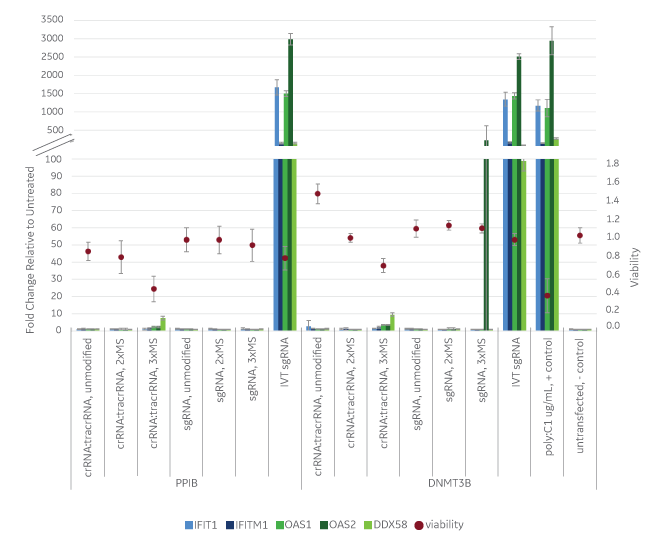
HEK293T Cas9ヌクレアーゼ発現細胞株に、PPIBおよびDNMT3B遺伝子を標的とする異なる化学合成ガイドRNAフォーマット(非修飾crRNA:tracrRNA、5’crRNAと3’ tracrRNAを2xMSで修飾したcrRNA:tracrRNA、crRNAとtracrRNA ともに5’および3’を3xMSで修飾したcrRNA:tracrRNA、そして非修飾化学合成sgRNA、5’および3’を2xMSまたは3xMSで修飾した化学合成sgRNA、およびin vitro転写sgRNA)をトランスフェクションしました。72時間後に、レサズリン還元アッセイ(赤い点)で生存率を評価し、5つの免疫応答遺伝子のレベルをRT-qPCRによって定量化しました。
*MS: 2’-O-メチルヌクレオチドとホスホロチオエート骨格結合
Edit-R 化学合成crRNAおよびtracrRNAのヌクレアーゼ耐性を改善するための修飾の位置と構造
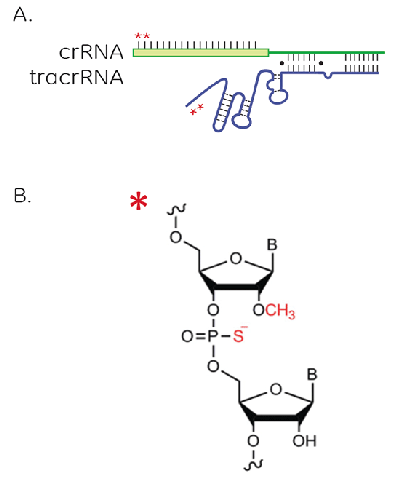
A. Edit-R 化学合成crRNAおよびtracrRNAでのヌクレアーゼ耐性の修飾の位置。 Edit-R 化学合成ガイドRNAは、crRNA(緑色鎖)の5’末端の2つのヌクレオチドとtracrRNA(青い鎖)の3’末端の2つのヌクレオチドに2ʹ-O-methyl (2ʹ-OMe)および骨格ホスホロチオエート結合(PS)を含みます。
B. 2ʹ-OMeおよびPS骨格のホスホロチオエート結合の構造
crRNAおよびtracrRNA必要量
この表は、推奨されるcrRNA:tracrRNA作業濃度(25 nM:25nM)を用いて、さまざまなプレート/ウェル形式でリピッドトランスフェクション法を実施できる概算の実験数を示しています。計算では、ピペッティングエラーは考慮されていません。
| crRNA nmol | tracrRNA nmol | 96-well plate 100 µL reaction volume | 24-well plate 500 µL reaction volume | 12-well plate 1000 µL reaction volume | 6-well plate 2500 µL reaction volume |
|---|---|---|---|---|---|
| 2 | 2 | 800 | 160 | 80 | 32 |
| 5 | 5 | 2000 | 400 | 200 | 80 |
| 10 | 10 | 4000 | 800 | 400 | 160 |
| 20 | 20 | 8000 | 1600 | 800 | 320 |
- R. Barrangou, A. Birmingham, et. al. Advances in CRISPR-Cas9 genome engineering: lessons learned from RNA interference. Nucleic Acids Research, 43(7) 3407-3419 (2015)
- M.L. Kelley, Ž. Strezoska, et al. Versatility of chemically synthesized guide RNAs for CRISPR-Cas9 genome editing. J. Biotechnol. 233, 74–83 (2016). doi:10.1016/j.jbiotec.2016.06.011
- Basila, M., M. L. Kelley, et al.. Minimal 2’-O-methyl phosphorothioate linkage modification pattern of synthetic guide RNAs for increased stability and efficient CRISPR-Cas9 gene editing avoiding cellular toxicity. PLoS One. 12, e0188593 (2017). doi: 10.1371/journal.pone.0188593
- Anderson, E.M., A. Haupt, et al. Systematic analysis of CRISPR-Cas9 mismatch tolerance reveals low levels of off-target activity. J. Biotechnol. 211, 56-65 (2015)
Application notes
-
A CRISPR-Cas9 gene engineering workflow: generating functional knockouts using Edit-R Cas9 and synthetic crRNA and tracrRNA - Application Note
-
CRISPR gene editing and transcriptional activation in human iPS cells - Application Note
-
Homology-directed repair with Dharmacon Edit-R CRISPR-Cas9 reagents and single-strand DNA oligos - Application Note
-
Microinjection of zebrafish embryos using Dharmacon Edit-R Cas9 Nuclease mRNA, synthetic crRNA, and tracrRNA for genome engineering - Application Note
-
Optimization of reverse transfection of Edit-R synthetic crRNA and tracrRNA components with DharmaFECT transfection reagent in a Cas9-expressing cell line - Application Note
Posters
-
2'-O-methyl phosphorothioate linkage-modified synthetic guide RNAs for efficient CRISPR-Cas9 genome editing and reduced cellular toxicity - Poster
-
A Synthetic CRISPR-Cas9 System for Homology-Directed Repair - Poster
-
An algorithm for selecting functional and specific CRISPR-Cas9 guide RNAs - Poster
-
An algorithm for selecting highly functional and specific guide RNAs for CRISPR-Cas9 gene knockout - Poster
-
Chemical modifications of synthetic guide RNA for enhanced RNA stability and reduced cellular toxicity in CRISPR-Cas9 genome editing - Poster
-
Complete alignment identification of CRISPR-Cas9 genomic off-targets using Edit-R CRISPR specificity tool - Poster
-
CRISPR-Cas9 genome editing utilizing chemically synthesized RNA - Poster
-
CRISPR-mediated transcriptional activation and simultaneous gene knockout and activation with synthetic guide RNAs
-
Homology-directed repair with Edit-R CRISPR-Cas9 and single-strand DNA oligos - Poster
-
Increasing gene editing efficiencies in eukaryotic cell lines by selection of appropriate CRISPR-Cas9 reagents - Poster
-
Simultaneous gene knockout and transcriptional activation using S. pyogenes VPR-Cas9 mRNA
Product data
Protocols
-
10 mM Tris-HCl buffer pH 7.4 - Protocol
-
Cas9 Nuclease mRNA Electroporation - Protocol
-
Cas9 nuclease protein electroporation - Protocol
-
Edit-R 化学合成ガイドRNA再溶解プロトコール(チューブ用)
-
Fluorescent Cas9 Nuclease mRNA for enrichment of transfected cells - Protocol
-
T7E1, TIDE, and NGS analysis protocol for Dharmacon™ Edit-R™ gene editing experiments
Quick protocols
Safety data sheets
Selection guides
Technical manuals
-
CRISPR-Cas9 genome engineering with Cas9 nuclease expression plasmids and Edit-R synthetic RNA - Technical Manual
-
CRISPR-Cas9 genome engineering with Cas9 nuclease mRNA and Edit-R synthetic RNA - Technical Manual
-
CRISPR-Cas9 genome engineering with Cas9 nuclease protein and Edit-R synthetic guide RNA - Technical Manual
-
CRISPR-Cas9 genome engineering with lentiviral Cas9 particles and Edit-R synthetic guide RNA - Technical Manual
Related Products
A DNA-free option for Cas9 nuclease expression. Purified Cas9 nuclease protein with an enhanced hybrid NLS composition enabling more efficient nuclear delivery and gene editing. Suitable for a wide range of RNP workflows including nucleofection or co-transfection with Edit-R synthetic guide RNA for a completely DNA-free genome engineering system.
精製済みCas9 nuclease mRNAは、化学合成ガイドRNAとのコトランスフェクションで使用することにより完全にDNA-freeのゲノム編集を行えます。