Our emails have transitioned to the Revvity.com domain, however our billing information is unchanged. Click for details.
- CRISPRi (CRISPR interference) 試薬
- CRISPRi synthetic sgRNA
CRISPRmod CRISPRi synthetic sgRNA(化学合成sgRNA)
タンパク質をコードするヒト遺伝子の特異的な転写抑制のためのデザイン済みCRISPRiガイドRNA
- 化学合成sgRNAは、最も迅速なCRISPRiシステムであり、トランスフェクション後24時間以内に抑制を観察可能
- レンチウイルスフリーのCRISPRiワークフローのためのCRISPRi dCas9-SALL1-SDS3 mRNAとのコトランスフェクトまたはエレクトロポレートが可能
- プール試薬または個別試薬として利用可能

CRISPRi synthetic sgRNA
1Start Here
2Choose
CRISPRiによる効率的な遺伝子転写抑制
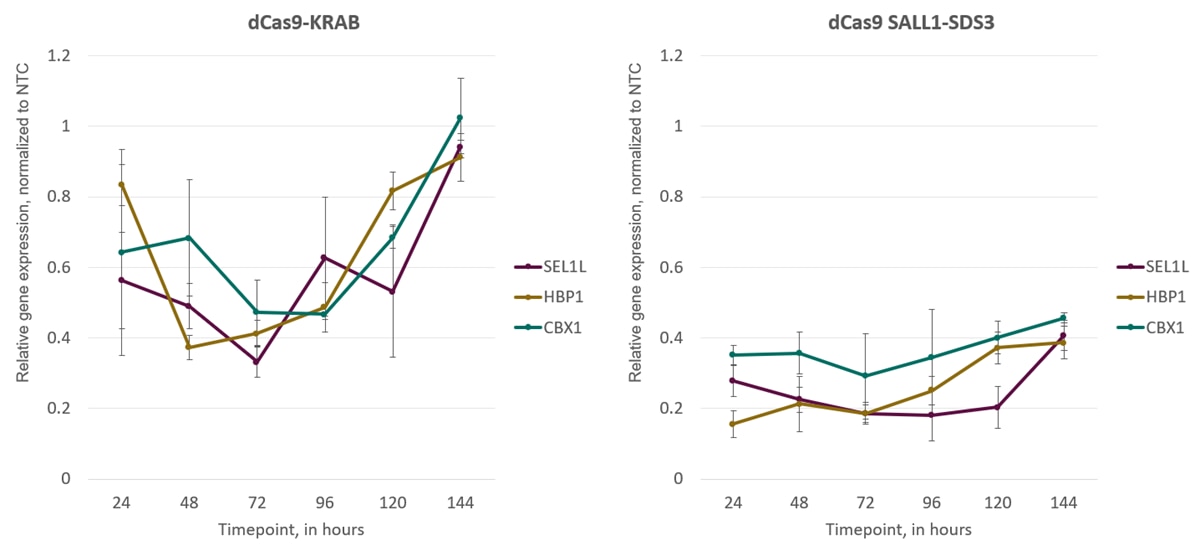
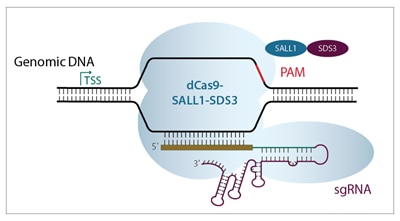
CRISPR interference(CRISPRi)は、CRISPR-Cas9遺伝子編集システムを改変したものです。HorizonのCRISPRmod CRISPRiシステムは、独自の転写リプレッサー(SALL1およびSDS3)に融合された触媒活性が不活性化されたCas9(dCas9)を利用します。転写開始部位(TSS)のすぐ下流の遺伝子を標的とするアルゴリズム設計のガイドRNAと組み合わせると、転写抑制が促進されます。
テクノロジーの概要は、 CRISPRiアプリケーションのページをご確認ください。
CRISPRi化学合成sgRNA試薬のハイライト
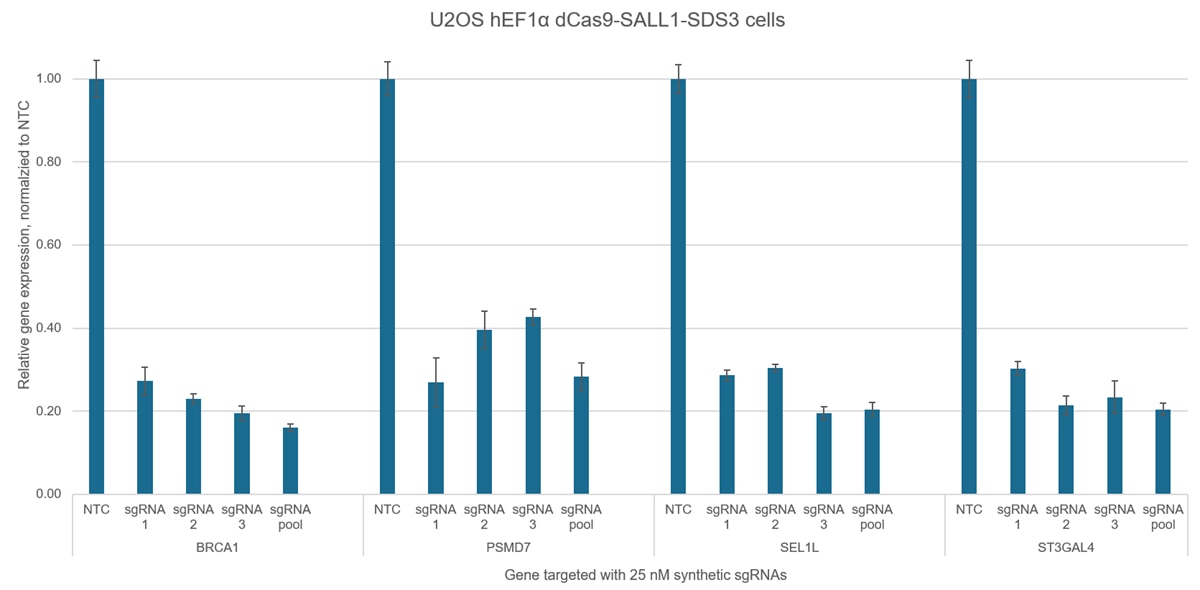
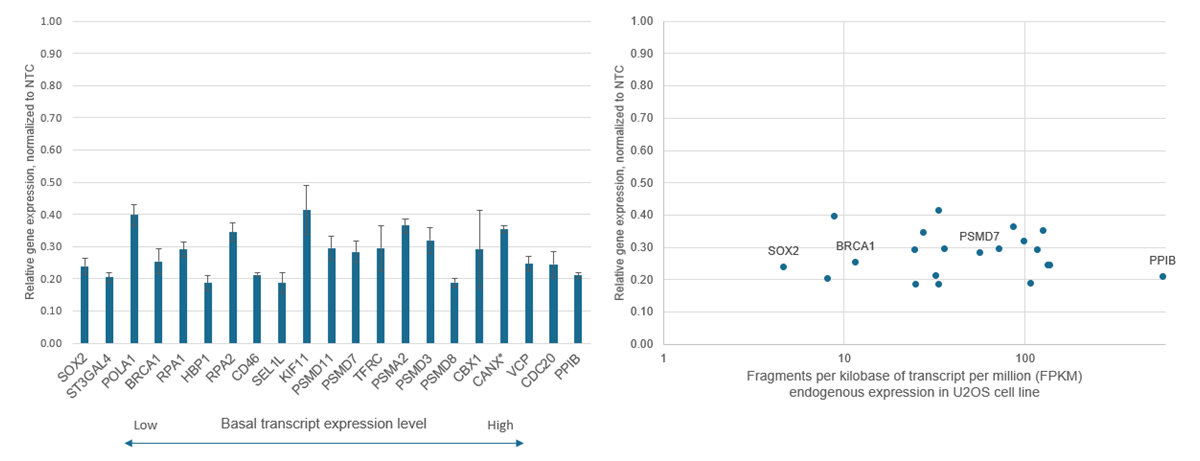
- CRISPRベースのPAMアンカーターゲティングは、非常に特異的な転写抑制を提供します(Supporting dataを参照)
- Horlbeckらによって公開されたアルゴリズムに基づいた設計です。最適化された設計による強力なレベルの遺伝子転写抑制を示しています(参考文献を参照)。
- 化学的に修飾されたCRISPRi sgRNAは、ヌクレアーゼ分解に対する安定性を高め、全体的なパフォーマンスを向上させます。
- 異なる転写開始部位を持つ遺伝子については、別個のガイドRNAデザインが利用可能です(P2とラベル付けされています)。
化学合成sgRNAを用いるCRISPRi実験に必要な試薬
- 標的遺伝子に対してデザインしたCRISPRi化学合成sgRNA。3種類のsgRNAを混合したPoolフォーマット、3本のチューブに個別に入れてセットとしたSet of 3フォーマット、個別包装のindividual フォーマットをご用意しています。
- CRISPRi dCas9-SALL1-SDS3 mRNA または lentiviral particles(ワークフローを参照)
- 適切なpositiveおよびnon-targetingコントロール
- ガイド導入用DharmaFECTトランスフェクション試薬またはエレクトロポレーション
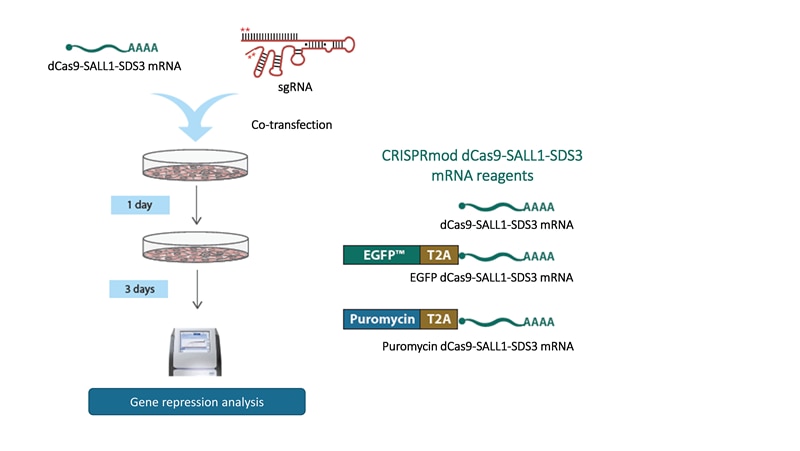
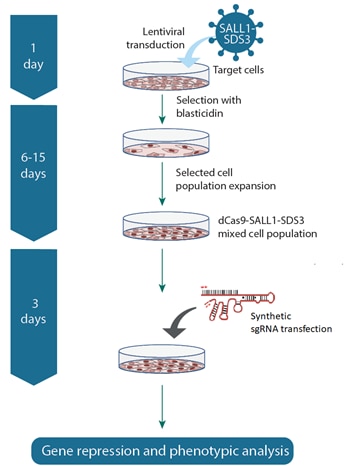
CRISPRi実験に推奨されるコントロールおよび関連試薬
CRISPRi dCas9-SALL1-SDS3
- CRISPRi dCas9-SALL1-SDS3 mRNAは、化学合成CRISPRi sgRNAとコトランスフェクトし、DNA-freeで実験を行うことができます。
Synthetic guide RNAコントロール
- CRISPRi synthetic sgRNA non-targetingコントロールは、遺伝子標的特異的sgRNAの非存在下でCRISPR-Cas9コンポーネントに対するベースライン細胞応答を評価するために使用します。ヒトゲノムの遺伝子を標的としないことが保証されている5つのスクランブル配列から選択します。
- 十分に特徴付けられた遺伝子を標的とするCRISPRi synthetic sgRNAポジティブコントロールは、最適な実験条件の評価のために使用します。PPIBまたはSEL1L1から選択します。
トランスフェクション試薬
- DharmaFECTトランスフェクション試薬は、デリバリーの改善と毒性の低減のために最適化されています。実験に最適なDharmaFECT試薬は、Cas9ヌクレアーゼソースと細胞タイプによって異なります。
| dCas9-SALL1-SDS3ソース | 推奨トランスフェクション試薬 |
|---|---|
| dCas9-SALL1-SDS3 lentiviral particles | DharmaFECT 1、2、3、または4を化学合成ガイドRNAのトランスフェクションに使用します。このDharmaFECT Cell Type Guideを参照し、細胞タイプに推奨されるDharmaFECT試薬をご確認ください。 |
| dCas9-SALL1-SDS3 mRNA | mRNAまたはタンパク質と化学合成ガイドRNAのコトランスフェクションの場合はDharmaFECT Duoを使用します。 |
- M. A. Horlbeck et al., Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760 (2016). https://doi.org/10.7554/eLife.19760
- C. Mills et al., A Novel CRISPR interference effector enabling functional gene characterization with synthetic guide RNAs. The CRISPR Journal. Vol 5, [6] (2022). http://doi.org/10.1089/crispr.2022.0056