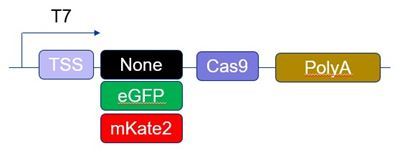
Cas9 nuclease mRNA
Cas9ヌクレアーゼ発現のためのDNA-freeオプション

CRISPR-Cas9システムにおけるCas9ヌクレアーゼ
CRISPR関連酵素Cas9は、ゲノムDNAの標的認識とDNA二本鎖切断(double-strand breaks: DSB)のためにガイドRNAを必要とするRNA-guidedエンドヌクレアーゼです。
Cas9 nuclease mRNAは、S. pyogenes Cas9ヌクレアーゼ遺伝子のヒトコドン最適化バージョンを2つの核局在化シグナル(nuclear localization signal: NLS)とともに発現しています。Cas9ヌクレアーゼは、ガイドRNAと複合体を形成し、ゲノムDNAの標的認識と切断を行います。Cas9 nuclease mRNAを用いて、完全なDNA-freeの方法でゲノム エンジニアリング実験を行うことができます。
Cas9 Nuclease mRNAは、mKate2またはEGFPのいずれかを共発現する形態もご用意しており、トランスフェクションの最適化、またはFACSを使用した濃縮を行うことができます。
Edit-R Cas9 mRNA highlights
Edit-R Cas9 mRNA highlights
- DNA-freeシステムは、不要な外来DNAを宿主細胞のゲノムに組み込む可能性を排除します。
- 蛍光Cas9 mRNAにより、編集された細胞集団の濃縮が可能になります (Supporting Data参照)。
- Cas9ヌクレアーゼの一過的な発現により、オフターゲットが低減し、より特異的な結果を得られます。
- Cas9プロモーターと細胞株の間の非適合性を考慮する必要がありません。
- DharmaFECT Duoトランスフェクション試薬を用いれば、Edit-R 化学合成ガイドRNAとCas9 mRNAを簡単にコトランスフェクトすることができます。または、トランスフェクションが困難な細胞にはコエレクトロポレーションします。
Cas9 nuclease mRNAおよびEdit-R 化学合成ガイドRNAを使用した遺伝子ノックアウト・ワークフロー
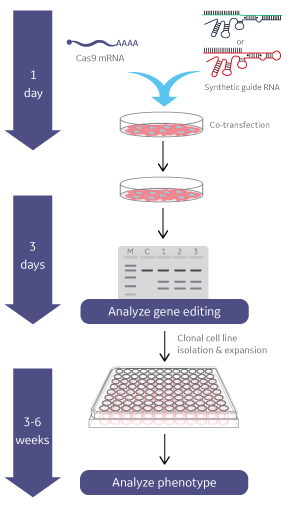
Cas9 Nuclease mRNAと化学合成gRNAあるいはcrRNA:tracrRNAによるゲノム編集は、DharmaFECT Duoトランスフェクション試薬(または目的の特定の細胞に適した他のDharmaFECTトランスフェクション試薬)を用いて、すべてのコンポーネントをコトランスフェクションすることにより行います。その後、表現型を直接観察することができます。DNAミスマッチ検出アッセイは、クローン細胞株の作製と特性評価の前に、ゲノム編集効率を推定するために使用できます。
蛍光Cas9 nuclease mRNAデータ
蛍光Cas9 nuclease mRNAは編集効率を維持する
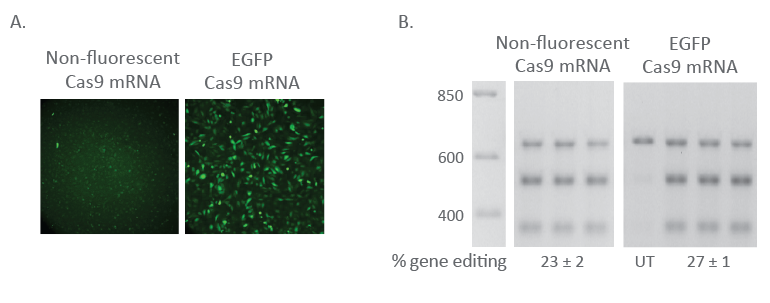
トランスフェクションの24時間前に、U2OS細胞を10,000細胞/ウェルで黒色の96ウェルプレートに播種しました。トランスフェクション時に、EGFP Cas9 nuclease mRNA(200 ng、カタログ番号:CAS11860)または非蛍光Cas9 nuclease mRNA(200 ng、カタログ番号:CAS11195)を、Edit-R PPIB化学合成crRNAコントロール(25 nM、カタログ番号:U-007000-01-05)およびtracrRNA(25 nM、カタログ番号:U-002005-05)と共に、DharmaFECT™ Duoトランスフェクション試薬(0.3 µL/ウェル)を使用して共導入しました。24時間後、Nikon Eclipse Ti-S/L100倒立顕微鏡を使用して、細胞のEGFP蛍光を画像化しました(A)。トランスフェクションの72時間後に細胞を回収し、DNAミスマッチ検出アッセイによってゲノム編集を測定しました(B)。
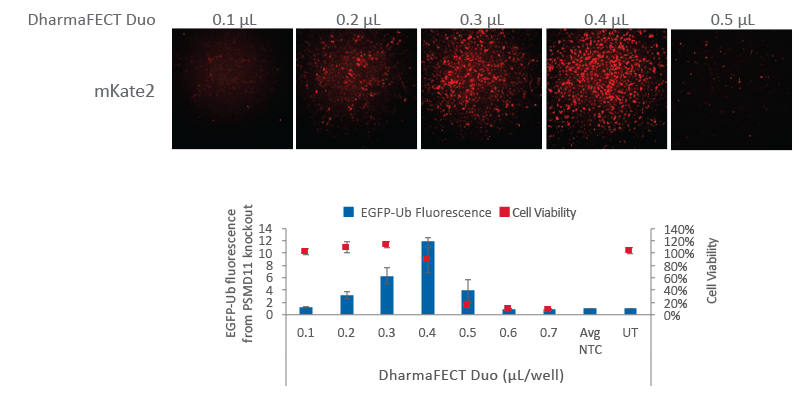
蛍光レポーターを含むCas9 nuclease mRNAの最適なトランスフェクション条件
ユビキチンタグ付きEGFPを発現するU2OS細胞を、黒色の96ウェルプレートに10,000細胞/ウェルで播種し、Cas9 mRNA(200 ng、カタログ番号:CAS11859)とともに、既知のプロテアソーム成分であるPSMD11(25 nM、カタログ番号:CM-085161-03)を標的とする化学合成crRNA、化学合成tracrRNA(25 nM、カタログ番号:U-002005- 05)をコトランスフェクションしました。Non-targetingコントロール(NTC、Cat #U-007501-xx)をネガティブコントロールとして使用しました。ウェルあたりのDharmaFECT Duoトランスフェクション試薬の量を変えてテストしました(0.1 µL~0.7 µL)。24時間後、細胞はIn Cell Analyzer 2200を使用してmKate2蛍光を画像化しました。トランスフェクションの72時間後、細胞のプロテアソームノックアウトから生じるEGFP蛍光を画像化しました。mKate2蛍光から、最適なトランスフェクション条件は、DharmaFECT Duoがウェルあたり0.3~0.4 μLである時であることがわかりますが、これは表現型効果の増加に対応しています。DharmaFECT Duoがウェルあたり0.4 μLを超えると、有意な細胞死が観察されます(グラフ、赤ボックス)。UT = untransfected sample
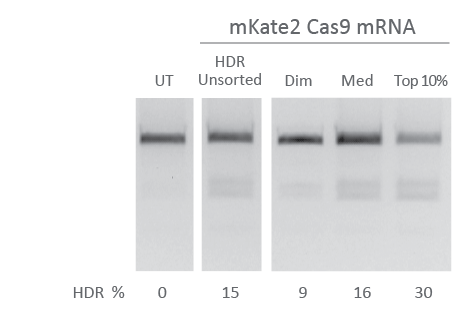
蛍光Cas9 mRNAによるHDR編集細胞の濃縮
U2OS細胞に、Edit-R mKate2-Cas9 mRNA(200 ng、カタログ番号:CAS11859)、EMX1を標的とするcrRNA(25 nM、カスタム合成)、tracrRNA(25 nM、カタログ番号:U-002005)および一本鎖ドナーをトランスフェクトしました。オリゴ(5 nM、Edit-R HDR Donor Designerで設計)を使用して、EMX1のC末端にNheI制限部位とFLAGタグ配列を挿入しました。96ウェルプレートのウェルあたり10,000 U2OS細胞をプレーティングしてから24時間後に、DharmaFECT Duoトランスフェクション試薬(0.3 μL/ウェル)を使用してトランスフェクションを行いました。トランスフェクションの24時間後に細胞を回収し、mKate2蛍光サブポピュレーション(Dim mKate2蛍光、Medium、またはTop 10%蛍光のいずれか)に分類しました。亜集団をさらに48時間増殖させ、EMX1 PCRアンプリコンをNheI制限酵素で処理することにより、制限酵素断片長多型(RFLP)によるNheI挿入について分析しました。
Cas9 nuclease mRNAデータ
3つの細胞株におけるCas9 nuclease mRNAを用いたゲノム編集
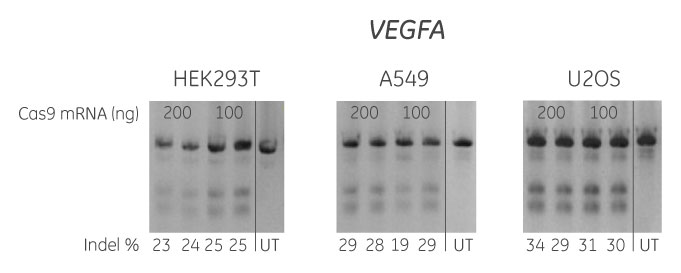
HEK293T、A549およびU2OS細胞を96ウェルプレートに20,000 細胞/ウェルで播種し、Cas9 mRNA(200 または100 ng)およびVEGFAを標的とする化学合成crRNA:tracrRNA(50 nM)を、DharmaFECT Duoトランスフェクション試薬を使用してコトランスフェクションしました。トランスフェクションの72時間後に細胞を回収し、T7エンドヌクレアーゼ I を用いたDNAミスマッチ検出アッセイに基づいて、ゲノム編集の相対頻度を計算しました。化学合成crRNAは、Dharmacon CRISPR Design Toolを使用して設計しました。UT = untransfected sample
効率的なゲノム編集のためのCas9ヌクレアーゼmRNAの用量曲線
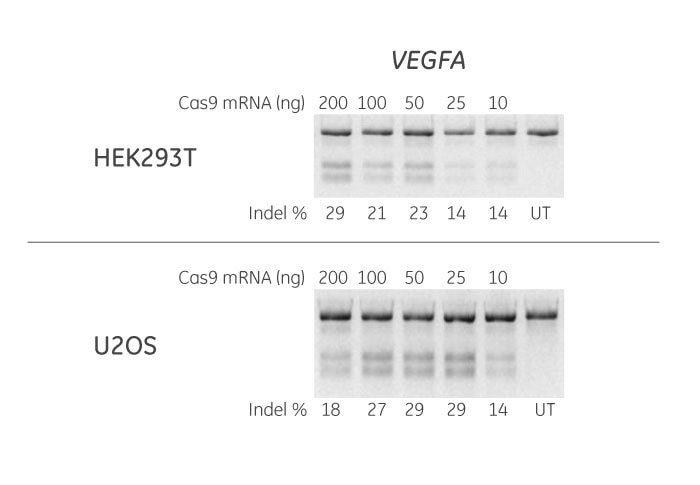
HEK293TおよびU2OS細胞を96ウェルプレートに20,000 細胞/ウェルで播種し、Cas9 mRNA(200、100、50、25または10 ng)および、VEGFAを標的とする化学合成crRNA:tracrRNA(50 nM)をDharmaFECT Duoトランスフェクション試薬を使用してコトランスフェクションしました。トランスフェクションの72時間後に細胞を採取し、T7エンドヌクレアーゼ IによるDNAミスマッチ検出アッセイに基づいてゲノム編集の相対頻度を計算しました。使用する細胞およびアプリケーションにおいて、Cas9 mRNAの量を最適化することをお勧めします(96ウェルで50~200 ng/ウェル形式)。VEGFAを標的とする化学合成crRNAは、Dharmacon CRISPR Design Toolを使用して設計しました。UT = untransfected sample
2x MS修飾または非修飾crRNA:tracrRNA によるゲノム編集の効率
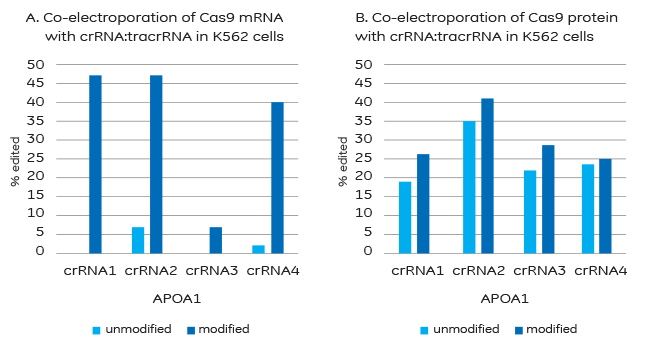
A. ヌクレアーゼによる分解をブロックするための修飾が、Cas9 mRNAとcrRNA:tracrRNAのコエレクトロポレーションを成功させるには必要です。
B. Cas9タンパク質とcrRNA:tracrRNAのコエレクトロポレーションは安定化修飾を必要としませんが、それらを追加することで結果が改善される場合があります。
Application notes
-
crRNAを使用したKO, KIのの濃縮のための蛍光Cas9 mRNA - Application Note
-
Dharmacon Edit-R 化学合成crRNAペアを用いたCRISPR-Cas9システムによるヒトmicroRNA(has-miR-221)の機能的ノックアウト - Application Note
-
ipsc-gene-knockout-appnote
-
Microinjection of zebrafish embryos using Dharmacon Edit-R Cas9 Nuclease mRNA, synthetic crRNA, and tracrRNA for genome engineering - Application Note
Posters
Protocols
-
Cas9 Nuclease mRNA Electroporation - Protocol
-
Fluorescent Cas9 Nuclease mRNA for enrichment of transfected cells - Protocol
-
T7E1, TIDE, and NGS analysis protocol for Dharmacon™ Edit-R™ gene editing experiments
-
Transfection of ssDNA donor oligonucleotides for HDR-mediated gene alterations using the Dharmacon Edit-R system - Protocol