Edit-R デザイン済みall-in-one Lentiviral sgRNA
sgRNAとCas9ヌクレアーゼの発現を1つのベクターに組み合わせ、導入を簡素化
- 目的の標的遺伝子の編集(DNA切断)が保証されています。
- ピューロマイシン耐性と蛍光レポーターを用いた濃縮が可能です。
- 形質導入に対応したガイドRNAにより、クローニングとin vitro転写ステップが不要です。
- タンパク質の機能的ノックアウトの可能性を最大化し、オフターゲット編集を最小化するようにデザインされています。
- グリセロールストックおよび高力価精製粒子をご用意しています。


Edit-R デザイン済みall-in-one Lentiviral sgRNA
1Start Here
2Choose
必要なすべてのコンポーネントを単一の試薬に組み合わせて、信頼性の高い遺伝子ノックアウトを実現します。
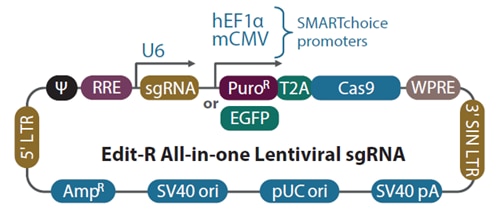
Edit-R all-in-oneレンチウイルスシステムを用いて、ノックアウト細胞株を簡単に作製できます。遺伝子特異的ガイドRNAとCas9ヌクレアーゼの両方を発現する単一のベクターを使用すると、逐次の形質導入を実行する必要がなくなります。
このガイドフォーマットは、トランスフェクションが困難な細胞タイプでノックアウトを作製するため、またはプール化レンチウイルスsgRNAライブラリーを用いるスクリーニング後のヒットをフォローアップするために最も使用されます。
ピューロマイシン耐性遺伝子またはEGFPマーカーを選択することで、ベクターの組み込みに成功した細胞を選択できます。蛍光が発現するとすぐにFACS分析を実行できるため、編集した細胞を迅速に濃縮するには、EGFP選択マーカーをお薦めします。これは、初代細胞など生存期間の短い細胞タイプの場合に特に役立ちます。
Edit-R デザイン済みall-in-one lentiviral sgRNAは、次の製品形態でご用意しています。
- 高品質の精製された濃縮レンチウイルス粒子(107 TU/mL以上)は、精製物中の毒性のあるデブリや夾雑物なしに、細胞に直接形質導入することができます。
- グリセロールストックは、ウイルスパッケージングに使用できます。
Edit-Rのガイドデザイン
CRISPR-Cas9は遺伝子機能を調べるための非常に効果的なツールですが、すべてのガイドRNAがタンパク質の機能的なノックアウトを実現するのに十分効果的であるとは限りません。この問題に対処するために、Dharmaconは、挿入または欠失を起こすだけでなく、機能的な遺伝子ノックアウトを生じる可能性が最も高いガイドを選択するように検証されたアルゴリズムを開発しました。広範なテストにより、このアプローチはノックアウト効率と特異性の比類のない組み合わせを提供することが示されています。
New! ヒトガイドRNAについて、機能性と特異性のスコアが利用可能になりました。
すべてのガイドRNAデザインは、各遺伝子についてアルゴリズムで選択した最上位のものです。機能性と特異性の定性的なランク付けにより、特定のアプリケーションに合わせて最適なヒトガイドRNAを選択できます。機能性スコアは、このガイドがどの程度機能的ノックアウトをもたらす可能性があるかを予測したものです。特異性スコアは、潜在的なオフターゲット部位での切断活性の予測リスクに基づいています。詳細については、Edit-RガイドRNAのアルゴリズムページをご覧ください。
Edit-RガイドRNA保証
すべてのデザイン済みEdit-RガイドRNAは、Edit-Rテクニカルマニュアルの記載に従い細胞導入した場合に、標的領域で編集(DNA切断)ができることを保証します。
Edit-RガイドRNA保証は、野生型S. pyogenes由来Cas9ヌクレアーゼ(タンパク質、mRNA、発現用プラスミド、または発現用レンチウイルスベクター)とともに使用した場合に適用されます。更にEdit-R 化学合成crRNAは、Edit-R tracrRNAとともに使用した場合に適用されます。
T7EIまたはSurveyorミスマッチ検出アッセイを使用して、試薬で処理された細胞集団の編集(DNA切断)の分析結果を提示する必要があります。同時並行で適切に実施されたEdit-R ポジティブコントロールが編集(DNA切断)に成功する一方で、Edit-Rデザイン済みガイドRNAによる編集(DNA切断)が成功しない場合、同じフォーマットで同じ容量の、異なるEdit-Rデザイン済みガイドRNAの交換製品が、1回限り無償で提供されます。
交換製品の提供は、テクニカルサポートチームとの話し合いでのみ承認されます。
DNAレベルでの編集(切断)の成功は、常に機能的な遺伝子ノックアウトにつながるとは限りません。複数のガイドRNAをテストして、標的遺伝子のノックアウトに最も効果的なガイドRNAを決定することをお薦めします。
この保証は付随する実験費用には適用されず、CRISPR Design Toolを介して注文されたガイドRNAには適用されず、また本保証により交換したガイドRNAには適用されません。
Edit-R all-in-one lentiviral sgRNAシステムを用いた遺伝子ノックアウトワークフロー
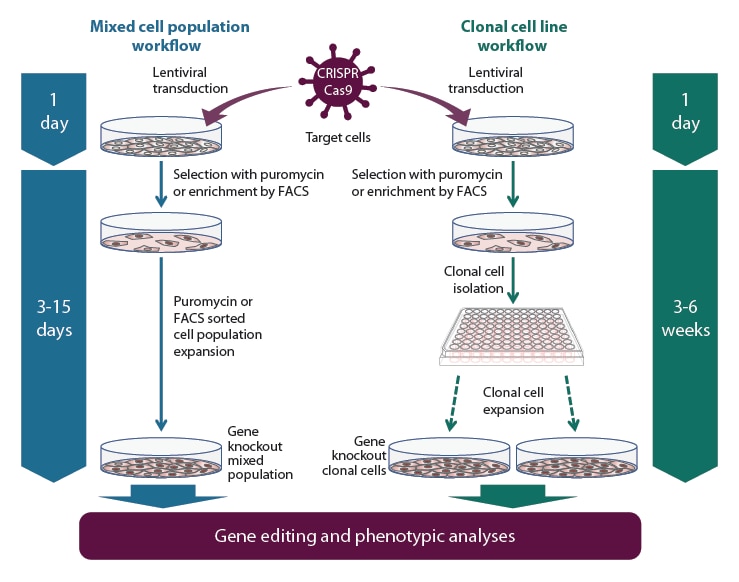
混合集団(左側)またはクローン細胞株(右側)実験アプローチを用いた all-in-one lentiviral sgRNA による遺伝子ノックアウトワークフロー。細胞集団全体にわたって均質な遺伝子減少が必要な場合は、クローン分離ワークフローを推奨します。更には、目的の遺伝子をノックアウトすることが致死的であったり、細胞増殖を阻害する場合、選択後の細胞は野生型を保持しているか、下流のタンパク質発現に影響を与えないインフレーム変異を有している可能性が高いことに留意してください。
Edit-R all-in-one lentiviral sgRNAコントロール
-
ポジティブコントロールと検出プライマー
all-in-one lentiviral sgRNA用コントロールを使用して、DNA二本鎖切断とゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。
-
non-targetingコントロール
all-in-one lentiviral sgRNAコンストラクトは、ヒト、マウス、またはラットのゲノム内の遺伝子を標的としないようにバイオインフォマティクスによって設計および検証されています。
-
Cutting controls
Lentiviral sgRNA constructs bioinformatically designed and validated to not target any gene in human or mouse genomes.
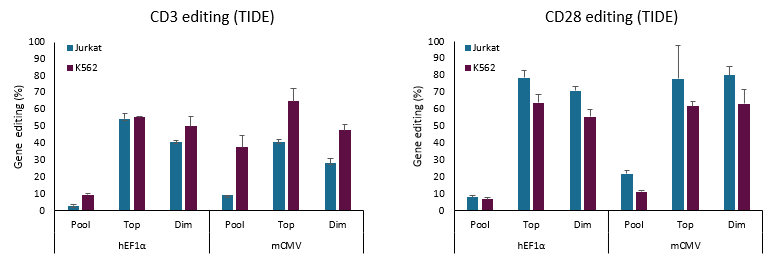
Edit-R all-in-oneレンチウイルスシステムを用いた場合の編集頻度(TIDE)
JurkatまたはK562細胞に、CD3またはCD28遺伝子のいずれかを標的とするEdit-R all-in-one EGFPレンチウイルスsgRNAベクターを用いて、0.3 MOIで形質導入しました。all-in-oneレンチウイルス粒子は、hEF1αまたはmCMVプロモーターの下で構成的にCas9ヌクレアーゼを発現します。non-targetingコントロール(NTC)ガイドを発現するレンチウイルス粒子をネガティブコントロールとして使用しました。レンチウイルスの形質導入と増殖の後、細胞をソーティングせず細胞集団(Pool)として分析するか、またはソーティングを行って、最も明るい(Top)または最も暗い(Dim)EGFP発現細胞集団を濃縮しました。次に、細胞のPool、Top、およびDimの集団をTIDE分析にかけ、ゲノム編集の割合を評価しました。
Edit-Rの 2ベクターシステムとall-in-oneシステムの比較
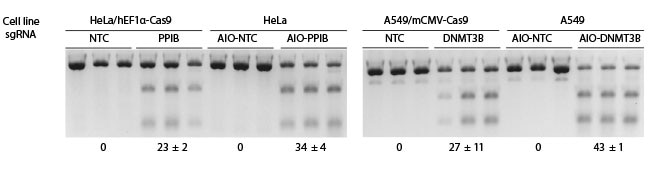
2ベクターシステムの場合、hEF1αまたはmCMVの制御下でCas9を構成的に発現するためのレンチウイルス粒子を細胞に0.3 MOIで形質導入し、10 µg/mLブラストサイジンで10日間選択した後、all-in-one non-targetingコントロール(NTC)あるいは、DNMT3BまたはPPIBをターゲットとするsgRNAレンチウイルス粒子を0.3 MOIで形質導入しました。all-in-oneシステムでは、all-in-one non-targetingコントロール(NTC)あるいは、DNMT3BまたはPPIBをターゲットとするAll-in-oneレンチウイルスsgRNAを野生型細胞に0.3 MOI にて形質導入し、2 µg/mLピューロマイシンで3日間選択しました。次に、細胞を溶解し、T7EIを用いたDNAミスマッチ検出アッセイを使用してindel(挿入・欠失)を分析しました。
Edit-R all-in-one Lentiviral sgRNAのベクターマップ
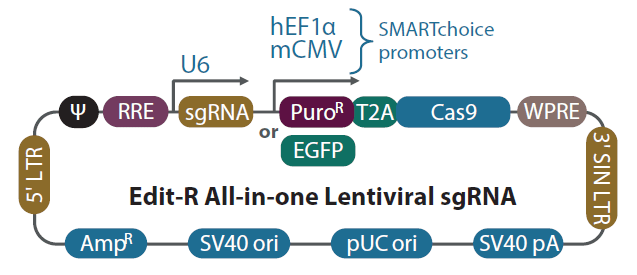
Edit-R all-in-one Lentiviral sgRNAベクターバックボーンでは、遺伝子特異的ガイドRNAはヒトU6プロモーターの制御下で発現し、Cas9およびピューロマイシン耐性マーカー(PuroR)またはEGFPマーカーの発現はヒトEF1αまたはマウスCMVプロモーターのいずれかから駆動されます。プラスミドには、大腸菌での増殖と選択のためのアンピシリン耐性マーカーが含まれています。
| Vector Element | Utility |
|---|---|
| Cas9 | Human codon-optimized S. pyogenes Cas9 nuclease for cleavage of targeted DNA when programmed with a sgRNA |
| T2A | Self-cleaving peptide allows for simultaneous expression of puromycin resistance and Cas9 protein from a single transcript |
| PuroR | Puromycin resistance marker permits antibiotic selection of transduced mammalian cells |
| EGFP | Fluorescent marker permits FACS selection of transduced mammalian cells |
| mCMV | Mouse cytomegalovirus immediate early promoter |
| hEF1α | Human elongation factor 1 alpha short promoter |
| U6 | Human RNA polymerase III promoter U6 |
| sgRNA | Optimized single guide RNA, a fusion of gene-specific crRNA with the tracrRNA scaffold |
| 5' LTR | 5' Long Terminal Repeat necessary for lentiviral particle production and integration of the construct into the host cell genome |
| Ψ | Psi packaging sequence allows lentiviral genome packaging using lentiviral packaging systems |
| RRE | Rev Response Element enhances titer by increasing packaging efficiency of full-length lentiviral genomes |
| WPRE | Woodchuck Hepatitis Post-transcriptional Regulatory Element enhances transgene expression in target cells |
| 3' SIN LTR | 3' Self-inactivating Long Terminal Repeat for generation of replication-incompetent lentiviral particles |
| SV40 pA | Simian virus 40 polyadenylation signal |
| pUC ori | pUC origin of replication |
| SV40 ori | Simian virus 40 origin of replication |
| AmpR | Ampicillin resistance gene for vector propagation in E. coli cultures |
LentiBOOST Lentivirus Transduction Enhancer is a uniquely formulated transduction reagent that can be used with or without lentivirus spinfection in order to increase successful viral transduction events while preserving cell viability. Especially critical for preserving precious primary cells from patient cohorts, or, for engineering complex animal models; improving transduction efficiency can save time and costs by increasing the success of each editing/transduction step, or, even avoid the loss of irreplaceable samples. Additionally, LentiBOOST technology is already used in the manufacturing of a number of clinical stage therapies providing the opportunity to demonstrate improved workflow applicability to the clinic.
LentiBOOST can be purchased through the Dharmacon Reagents catalog.
To learn more about LentiBOOST technology visit the Revvity LentiBOOST webpage.
Supporting Data
Improved CD8+ T-cell SMARTvector™ shRNA lentiviral system transduction using LentiBOOST™ Lentivirus Transduction Enhancer
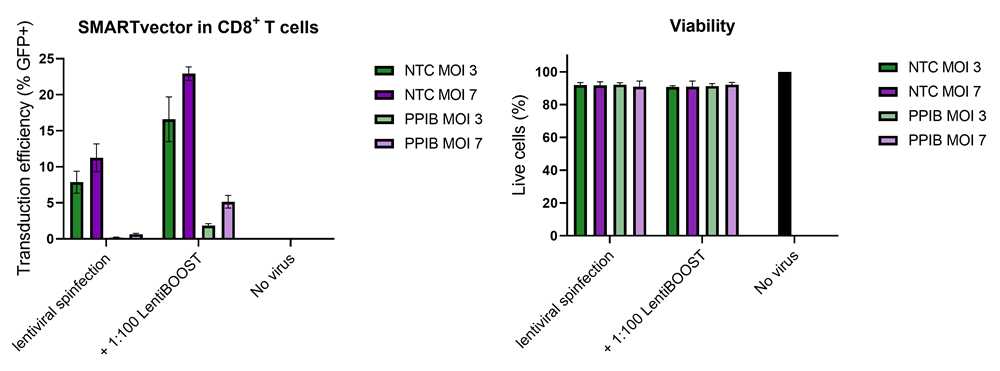
100,000 primary human CD8+ T cells were transduced with either 30,000 (MOI 3, green) or 70,000 (MOI 7, purple) TUs of SMARTVector™ mCMV tGFP Lentiviral Control Particles targeting either NTC or PPIB along with 1:100 LentiBOOST transduction enhancer. Cells were centrifuged at 800 x g for one hour at 32 °C followed by a four hour incubation prior to removal of lentiviral particles and transduction enhancer. Transduction efficiency (%GFP+ out of live cells) and viability were determined 5 days post-transduction by flow cytometry. The addition of LentiBOOST technology markedly improved transduction efficiencies without significantly impacting cell viability.
Improved CD4+ and CD8+ T-cell Edit-R™ All-in-one sgRNA/Cas9 lentiviral system transduction using LentiBOOST™ Lentivirus Transduction Enhancer
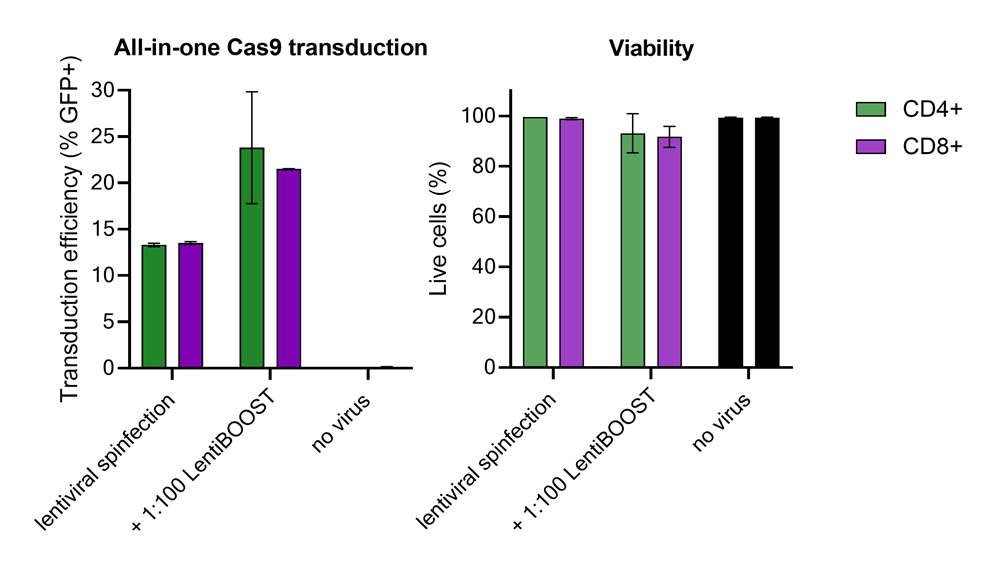
100,000 primary human CD4+ and CD8+ T cells from two donors were transduced with 250,000 TUs of Edit-R GFP Delivery controls mCMV along with 1:100 LentiBOOST transduction enhancer. Cells were centrifuged at 800 x g for one hour at 32 °C followed by an overnight incubation prior to removal of lentiviral particles and transduction enhancer. Transduction efficiency and viability were determined 72 hours post-transduction by flow cytometry. The addition of LentiBOOST technology markedly improved transduction efficiencies without significantly impacting cell viability.
Improved transduction of human induced pluripotent stem cells (hiPSCs) with the Strict-R™ Inducible CRISPRa lentiviral system transduction using LentiBOOST™ Lentivirus Transduction Enhancer

10,000 WTC hiPS cells were transduced with either 20,000 (MOI 2, green) or 40,000 (MOI 4, purple) TUs of Strict-R™ Inducible EGFP dCas9-VPR Lentiviral Particles along with 1:100 LentiBOOST transduction enhancer. Cells were centrifuged at 800 x g for one hour at 32 °C followed by an overnight incubation prior to removal of lentiviral particles and transduction enhancer. Transduction efficiency and viability were determined 72 hours post-transduction by flow cytometry. The addition of LentiBOOST markedly improved transduction efficiencies without significantly impacting cell viability.
Application notes
Product inserts
Protocols
Safety data sheets
Selection guides
Related Products
Edit-R all-in-one lentiviral sgRNA non-targetingコントロールは、実験ベースラインを確立し、配列特異的な生物学的効果と非特異的効果を区別します。製品形態は、高力価の精製レンチウイルス粒子およびグリセロールストックフォーマットをご用意しています。
DNA二本鎖切断およびゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用するコントロール
LentiBOOST transduction enhancer can increase successful viral transduction in challenging to transduce cells, or, complex cellular engineering work; while preserving cell viability and minimizing the amount of viral particles required for your experiment. LentiBOOST technology is actively used in the production of clinical stage lentivirally delivered therapies, including some approved therapies, providing a direct path to therapeutic applicability for your research studies. Tested with Dharmacon Lentiviral reagents.
信頼性の高い遺伝子ノックアウトに必要なすべてのコンポーネントを単一の試薬に統合することにより、コストのかかるインフラを必要とせずに、偏りのない表現型ノックアウトスクリーニングを実施できます。