- Dharmaconスクリーニングライブラリー
- Edit-R synthetic sgRNAライブラリー
Edit-R synthetic sgRNA(化学合成sgRNA)ライブラリー
初代細胞を含むあらゆる細胞タイプでCRISPRゲノム編集を簡素化するCRISPRライブラリー
アルゴリズムにより最適化された化学合成シングルガイドRNAが、スクリーニングからのヒットの特異性を確保します。
ヒトの遺伝子ファミリー全体にわたるスクリーニングのための、デザイン済み化学合成sgRNAのアレイ化ライブラリーコレクションです。迅速な機能喪失研究のために、ヒト遺伝子ファミリーおよびパスウェイを標的とするアレイ化されたライブラリーとしてご提供します。

Edit-R™ 化学合成sgRNAライブラリーを使用したCRISPR-Cas9ゲノム編集で、機能ゲノミクス研究を推進してください。遺伝子ごとに複数の標的部位に対してデザインしたsgRNAを用いて数百の遺伝子を分析します。コレクションには、全ゲノム、ヒトDruggableゲノム、および汎用される遺伝子ファミリーライブラリーをご用意しています。
アレイ化ライブラリーフォーマットにより、ハイコンテントアッセイやその他の形態学的またはレポーターアッセイなどの多くの表現型アッセイが単一ウェルで可能になります。
New! Edit-R human sgRNA designs have been updated to the latest RefSeq in 2025 providing the most specific and genomically relevant guides for producing efficient protein knockout. This allows the Edit-R algorithm to target the latest genome annotations more accurately and efficiently providing you with the best solution for your research needs. Please reach out to Scientific Supportif you have any questions.
Highlights:
- sgRNAは、Edit-R™アルゴリズムによって選択されます。高機能であり、特異的な配列を標的とするため、堅牢で信頼性の高い遺伝子ノックアウトを実現します。ヌクレアーゼ耐性を改善するために化学修飾しています。
- New! Edit-R human sgRNA designs have been updated to the latest RefSeq in 2025 providing the most specific and genomically relevant guides for producing efficient protein knockout. This allows the Edit-R algorithm to target the latest genome annotations more accurately and efficiently providing you with the best solution for your research needs. Please reach out to Scientific Support if you have any questions.
-
信頼性の高い表現型の結果を得るために、遺伝子ごとに3つの配列の異なるsgRNAがデザインされ、プール(混合)されて提供されます。
- 96ウェルまたは384ウェルプレートにアレイ化された遺伝子ファミリーコレクションとしてご用意しています。ご要望に応じて、Echo-qualified 384ウェルプレートをご利用可能です。
- ご自身のご希望の遺伝子リストをアップロードしてプレートをカスタマイズできるsgRNA cherry-pickライブラリーもご利用いただけます。
Human Druggableは、Proteases、Protein Kinases、Phosphatases、Transcription Factors、Ubiquitin Enzymes、GPCRs、Ion Channels and Drug Targetsで構成されています。
| Human synthetic sgRNA libraries | Approximate number of genes |
|---|---|
| Whole Genome | 18,880 |
| Druggable Genome | 8,270 |
| GPCRs | 470 |
| Ion Channels | 430 |
| Phosphatases | 310 |
| Proteases | 700 |
| Protein Kinases | 760 |
| Ubiquitin Enzymes | 750 |
| Transcription Factors | 1580 |
| Drug Targets | 3300 |
| Apoptosis | 550 |
| Cell Cycle Regulation | 170 |
| Cytokine Receptors | 140 |
| Deubiquitinating Enzymes | 110 |
| DNA Damage Response | 240 |
| Epigenetics | 860 |
| Membrane Trafficking | 140 |
| Nuclear Receptors | 50 |
| Tyrosine Kinases | 90 |
標的遺伝子ごとにデザイン済みの化学合成sgRNA混合物(プール)を用いた、CD4 + T細胞におけるタンパク質ノックアウトのフローサイトメトリー分析
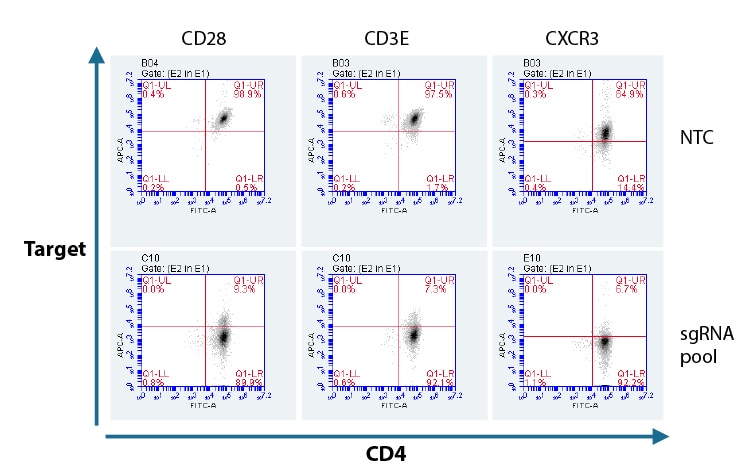
Lonza 96-well Shuttleシステムにより、初代ヒトCD4 + T細胞に、標的遺伝子ごとにデザイン済みの個別のEdit-R化学合成sgRNAの3つの混合物(プール)またはNon-targetingコントロール(NTC)とCas9からなるRNPをヌクレオフェクションしました。72時間後、FACS分析により、CD28、CD3E、およびCXCR3の機能的ノックアウトを、標的遺伝子を発現していない細胞の割合として評価しました。陽性発現コントロールとして、細胞のCD4発現をAlexa Fluor 488標識抗体で染色し、APC標識一次抗体を使用してCD28、CD3EおよびCXCR3各標的遺伝子ノックアウトと比較しました。
CD4+ T細胞の遺伝子ノックアウトにおける化学合成ガイドRNAフォーマットの比較
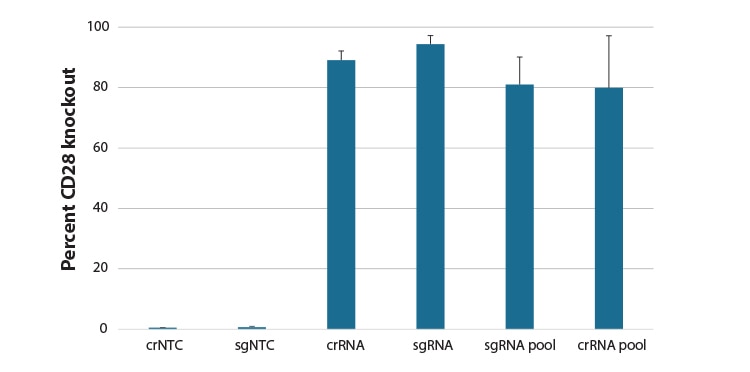
ヒト初代CD4+ T細胞は、Edit-Rデザイン済み化学合成crRNA:tracrRNA、化学合成sgRNA、3つの異なる配列のデザイン済み化学合成sgRNAの混合物(プール)、またはCD28を標的とする4つの異なる配列のデザイン済みcrRNA:tracrRNAsの混合物(プール)、またはnon-targetingコントロール(NTC)を使用して、Lonza 96-well ShuttleシステムによりCas9 RNPでヌクレオフェクトされました。72時間後、CD28に対するAPC標識一次抗体を用いたFACS分析により、CD28の機能的ノックアウトをCD28陰性細胞のパーセントとして評価しました。
化学合成sgRNAの混合物(プール)によるFUS遺伝子の機能的ノックアウト
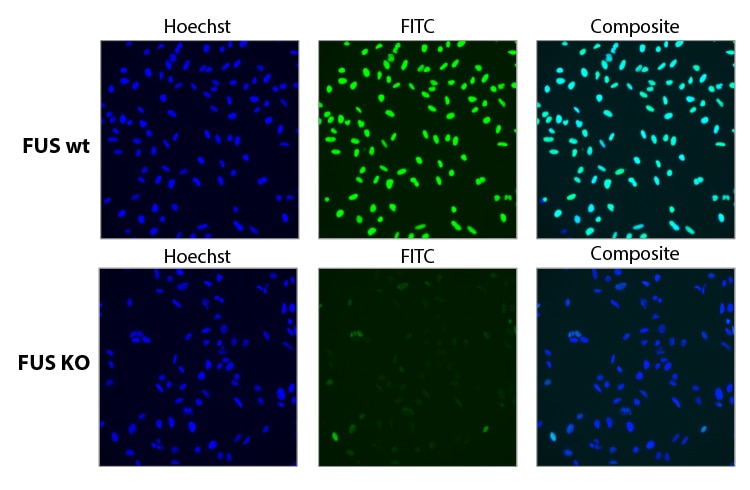
FUSの機能的ノックアウトを、CAGプロモーター制御下でCas9を恒常的に発現するU2OS細胞で評価しました。0.04 µL DharmaFECT 4を用いて、FUSをターゲットとする3つの異なるEdit-R デザイン済みsgRNAプール(25 nM)を細胞にトランスフェクトしました。トランスフェクションから96時間後に細胞固定後、FUSを標的とする一次抗体およびAlexa Fluor 488蛍光標識二次抗体で染色しました。核識別のためにHoechst染色を使用しました。
ヌクレアーゼ安定化修飾によりゲノム編集効率が向上
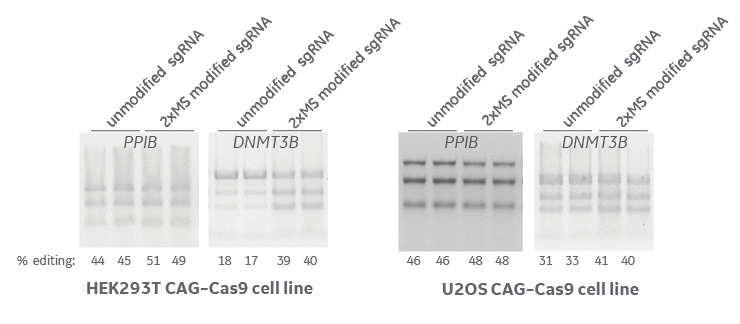
CAGプロモーター制御下でCas9を発現するHEK293TおよびU2OS細胞株に、PPIBおよびDNMT3B遺伝子を標的とする未修飾および5'および3' 2 xMS修飾化学合成sgRNAをトランスフェクトしました。72時間後に、編集効率をミスマッチ検出アッセイ、T7エンドヌクレアーゼI (T7EI)で評価しました。いずれの場合も、安定化修飾により遺伝子編集効率が向上しました。
MS:2'-O-メチルヌクレオチドおよびホスホロチオエートバックボーン結合
Edit-R化学合成ガイドRNAは、in vitroで転写されたガイドRNAと比較して、自然免疫応答や毒性を実質的に引き起こさない

Cas9を発現するHEK293T細胞株に、未修飾のcrRNA:tracrRNA、crRNAの5'末端およびtracrRNAの3'末端を2xMSで修飾したcrRNA:tracrRNA、crRNAの5'末端およびtracrRNAの3'末端を3xMSで修飾したcrRNA:tracrRNA、未修飾化学合成sgRNA、5'および3' 末端を2xMSまたは3xMSで修飾した化学合成sgRNA、in vitro転写(IVT)sgRNAを含むさまざまな化学合成ガイドRNA(PPIBおよびDNMT3B遺伝子を標的)フォーマットをトランスフェクトしました。72時間後にレサズリン還元アッセイ(赤点)で生存率を評価し、5つの免疫応答遺伝子のレベルをRT-qPCRで定量化しました。
MS:2'-O-メチルヌクレオチドおよびホスホロチオエートバックボーン結合
Edit-R化学合成sgRNAのヌクレアーゼ耐性を改善するための修飾の構造
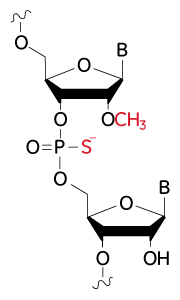
ヌクレアーゼ耐性を改善するための修飾:2'-O-メチル修飾ヌクレオチドおよびホスホロチオエート骨格結合
Edit-R化学合成シングルガイドRNAの配列構造
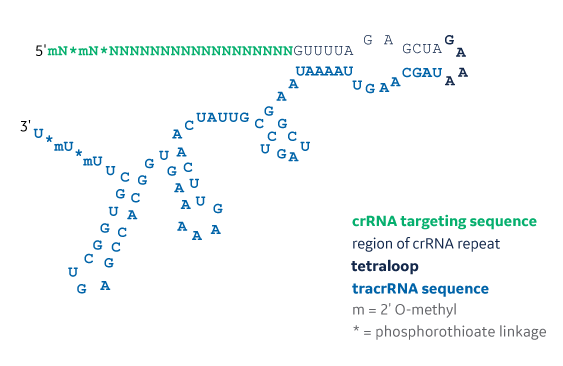
分子の5'末端と3'末端の両方において2xMSヌクレアーゼ安定性修飾を持つEdit-R化学合成シングルガイドRNAの構造。この構造には、20 ntターゲティングシーケンス(ポリNsとして緑色で表示)、12 nt crRNAリピートシーケンス(薄グレーで表示)、4 ntテトラループシーケンス(太字の黒で表示)、および64 nt tracrRNAシーケンス(青で表示)が含まれます。)。
MS:2'O-メチルヌクレオチドおよびホスホロチオエート結合(MS)
- R. Barrangou, A. Birmingham, et. al. Advances in CRISPR-Cas9 genome engineering: lessons learned from RNA interference. Nucleic Acids Research, 43(7) 3407-3419 (2015)
- M.L. Kelley, Ž. Strezoska, et al. Versatility of chemically synthesized guide RNAs for CRISPR-Cas9 genome editing. J. Biotechnol. 233, 74–83 (2016). doi:10.1016/j.jbiotec.2016.06.011
- Basila, M., M. L. Kelley, et al.. Minimal 2’-O-methyl phosphorothioate linkage modification pattern of synthetic guide RNAs for increased stability and efficient CRISPR-Cas9 gene editing avoiding cellular toxicity. PLoS One. 12, e0188593 (2017). doi: 10.1371/journal.pone.0188593
- Anderson, E.M., A. Haupt, et al. Systematic analysis of CRISPR-Cas9 mismatch tolerance reveals low levels of off-target activity. J. Biotechnol. 211, 56-65 (2015)
- He, K., E. Chou, et al. Conjugation and evaluation of triazole-linked single guide RNA for CRISPR-Cas9 gene editing. ChemBioChem. DOI: 10.1002/cbic.201600320 (2016)
