コストのかかる自動化を必要とせずに、数百または数千の遺伝子をスクリーニング
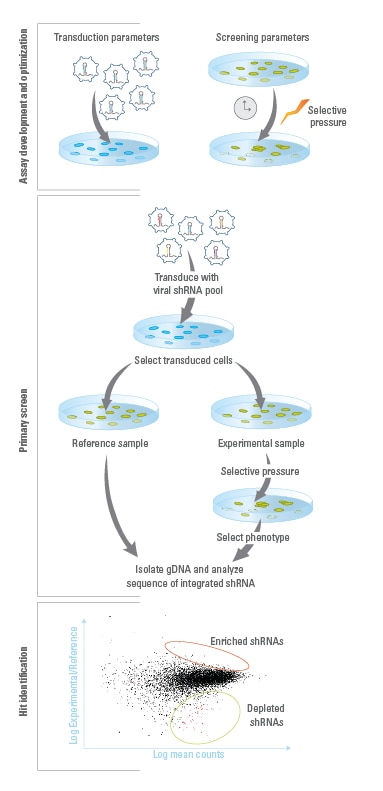
プール化レンチウイルススクリーニングを利用し、細胞応答とシグナル伝達経路を調節する遺伝子を特定したり、新しい遺伝子機能を発見できます。プール化スクリーニングライブラリーは、わずか50個のコンストラクトから最大1000個のコンストラクトまで構成することができます。アレイ化スクリーニング(個別にアレイ化された試薬を使用、リードアウトを自動化で測定)と比較すると、プール化スクリーニング用ライブラリーは、少数の細胞培養用ディッシュ内で形質導入し、細胞集団をスクリーニングすることができます。
高いfold-representationを維持するために必要な条件など、プール化レンチウイルススクリーニングを成功させるための重要なパラメーターの詳細については、次の出版物をダウンロードしてください: Ž. Strezoska, A. Licon, Optimized PCR Conditions and Increased shRNA Fold Representation Improve Reproducibility of Pooled shRNA Screens. PLoS One 7, e42341 (2012).
すべてのHorizonプール化レンチウイルスライブラリーには以下が含まれます:
- 精製され濃縮されたレンチウイルス粒子として提供される高品質のプール
- 遺伝子ファミリーまたは全ゲノムライブラリー
- カスタムコレクションライブラリーの提供可能。こちらからお見積りをご依頼ください。
- プール化レンチウイルススクリーニングを成功させるための実験計画用ワークシート、および検証済みのプロトコール
検証済みのプール化レンチウイルスのスクリーニングプロトコールで推奨しているHorizon製品:
- 最適化および検証済みのベクター特異的プライマーキット(以下の目的用に設計されています)
- バイアスを最小限に抑えた効率的なゲノムDNAのPCR増幅
- ヒット同定のためのハイスループットマルチプレックスシーケンシング
Learn more about Custom Pools
SMARTvector lentiviral shRNA、SMARTvector Inducible Lentiviral shRNA、shMIMIC Lentiviral microRNA、shMIMIC Inducible Lentiviral microRNA、またはヒト、マウス、またはラット用のGIPZコンストラクト(利用可能な場合)から選択できます。
- 利用可能なプールサイズ:50~10,000コンストラクト
- プールは、精製され濃縮されたレンチウイルス粒子として提供されます(構成ベクターの場合は5 x 108 TU/mL以上、誘導ベクターの場合は1 x 107 TU/mL以上)。
- レンチウイルス粒子の量を選択します(最小量100 μL)。
- ウイルス産生の前に、プールはNGS分析によってコンストラクトの回収と均一性について分析します。
- コンストラクト配列、RefSeq遺伝子アクセッション、Entrez遺伝子IDまたはmiRBase遺伝子IDを含むデータファイルを提供します。
検証済みのプール化レンチウイルススクリーニングプロトコールで推奨しているHorizon製品:
- トランスダクション最適化のためのnon-targetingコントロールレンチウイルス
- Vector-specific Indexing PCRおよびSequencing Primer Kitsには、最適化され、実験的に検証されたプライマーを含む
- バイアスを最小限に抑えた効率的なゲノムDNAのPCR増幅
- ヒット同定のためのハイスループットマルチプレックスシーケンシング
- プール化レンチウイルススクリーニングを成功させるための実験的にテストされたプロトコールと計画ツール
見積もりをリクエストする前に、次のマニュアルやツールをダウンロードして、プール化レンチウイルススクリーニングを慎重に計画し、必要なコンポーネントの量を計算することが非常に重要です。
重要なお知らせ
プール化レンチウイルススクリーニング用ライブラリーおよび試薬は、以下に示す封じ込め措置および適用される法律および規制が満たされている研究所での内部研究使用のみを目的としています(製品利用規約に規定されているとおり)。製品は、診断、治療、またはその他の商業目的で使用することはできません。また、ヒトに対してはいかなる目的であっても投与することはできず、動物に対しては治療目的で投与することはできません。レンチウイルス粒子として提供されるすべてのプール化レンチウイルススクリーニング用ライブラリーは、複製能力がなく、自己不活化(self-inactivating: SIN)であり、非病原性(感染性ヒト疾患を引き起こさない)です。
レンチウイルス粒子製品を購入する研究者は、レンチウイルスベクター粒子の取り扱いに関する特定のガイドラインについて、所属機関の健康およびバイオセーフティ担当者と相談する責任があります。さらに、各研究者は、複製能のないSINレンチウイルスベクターおよび複製欠損レンチウイルス粒子を使用した研究について、その地域の管轄区域および機関における必要な許可を取得する全責任を負います。