Horizonの遺伝子解析用標準サンプルの全エクソームシーケンス(WES)データがOncoSpan以外の製品についてもご利用可能となりました。高品質のNGSデータセット(500倍を超える非常に高いリード深度)を提供しており、解析したデータセットの偽陽性を検出し、真陽性の確認を可能にします。
このブログでは、VCFファイルで何が期待できるか、そしてこれがアプリケーションの信頼を構築するのにどのように役立つかを探ります。
エクソームシーケンスされたOncoSpan製品のデータは開発製品の分析にとって非常に価値があるというフィードバックを多くいただきました。このたび、より多くのgDNA、cfDNAフォーマット製品においてもWESデータの提供を開始しました。
シーケンスデータが提供される製品は?
| Catalog number | Description | Batch number sequenced | Source material |
|---|---|---|---|
| HD728 | Tru-Q1 | 40707 | gDNA |
| HD729 | Tru-Q2 | 40755 | gDNA |
| HD730 | Tru-Q3 | 40780 | gDNA |
| HD731 | Tru-Q4 | 40833 | gDNA |
| HD734 | Tru-Q7 | 40919 | gDNA |
| HD752 | Tru-Q0 | 43262 | gDNA |
| HD753 | Structural Multiplex gDNA | 41130 | gDNA |
| HD776 | Multiplex cfDNA 0% | 46125 | gDNA |
| HD777 | Multiplex cfDNA 5% | 46125 | gDNA |
| HD793 | BRCA Germline | 46024 | gDNA |
| HD795 | BRCA Somatic Multiplex I | 46500 | gDNA |
| HD802 | EGFR Quantitative Multiplex | 46026 | gDNA |
| HD829 | Myeloid gDNA | 47035 | gDNA |
Table 1. シーケンスデータが提供される製品(関連情報を含む)
製品はどのようにシーケンスされましたか?
すべてのサンプルは、米国ペンシルバニア州ピッツバーグのGenomics facilityでシーケンスされました。シーケンスワークフローで使用されたキットをTable 2に示します。
| Product type | Library generation kit | WES probe | Capture kit | Sequencer |
|---|---|---|---|---|
| gDNA | NOVA-5188-01-NEXTflex-Rapid-DNA-Seq-kit-2.0_v19.12_v5 | AGILENT SureSelect Clinical Research Exome v3 (CREV3) | NEXTFLEX™ Pre- & Post- Capture Combo Kit- Set A Manual (2613_0) | Illumina NovaSeq 6000 |
| cfDNA | NOVA-5150-01_NEXTFLEX_Cell_Free_DNA-Seq_Kit_19.02-002 | AGILENT SureSelect Clinical Research Exome v3 (CREV3) | NEXTFLEX™ Pre- & Post- Capture Combo Kit- Set A Manual (2613_0) | Illumina NovaSeq 6000 |
Table 2. シーケンスされた製品で使用されたNGSワークフローの詳細
製品ごとに1つのバッチがシーケンスされたため、すべてのバッチで内在性バリアントの結果が検証されているとは限りません。ただし、データのカバレッジが高い(> 500x)ため、結果には信頼性があります。
シーケンスされた製品についてどのような情報が提供されますか?
各製品には、2つのVCFファイル(GRCh37 / hg19またはGRCh38 / hg38)があり、次のバリアント情報を含みます。
- 染色体上の位置
- 遺伝子名
- バリアントのゲノム座標(GRCh37 / hg19またはGRCh38 / hg38)
- dbSNPID
- ヌクレオチド置換
- リード深度
VCFは大きく(200MB以上)、テキストベースのファイルで、ご使用のバイオインフォマティクス解析ソフトウェア(bedtools、VCFtoolsなど)を使用して開くことができます。または、7-Zipなどのツールを使用して解凍し、テキストエディター(Sublime Text、Notepad++ など)を使用して開くこともできます。
VCFファイルで見つかったものとは異なるバリアントが検出されました。何が、原因でしょうか?
生物学的および技術的なばらつきにより、同じ細胞株ブレンドを使用して製造された製品のわずかなフォーマットによる違い、またはバッチによる違いがあると推定されます。バリアントの検出は、使用するNGS法とバイオインフォマティクスパイプラインにも依存します。
データ分析のサポート
SNPとMNPの検出
多塩基多型(MNP)は、2つ以上のSNPが隣り合って発生することを特徴とし、これらのバリアントはMNPと呼ばれることもあります。
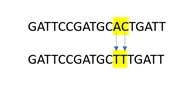
Horizonは、研究者がシーケンスでMNPをコールする可能性があることを理解しています。ただし、データ分析に使用されるソフトウェアは、各バリアントをSNPとしてコールしており、MNPバリアントとしてはリストしていません。連続したSNPの共起は確認していないため、各SNPのアレル頻度(AF)とそれらの同時発生によっては、MNPバリアントが存在する可能性があります。しかしながら、これらのデータでは、個々のSNP情報のゲノム座標とリード深度が利用可能であり、バリアント(SNPまたはMNP)の性質を推測するのに役立ちます。
真陽性と偽陰性の検出
Horizon製品のWESデータが利用できるため、真陽性と偽陰性の確認が容易になります。私共のWESデータは、他社が使用していると思われるターゲットパネルシーケンスと比較して包括的です。ddPCRによって検証されたバリアントのみ、確信を持ってコールすることができます(各製品ページで入手可能です)。
内在性バリアントの存在は、提供されたWESデータで確認できます。カバレッジの高いデータセットにより、バリアントの存在を確認する確信が得られます。ただし、検出されなかったバリアントが存在しないことを確認することはできません。使用されたアッセイデザインまたは分析方法による理由で、真の陰性または検出されなかった可能性を区別できないためです。
アッセイの種類、アッセイデザイン、およびお客様が使用するデータ分析方法に基づいて、コールされたバリアントまたはアレル頻度に違いが生じる可能性があります。 私共はイルミナNovaSeq 6000を使用しました。使用したキットとデータ分析情報は、エクソームシーケンスページで提供しています。
他のフォーマットのシーケンスデータを取得できますか?
Horizonが複数のフォーマットを販売している製品(例えばStructural multiplex ではgDNA(HD753)、cfDNA(HD786)、FFPE(HD789))では、意図的にgDNAフォーマットをシーケンスするフォーマットとして選択しました。これは、cfDNAおよびFFPE製品が超音波処理または固定によりアーチファクトを示す可能性があるためです。gDNAのシーケンスにより、最も信頼性の高いバリアントコールが保証されます。
cfDNA製品の選択
シーケンスされた2つのcfDNA製品は、HD780 Multiplex cfDNA setのものです。Claimされたバリアントを確信を持って検証できるように、5% cfDNA製品(HD777)を選択しました。野生型(WT)製品(HD776)のデータは、真陰性を検証するだけでなく、他の内在性バリアントに関する情報も提供します。
Written by Bernice Freeman & Jesse Stombaugh

 Jesse is a Senior Bioinformatics Scientist in the Bioinformatics team at Horizon Discovery, US. He obtained his PhD from Bowling Green State University, where he focused on RNA 3D structure prediction. At Horizon, he specializes in developing and scaling software solutions for processing and analyzing NGS data. These solutions are used for developing new products and assessing the quality of our current products.
Jesse is a Senior Bioinformatics Scientist in the Bioinformatics team at Horizon Discovery, US. He obtained his PhD from Bowling Green State University, where he focused on RNA 3D structure prediction. At Horizon, he specializes in developing and scaling software solutions for processing and analyzing NGS data. These solutions are used for developing new products and assessing the quality of our current products.
