オフターゲット効果の存在は、実験データの解釈を非常に複雑にする場合があります。Dharmacon™ ON-TARGETplus™ siRNAおよびCRISPRmod CRISPRiシステムは、オフターゲット効果を最小限に抑えて標的遺伝子をノックダウンするように設計されており、直交検証戦略のための強力なツールであり、特異性の高いリードアウトが得られます。
細胞への不自然な成分の導入によって引き起こされるオフターゲット効果は広く認識されています。ただし、それらは最小限に抑えることができ、真の表現型と区別するためのさまざまな戦略があります。
RNAiは、単一試薬を用いたアッセイからライブラリーによるスクリーニングまで幅広く使用されています。RNAi試薬は、何十年にもわたって私たちによって広範囲に特徴付けられ、最適化されてきました。先ごろ、Dharmacon製品ファミリーに、ゲノムDNAの特定の位置を標的とし、DNAを切断することなく転写をブロックする新しい試薬、CRISPRmod CRISPRi試薬が加わりました。これにより、mRNA発現レベルを制御して翻訳を抑制するRNAi試薬に加えて、標的遺伝子の転写をDNAレベルで特異的に抑制する新たな戦略が可能になりました。
私達は、複数の細胞発現の機構に関する直交検証戦略を推奨しています。特異的な遺伝子ノックダウン方法を組み合わせて使用すると、より深いヒット検証が可能となります。
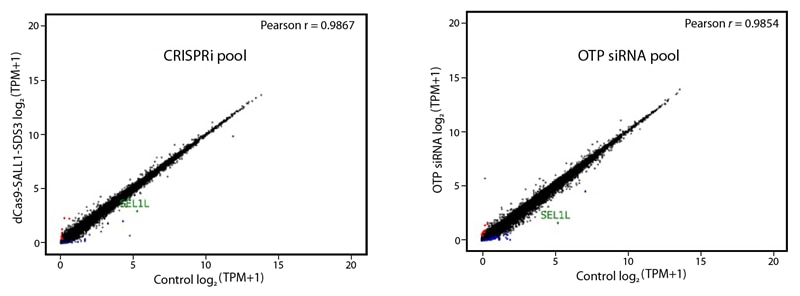
両方の技術の特異性を検討するために、USOS細胞のSEL1遺伝子を標的とし、新規のCRISPRmod CRISPRiおよびON-TARGETplus siRNA試薬を比較したオフターゲットシグネチャ遺伝子発現分析を実行しました。SEL1遺伝子をノックダウンした際の遺伝子発現レベル(log2 TPM、Transcripts Per Million)を測定し、non-targetingコントロールをトランスフェクションした際の発現レベルと比較しました。
両方の技術における作用機序の違いにもかかわらず、当社のCRISPRiおよびRNAi試薬の測定値は、両方の遺伝子発現シグネチャにおけるピアソンの相関係数に示されるように、非常に特異的であることが判明しました(Figure 1)。
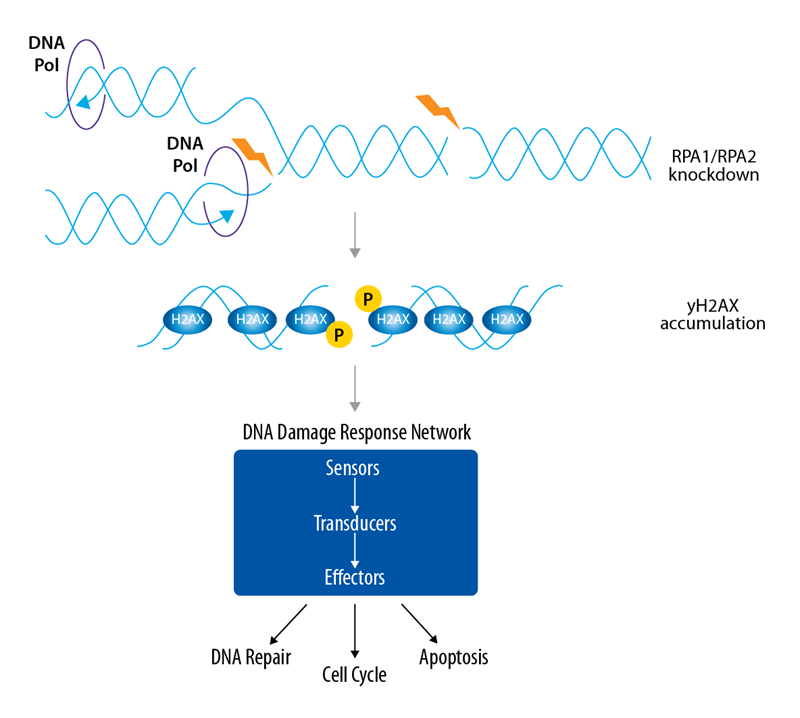
これに基づいて、両方の方法が直交検証ステップにも使用できることを示唆するコンパニオンDNA損傷応答アッセイを実行しました。Replication protein A(RPA)は、RPA1、RPA2、およびRPA3サブユニットで構成され、一本鎖DNAに対して高い結合親和性を持ち、DNA損傷応答における重要な調節機能を果たしていることが示されています1, 2(Figure 2)。
予想どおり、RPA1とRPA2のノックダウンは、リン酸化H2AXレベル(γ-h2AX)の増加を介して監視できる、修復されていない二本鎖DNA切断の蓄積をもたらしました。両方の遺伝子発現調節ツールは、それぞれの野生型コントロールで比較を行い、確認結果を示しました(Figure 3)。
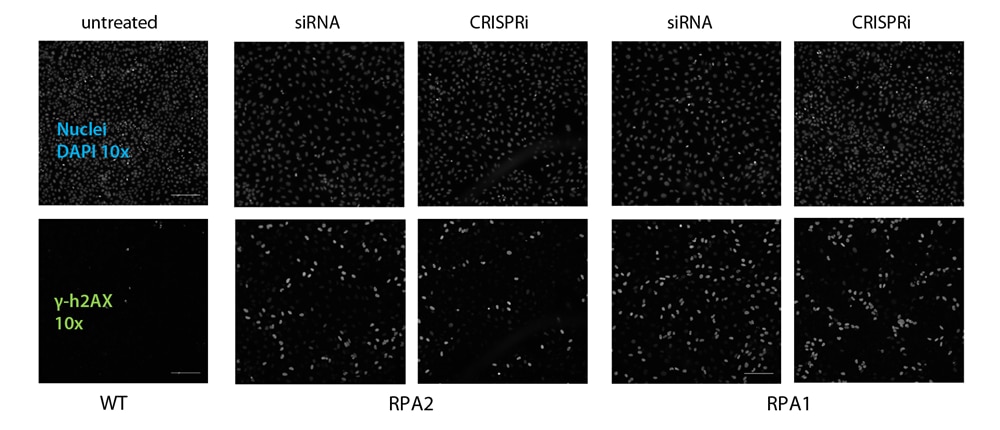
これらの結果は、標的遺伝子に対するDNA転写(CRISPRi)とmRNA翻訳(RNAi)の抑制による両方の遺伝子発現調節技術の特異性を裏付けています。最終的に、機能性タンパク質の発現は複数の角度からダウンレギュレートされ、信頼性の高いデータセットにつながります。
当社は、世界中で使用されているRNAi試薬に加えて、新しいCRISPRi試薬を幅広く提供しています。Horizonのテクニカルサポートは、ゲノム編集および遺伝子発現調節技術をサポートする訓練を受けたサイエンティストで構成されるチームです。プロジェクトについて、ぜひご相談ください。
CRISPRi resources
Welcome to the toolbox, CRISPRi - Blog article
新規dCas9融合タンパク質と化学合成ガイドRNAによるCRISPR媒介転写抑制 - Poster
CRISPRmod (CRISPRa & CRISPRi) - Brochure
RNAi resources
RNA干渉実験におけるオフターゲット効果の低減 – Blog article
RNAi 実験を成功させるための適切なコントロールの使用 – Blog article
siRNAスクリーニング:ヒット層別化戦略の開発 - Poster
オフターゲット効果: RNA interference (RNAi)によるノックダウンを妨げるものとは? – Application note
Dharmaconリーディングリスト(RNAi specificity & functionalityのセクションを参照)– Recommended Reading

