ウエスタンブロットによるノックアウト細胞株の検証の落とし穴を克服する
フレームシフト変異の後、タンパク質はどうなるのか?
フレームシフト変異は、しばしばノックアウトにつながると推定される、早すぎる停止コドンを生成する可能性があります。ガイドRNAが標的とする領域、DNAが細胞でどのように修復されるか、そして停止コドンの位置1に依って、タンパク質レベルでは複数の結果が考えられます:
- 翻訳前にナンセンス変異依存分解(nonsense-mediated decay: NMD)によってmRNAが分解されるため、タンパク質は生成されない(Figure 1b)
- 翻訳によって不完全なタンパク質が作られる(Figure 1c)
- 未成熟停止コドンの後の代替開始コドンから翻訳が始まり、不完全なタンパク質が作られる(Figure 1d)
- インデルを含むエクソンを除去するエクソンスキッピングにより、スプライスバリアントを生成する(Figure 1e)
- 野生型タンパク質の不完全部分と新規アミノ酸配列が翻訳される(Figure 1f)
これらの宿命のいくつかは、同じ細胞株で共存することがあります。例えば、標的エクソンがスキップされたmRNAが、標的エクソンを含むmRNAと同時に存在することがあります2 。
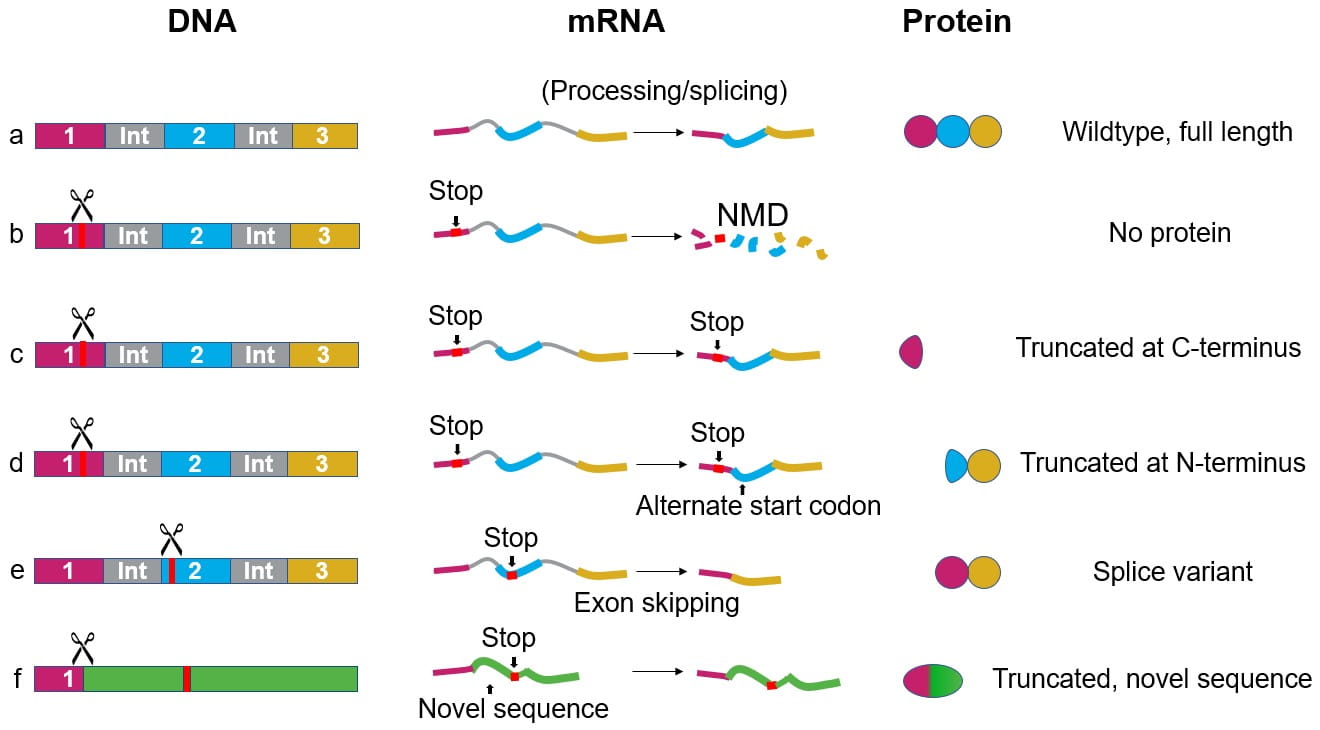 Figure 1. 野生型細胞株におけるDNA、mRNA、タンパク質(a)と、フレームシフト変異後に起こりうる結果(b-f)
Figure 1. 野生型細胞株におけるDNA、mRNA、タンパク質(a)と、フレームシフト変異後に起こりうる結果(b-f)
クローンのノックアウトをチェックする一般的な方法はウエスタンブロットです。ウエスタンブロットの結果は、抗体が結合する場所と、細胞株が発現するタンパク質によって異なります。Figure 2と3は、ウエスタンブロットがどのように見えるかの例を示しています。
ノックアウト細胞株を用いたウエスタンブロットでバンドが見えるのはなぜですか?
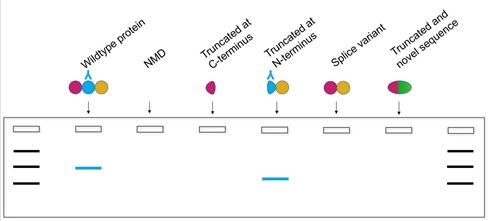
Figure 2のウエスタンブロットでは、野生型タンパク質のN末端にあるエピトープを認識する抗体を用いています。mRNAがNMDによって分解された場合、あるいはN末端を持たないスプライスバリアントが生じた場合、ブロットにバンドは観察されません。しかし、タンパク質がC末端で切断されていたり、野生型のN末端に新規のアミノ酸配列が続いていたりすると、バンドが観察されることがあります。このようなバンドは結果の解釈を誤らせる可能性があります。
 Figure 2. Figure 1に概説した各タンパク質について、野生型タンパク質のN末端に対する抗体で検出したタンパク質のバンドを示すウエスタンブロットの模式図
Figure 2. Figure 1に概説した各タンパク質について、野生型タンパク質のN末端に対する抗体で検出したタンパク質のバンドを示すウエスタンブロットの模式図
二番目の抗体、例えばタンパク質の中心にあるエピトープに結合する抗体(Figure 3)を用いると、より詳しい情報が得られます。両方のブロットで観察されたバンドを分析することで、野生型タンパク質がまだ存在しているかどうかについての追加的な洞察が得られますが、検出されたタンパク質が機能しているかどうかを確認することはできません。
したがって、下流の実験にとって重要な機能を評価するために、目的のタンパク質に特異的な機能アッセイを使用することを推奨します。以下に、当社のHAP1ノックアウト細胞株で研究者が使用した機能アッセイのリストを示します。このアッセイのリストは、どのノックアウト細胞株でもタンパク質の機能をチェックするのに適用できます。
ノックアウト細胞株はどのように検証すればよいのでしょうか?
問題の細胞株がHAP1ノックアウト細胞株であろうと、癌に関連したノックアウト細胞株であろうと、あるいは自分で作ったノックアウト細胞株であろうと、ノックアウトを検証するためのヒントは以下の通りです:
- 可能であれば、標的タンパク質の重要な機能を調べるために機能的アッセイを用いる。
- 全てのアッセイにおいて、細胞株特有の影響を考慮するため、親細胞株をコントロールとして用いる。
- 抗体を使用する場合は、ポジティブコントロールとネガティブコントロールを用い、抗体が正しい標的に結合していることを確認する。
- 全ての実験において、アッセイコントロールを含む適切なコントロールを考慮する。
References
-
Popp, M. W. and Maquat, L. E., Leveraging Rules of Nonsense-Mediated mRNA Decay for Genome Engineering and Personalized Medicine. Cell 165 (2016). https://doi.org/10.1016/j.cell.2016.05.053
-
Tuladhar, R., Yeu, Y., Tyler Piazza, J. et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun 10, 4056 (2019). https://doi.org/10.1038/s41467-019-12028-5
関連する他のリソース
• Antibody validation: Why is antibody validation important?
• Blog: Is your antibody binding the right target?
• Blog: Spying on proteins - how observation can affect the perception of reality
• Blog: Strategies to detect and validate your CRISPR gene edit
• Video: What happens to the protein in a knockout cell line?
