- ゲノム編集
- Pin-point™ 試薬
- Pin-point™化学合成sgRNA検証済みコントロール
Pin-point™化学合成sgRNA検証済みコントロール
| Pin-point化学合成sgRNAは、crRNA、tracrRNA、および独自のアプタマー配列からなる128塩基キメラ融合体です。分子の5'末端と3'末端の両方がヌクレアーゼ耐性を持つように化学修飾されています。
sgRNAコントロールは、Pin-point nCas9およびPin-point Rat APOBEC mRNAを用いた塩基編集実験のポジティブコントロールとして推奨されます。 遺伝子特異的ポジティブコントロールおよびキットは、塩基編集および表現型タンパク質ノックアウトの評価用に設計および検証されています。以下のように構成されています:
|
128ntのPin-point sgRNAはこちらからお申し込みいただけます。
|
Pin-pointプラットフォームによる塩基編集は、DNAの二本鎖切断やインデル(indel)を形成することなく、特異的なヌクレオチド変化を誘導します。
このシステムは3つのコンポーネントから構成されています:
[1] DNAの一本鎖のみを切断または「ニッキング」するウラシルグリコシラーゼ(UGI)阻害剤と融合したヌクレアーゼ欠損「ニッカーゼ」nCas9(Komor, 2016)
[2] アプタマー結合タンパク質と融合したシチジンデアミナーゼ塩基編集酵素(Rat APOBEC)。シチジンデアミナーゼ酵素は、塩基対が編集ウィンドウ(プロトスペーサーのPAM遠位端の4から8位)内にある場合、C-G塩基対をT-A塩基対に変換します(Collantes, 2021)。
[3] nCas9とアプタマー:デアミナーゼ融合体を特定のDNA標的部位に誘導するアプタマー性シングルガイドRNA(sgRNA)。
哺乳類細胞に3つの構成要素すべてを導入することで、標的部位のC-GからT-Aへの塩基変換が誘導されます。このシステムは、遺伝子ノックアウト(未成熟終止コドンの導入(Billon, 2017)、スプライスドナーおよびスプライスアクセプター部位の破壊(Webber, 2019))、または点突然変異の導入に使用できます。
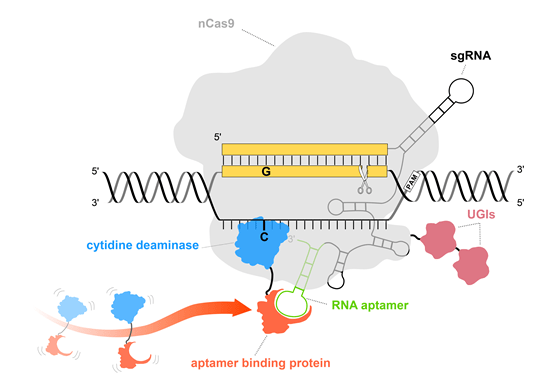
Pin-point塩基編集システムの概要図
nCas9(薄い灰色)を利用することで、DNAの二本鎖切断が起こらず、DNA損傷応答経路がトリガーされません。Pin-point sgRNAにはアプタマー(緑)が含まれており、アプタマー結合タンパク質(オレンジ)を介してデアミナーゼ(青)をリクルートし、塩基編集を行います。
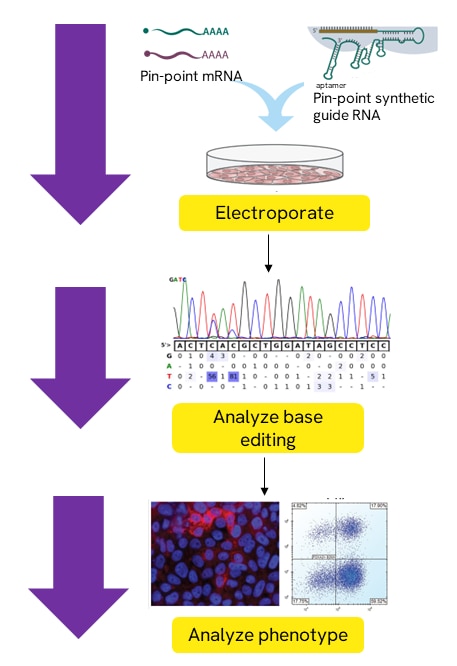
Pin-point nCas9 mRNA、Pin-point Rat APOBEC mRNA、Pin-point synthetic sgRNAを用いた塩基編集
3つのコンポーネントすべてを目的の特定の細胞にエレクトロポレーションします。その後、目的の塩基編集領域を増幅し配列決定するための解析プライマーを用いて塩基編集を観察することができます。表現型を解析し、標的タンパク質のノックアウトを評価することができます。
Table 1 Pin-point mRNA必要量
| ウェル/フォーマットサイズごとの反応数* | ||||
|---|---|---|---|---|
| HEK 293T細胞(96ウェルプレートで50,000細胞/ウェル) | T細胞(24ウェルプレートで250,000細胞/ウェル) | |||
| Pin-point nCas9 mRNA | Pin-point Rat APOBEC mRNA | Pin-point nCas9 mRNA | Pin-point Rat APOBEC mRNA | |
| 1 µg/well | 0.1 µg/well | 1.56 µg/well | 0.22 µg/well | |
| 20 µg | 20 | 200 | 12 | 90 |
| 100 µg | 100 | 1000 | 64 | 450 |
| 500 µg | 500 | 5000 | 320 | 2252 |
Table 2 Pin-point sgRNA必要量
| ウェル/フォーマットサイズごとの反応数* | ||||
|---|---|---|---|---|
| HEK 293T細胞(96ウェルプレートで50,000細胞/ウェル) | T細胞(24ウェルプレートで250,000細胞/ウェル) | |||
| 60 pmol/well | Singleplex 20 pmol/well or Multiplex (3 guides) at 20 pmol/well each |
|||
| 2 nmol | 33 | 100 | ||
| 5 nmol | 83 | 250 | ||
| 10 nmol | 166 | 500 | ||
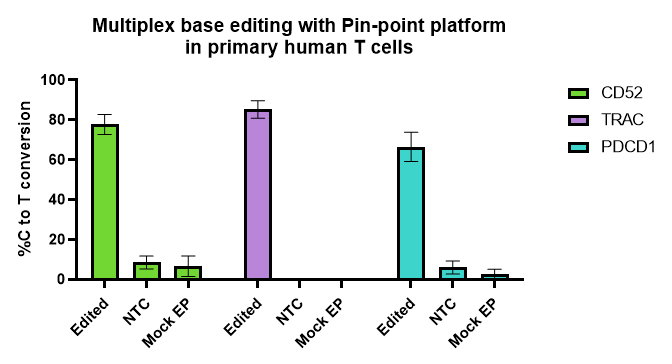
初代ヒトT細胞におけるマルチプレックス塩基編集
Pin-point塩基編集プラットフォームは、多くの実験やT細胞ドナーにおいて効率的で再現性が良好です。活性化T細胞に、Pin-point mRNAと3つの標的(CD52、PDCD1、TRAC)に対するPin-point化学合成sgRNA、またはnon-targetingコントロール(NTC #1)sgRNAをエレクトロポレーションしました。Mockエレクトロポレーション(EP)細胞をネガティブコントロールとして使用しました。エレクトロポレーション後6~7日目に細胞を回収し、Pin-point解析用プライマーを用いてPCR産物のサンガー配列決定データから塩基編集レベルを算出しました。
T細胞ドナー(N = 8)、5つの独立した実験、二重または三重のテクニカルレプリケート。ヒゲは平均値 ± SD
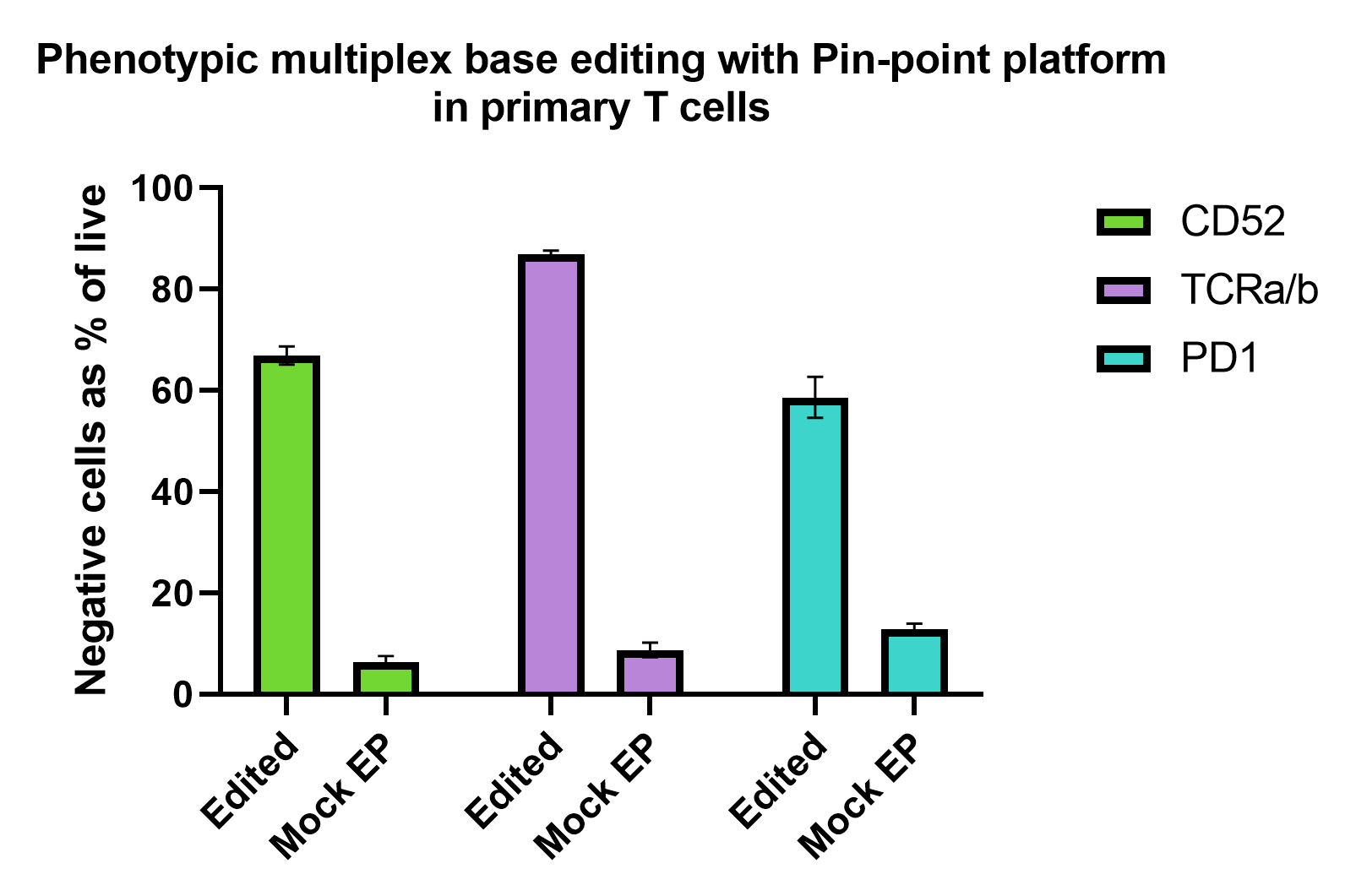
初代ヒトT細胞におけるマルチプレックス塩基編集の表現型解析
Pin-point塩基編集プラットフォームは、初代T細胞を効率的に多重遺伝子ノックアウトします。活性化T細胞に、3つの標的(CD52、PDCD1、TRAC)に対するPin-point mRNAおよびPin-point化学合成sgRNAをエレクトロポレーションし、7日後にフローサイトメトリーで解析しました。PD1の発現を誘導するために、T細胞は48時間前にPMA(phorbol 12- myristate 13-acetate)とイオノマイシンの存在下で培養しました。CD52とTCRa/bの発現は非刺激細胞を用いて分析しました。図は、編集サンプルおよびモックエレクトロポレーション(EP)サンプルの、生細胞における各マーカー陰性細胞の割合示しています。
T細胞ドナー(N = 2)の二重または三重のテクニカルレプリケート。ヒゲは平均値 ± SD
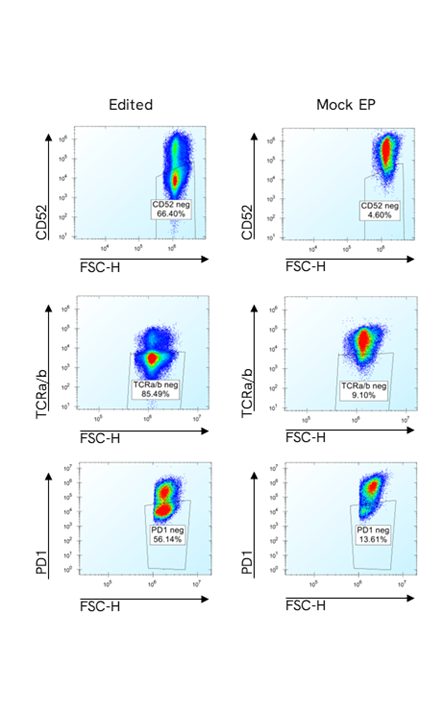
初代ヒトT細胞におけるマルチプレックス塩基編集のフローサイトメトリープロット
編集サンプルとコントロールサンプルの、3つの各マーカーの代表的フローサイトメトリープロット。各マーカーの陰性細胞のゲーティングは生細胞で行われています。

塩基編集によるトリプルノックアウトの編集効率
サンプル中の3つのマーカー(CD52、PD1、TCRa/b)が同時に陰性である細胞の割合。Pin-pointプラットフォームで編集したサンプル、およびモックエレクトロポレーション(EP)コントロールで、追加濃縮を行わず、生細胞における割合として示されています。PD1の発現を誘導するため、3つのマーカーを同時に定量できるように、分析前にT細胞をPMA(phorbol 12- myristate 13-acetate)とイオノマイシンの存在下で48時間培養しました。
T細胞ドナー(N = 2)、二重または三重のテクニカルレプリケート。ヒゲは平均値 ± SD
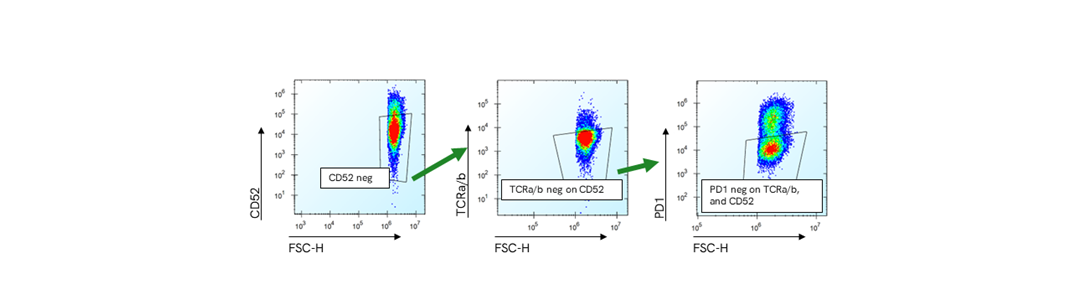
トリプルノックアウト塩基編集のフローサイトメトリーゲーティング戦略
編集サンプルの代表的フローサイトメトリープロットでゲーティング戦略を示しています。左より、生細胞でゲーティングされたCD52陰性集団、CD52陰性集団でゲーティングされたTCRa/b陰性集団、CD52とTCRa/b二重陰性集団でゲーティングされたPD1陰性集団。右端のゲートの細胞数は、3つのマーカーすべてが陰性細胞となります。トリプル陰性の細胞数は生細胞数で正規化し、上図にその割合を示しています。
Posters
-
Performance and modularity of Revvity's Pin-point base editing system characterized by arrayed and pooled screening platforms - poster
-
Pin-point™ base editing system: a versatile editing platform driving cell therapies - poster
-
The Pin-point™ base editing platform streamlines the generation of hypoimmunogenic iPSCs for allogeneic cell therapy - poster
Quick protocols
Safety data sheets
Related Products
Pin-point™塩基編集mRNAシステムは、二本鎖切断を誘発することなく、また相同性組換え修復を必要とすることなく、正確に指向性のある点突然変異の編集を可能にします。
Pin-point塩基編集を評価するためのNon-targeting化学合成コントロール。バイオインフォマティクスに基づき、ヒトゲノムのどの遺伝子も標的としないように設計されています。
