多様な抽出プラットフォームと適合性のある合成マトリックスCell Free DNAは、標準サンプルの選択の幅を広げます。
リキッドバイオプシーとは?

がんの診断、臓器移植後のモニタリング、出生前の遺伝子検査では、「早期発見が極めて重要な役割を果たす」と考えられています。非侵襲的であるリキッドバイオプシーの技術はここ数年間で大きく進歩し、侵襲的検査に代わるバイオマーカーの検査法として注目されています。リキッドバイオプシーではさまざまな種類の体液検体を使用できますが、がんの診断に於いて最も一般的に用いられるのは血液です。低侵襲で簡単にできる採血により、患者の負担を減らしたがんの早期発見や治療後のモニタリングが可能になります。 血液には、腫瘍を含む各組織から細胞またはDNAを含む細胞内容物が放出され存在しますが、がん患者の血液から得られるセルフリーDNA(cfDNA)の解析は、固形腫瘍の診断やモニタリングの判定方法として開発の勢いを増しており、いくつかのNGSベースのコンパニオン診断薬がFDAで承認されています(Guardant360 Dx、FoundationOne Liquid CDxなど)。
cell-free DNAの分離と精度管理
正常細胞と腫瘍細胞の両方から断片化されたDNA(cfDNA)が血液中に放出されますが、腫瘍特異的なcfDNAは非常に低濃度です。この低濃度と小さなフラグメントサイズは、標準的なゲノムDNA分離技術がcfDNA分離に適用できないことを意味します。したがって、適切なcfDNA分離プロセスを選択することはcfDNAの純度と収量を決定する上で重要であり、下流の解析プロセスにも影響を与えます。cfDNA抽出後のワークフローの妥当性は、解析法の感度と特異性に依存します。
cfDNAの抽出および解析ワークフローの至適化や検証用に、Horizon Discoveryは、ヒト血漿に近い合成マトリックスまたはバッファーに溶解されたcfDNA標準サンプルを提供しています。これらはヒト細胞株由来の標準サンプルであり、臨床検体に近い条件で解析できます。標準サンプルに含まれる変異やアレル比は検証されており、cfDNAの定性解析または定量解析の解析系の開発や検証、および日常的な精度管理に役立ちます。
血漿からcfDNAを分離するために、さまざまな抽出法が開発されています。主なものには、シリカベースの精製カラム法と磁気ビーズ法があります。複数の研究において様々な抽出方法の有効性が分析されており、回収されたcfDNAの収量とフラグメントサイズにばらつきがあることが示されています(参考文献1-4)。DNA抽出プロセスに影響を与える可能性のある他の要因は、pH、温度、イオン強度などの物理的条件です。cfDNA抽出物中の高分子量ゲノムDNA、タンパク質、金属イオン、およびその他の一般的な不純物などの存在も、下流の解析プロセスに悪影響を与える可能性があります。
異なる性能をもつ様々な抽出キットに対応するために、Horizon Discoveryは、一般的に広く使用されているいくつかの抽出キットと適合性のある、新しい合成マトリックスcfDNA標準サンプルを開発しました。この新製品の発売により、Horizon Discoveryは、現在、2種類の合成マトリックスMultiplex I cfDNA標準サンプルを提供しています。 Multiplex I cfDNA in Synthetic Matrix I(HD817)とMultiplex I cfDNA in Synthetic Matrix II(HD917)です。これらの製品は、人工的に作られたマトリックス製剤の組成が異なるため、それぞれの抽出キットとの適合性のレベルが異なります(図1)。2種類の合成マトリックス標準サンプルにより、使用する抽出キットに合わせた適切な標準サンプルを選択いただけるようになりました。
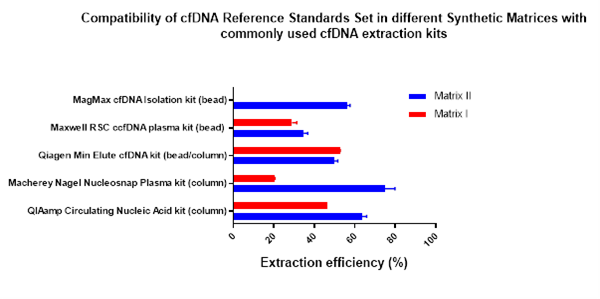
標準サンプルの解析評価
cfDNAによるがんの早期発見と移植拒絶反応のモニタリングにおける重要な課題の1つは、患者の血液中の変異DNAまたはドナーDNAの存在比が非常に低いことです。cfDNAは断片化されており血中濃度が低く、また変異のアレル比も低いため、変異の正確な検出が難しく、臨床的有用性が限られる可能性があります。したがって、cfDNAを正確に解析するためには、変異の特性が明らかにされている適切な標準品を使用する必要があります。マトリックスIおよびIIのcfDNA標準サンプルは、どちらも5%、1%、0.1%の変異アレル比および、野生型(アレル比0%)のセットで提供しています。
cfDNA標準サンプルの主要な変異は、遺伝子組成の正確度と一貫性を確保するために、細胞混合段階から、断片化されたcfDNAが人工血漿から回収される最終段階まで、製造プロセスの複数の段階で検証されます。デジタルPCRによる解析で、HD917から抽出したcfDNAの変異とアレル比が、高い再現性で確認できました(図2)。注:図2に示されているデータはHD917のみを使用していますが、いずれの合成マトリックスcfDNA(HD817を含む)でも同様のデータが得られると考えられます。
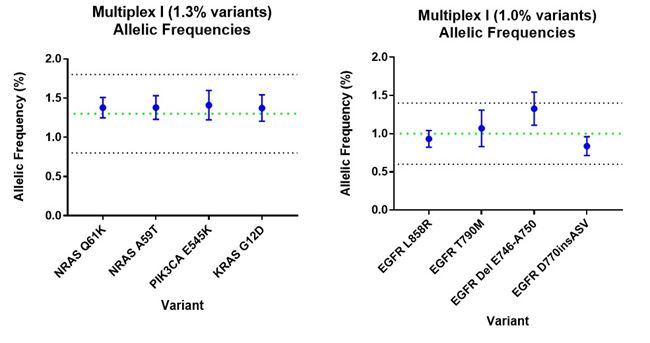
標準サンプルを作製するために使用された細胞ブレンドの全エクソームシーケンス解析でも“検証済み変異”とされた変異が正確に検出できることが確認され、複数の診断用試験でその有用性が証明されました。製品のバリデーションにおいては、変異の検証に加えて、DNAのフラグメント長(平均170 bp)とDNA濃度も実施されます。
サマリー
Horizonの標準サンプルは細胞株由来であるため、ヒトゲノムの複雑さに近い性状で安定した品質の製品を安定して提供します。合成マトリックスを使用して構築されたcfDNA標準サンプルは、ロット間のばらつきに悩まされ特別な保管を必要とするヒト血漿に代わり、費用対効果の高い標準サンプルです。また、複数のcfDNA抽出キットとの適合性がある2つの異なる合成マトリックス製品が選択できるHorizonのcfDNA標準サンプルは、リキッドバイオプシーアッセイの全行程で信頼性を担保するための優れたアッセイコントロールと言えます。
Written by Prabha Nagarajan
Prabhaは、Horizon Discovery(英国)の診断チームの上級研究員です。彼女はロチェスター大学で博士号を取得し、ミトコンドリアDNA修復メカニズムを研究しました。Horizon Discovery入社以前は、インドのStrand LifeSciencesの臨床診断チームに所属していました。Horizonにおける彼女の現在の役割には、腫瘍学およびその他の遺伝子プロファイリングテストで使用するための診断用標準サンプルの開発が含まれます。
Interested in our liquid biopsy controls?
Want to learn more?
Using reference standards in the development of new liquid biopsy platforms - blog
cfDNA合成マトリックス標準サンプルによるリキッドバイオプシーの課題への挑戦 - blog
Questions? Feel free to reach out to our support team.
References
- Fong SL, Zhang JT, Lim CK, et al. Comparison of 7 methods for extracting cell-free DNA from serum samples of colorectal cancer patients. Clin Chem 2009;55:587-9. doi: 10.1373/clinchem.2008.110122
- Pérez-Barrios C, Nieto-Alcolado I, Torrente M, Jiménez-Sánchez C, Calvo V, Gutierrez-Sanz L, Palka M, Donoso-Navarro E, Provencio M, Romero A. Comparison of methods for circulating cell-free DNA isolation using blood from cancer patients: impact on biomarker testing. Transl Lung Cancer Res 2016;5(6):665-672. doi: 10.21037/ tlcr.2016.12.03
- Repiská G, Sedláčková T, Szemes T, et al. Selection of the optimal manual method of cell free fetal DNA isolation from maternal plasma. Clin Chem Lab Med 2013;51:1185-9.doi: 10.1515/cclm-2012-0418
- Holmberg RC, Gindlesperger A, Stokes T, et al. Akonni TruTip(®) and Qiagen(®) methods for extraction of fetal circulating DNA--evaluation by real-time and digital PCR doi: 10.1371/journal.pone.0073068

