Edit-R デザイン済みsynthetic sgRNA(化学合成sgRNA)
Synthetic single guide RNAs for efficient gene knockout & unparalleled specificity
化学合成シングルガイドRNAは、効率的な遺伝子ノックアウトと高い特異性を提供します。
- 目的のターゲット遺伝子の編集(DNA切断)が保証されています。
- 機能的タンパク質ノックアウトの可能性をアルゴリズムで最大化し、オフターゲット編集を最小化するようにデザインされています。
- トランスフェクションに対応した化学合成RNAにより、クローニングとin vitro転写ステップが不要です。
- ヌクレアーゼ耐性を改善するために、化学修飾が導入されています。
- New! Edit-R human sgRNA designs have been updated to the latest RefSeq as of August 22, 2025.

Edit-R デザイン済みsynthetic sgRNA(化学合成sgRNA)
1Start Here
2Choose
化学合成シングルガイドRNAは、効率的な遺伝子ノックアウトと高い特異性を提供します。
Edit-Rのデザイン済み化学合成sgRNAは、S. pyogenes由来Cas9ヌクレアーゼを用いたゲノム編集に用いられ、目的のゲノム領域においてCas9を介したDNA二本鎖切断を起こす化学合成ガイドRNAです。すべてのEdit-R ガイドRNAは、挿入または欠失を起こすだけでなく、機能的なタンパク質ノックアウトを効率的に生成するようにアルゴリズムによりデザインされています。さらに、すべてのEdit-R 化学合成ガイドRNA製品には化学修飾が導入されおり、ヌクレアーゼによる分解が減少するため、全体的なゲノム編集性能が向上しています。
このような他に類を見ない正確性と効率により、Edit-Rのデザイン済み化学合成sgRNAは、パスウェイ解析や化学合成sgRNAライブラリースクリーニングからのヒットのフォローアップに理想的なツールとなっています。
New! Edit-R human sgRNA designs have been updated to the latest RefSeq in 2025 providing the most specific and genomically relevant guides for producing efficient protein knockout. This allows the Edit-R algorithm to target the latest genome annotations more accurately and efficiently providing you with the best solution for your research needs. Please reach out to Scientific Support if you have any questions.
すべてのガイドRNAデザインは、各遺伝子についてアルゴリズムで選択した最上位のものです。機能性と特異性の定性的なランク付けにより、特定のアプリケーションに合わせて最適なヒトガイドRNAを選択できます。機能性スコアは、このガイドがどの程度機能的ノックアウトをもたらす可能性があるかを予測したものです。特異性スコアは、潜在的なオフターゲット部位での切断活性の予測リスクに基づいています。詳細については、Edit-RガイドRNAのアルゴリズムページをご覧ください。
化学合成sgRNAを用いたCRISPRゲノム編集実験のためのコンポーネント
- 目的の遺伝子を標的とするように設計されたEdit-R化学合成sgRNA:機能的ノックダウンの可能性を高めるために、個別のガイドRNA、または3つのガイドRNAがセットになったsets of 3から選択してください。
- Cas9ヌクレアーゼ(タンパク質、mRNA、発現用プラスミド、発現用レンチウイルスベクター)、またはCas9安定発現細胞株から選択してください。
- 適切なポジティブコントロール、およびnon-targetingコントロール
- ガイドRNAを送達するためのDharmaFECT transfection試薬またはエレクトロポレーション
大規模な実験には:ヒトゲノムおよび遺伝子ファミリーを網羅するスクリーニングのための、デザイン済み化学合成sgRNAライブラリーのアレイ化コレクションをご選択いただくか、またはCherry-Pickライブラリーツールを使用して独自のカスタムミニライブラリーをデザインすることができます。
Edit-RガイドRNA保証
すべてのデザイン済みEdit-RガイドRNAは、Edit-Rテクニカルマニュアルの記載に従い細胞導入した場合に、標的領域で編集(DNA切断)ができることを保証します。
Edit-RガイドRNA保証は、野生型S. pyogenes由来Cas9ヌクレアーゼ(タンパク質、mRNA、発現用プラスミド、または発現用レンチウイルスベクター)とともに使用した場合に適用されます。
T7EIまたはSurveyorミスマッチ検出アッセイを使用して、試薬で処理された細胞集団の編集(DNA切断)の分析結果を提示する必要があります。同時並行で適切に実施されたEdit-R ポジティブコントロールが編集(DNA切断)に成功する一方で、Edit-Rデザイン済みガイドRNAによる編集(DNA切断)が成功しない場合、同じフォーマットで同じ容量の、異なるEdit-Rデザイン済みガイドRNAの交換製品が、1回限り無償で提供されます。
交換製品の提供は、テクニカルサポートチームとの話し合いでのみ承認されます。
DNAレベルでの編集(DNA切断)の成功は、常に機能的な遺伝子ノックアウトにつながるとは限りません。複数のガイドRNAをテストして、標的遺伝子のノックアウトに最も効果的なガイドRNAを決定することをお薦めします。
この保証は付随する実験費用には適用されず、CRISPR Design Toolを介して注文されたガイドRNAには適用されず、また本保証により交換したガイドRNAには適用されません。
Gene knockout workflow using Cas9 nuclease protein and synthetic sgRNA or crRNA:tracrRNA
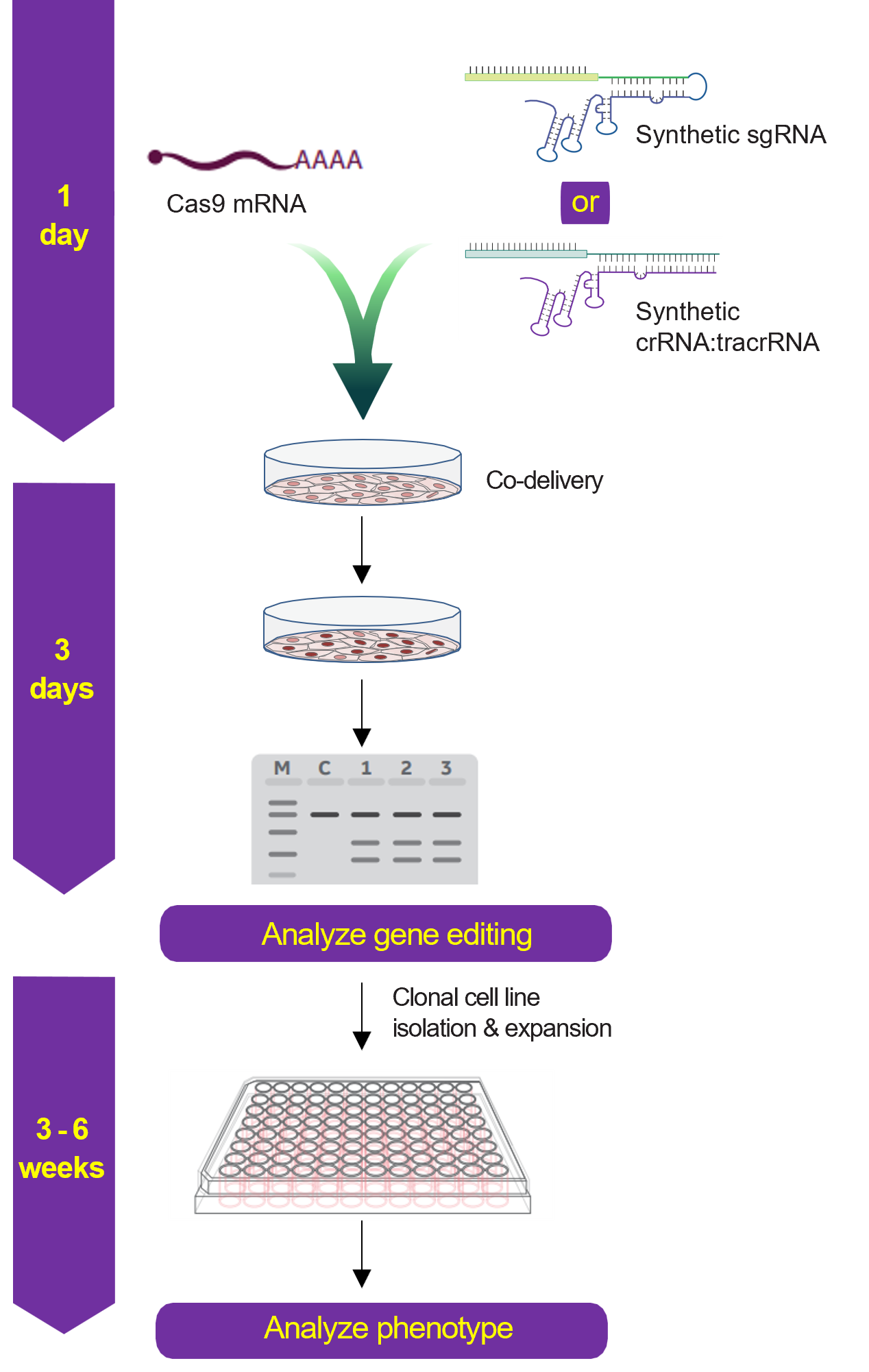
Gene knockout workflow using Cas9 nuclease protein and synthetic sgRNA or crRNA:tracrRNA

Gene knockout workflow using lentiviral Cas9 expression particles and synthetic sgRNA or crRNA:tracrRNA

以下のサポート試薬をご注文に追加して、ノックアウト実験を完成させてください。
Cas9ヌクレアーゼオプション
- Cas9 mRNA またはCas9 protein NLS:Edit-R化学合成sgRNAと共導入することで、完全なDNA-freeのワークフローを実現できます。
- Cas9安定発現細胞株:transfect-readyの細胞株であり、ただちにEdit-R化学合成sgRNAをトランスフェクトし、遺伝子ノックアウト実験を行うことができます。
化学合成ガイドRNAコントロール
- Non-targetingコントロール:標的遺伝子特異的sgRNAの非存在下でCRISPR-Cas9コンポーネントに対するベースラインの細胞応答を評価するために使用します。ヒトまたはマウスゲノムのどの遺伝子も標的としないことが保証された5つのスクランブル配列からお選びいただけます。
-
Cutting controls
Cutting control (safe harbor) synthetic sgRNAs recommended for determination of baseline cellular responses in CRISPR-Cas9 experiments.
- ポジティブコントロール:十分に特徴付けられた遺伝子を標的としており、ゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。PPIBまたはDNMT3Bからお選びいただけます。
トランスフェクション試薬
- DharmaFECTトランスフェクション試薬:送達の改善と毒性の低減のために最適化されています。実験に最適なDharmaFECT試薬は、お使いのCas9ヌクレアーゼと細胞タイプによって異なります。
| Cas9ヌクレアーゼ | 推奨されるトランスフェクション試薬 |
|---|---|
| Cas9安定発現細胞株、またはCas9ヌクレアーゼ・レンティウイルス粒子 |
DharmaFECT 1、2、3、4:化学合成ガイドRNAのトランスフェクションにご使用ください。この選択ガイドを使用して、お客様の細胞タイプに推奨されるDharmaFECT試薬をお探しください。 |
| Cas9 mRNAまたはprotein NLS |
DharmaFECT Duo:mRNAまたはタンパク質と化学合成ガイドRNAの共導入用 |
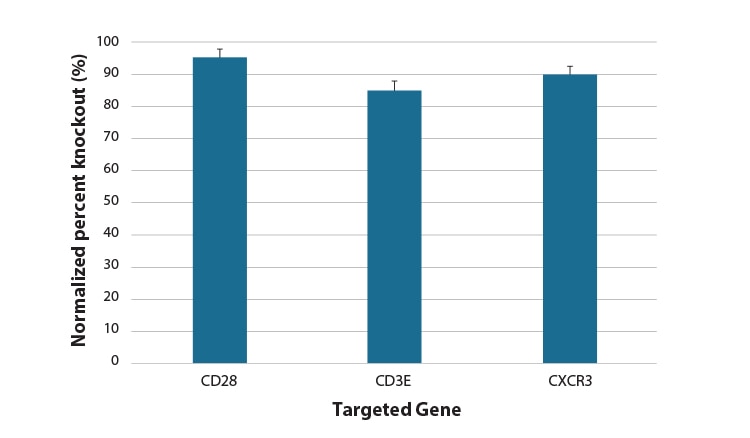
CD4+T細胞に、標的遺伝子あたり単一のデザイン済み化学合成sgRNAを用いた、タンパク質ノックアウトのフローサイトメトリー分析
 |
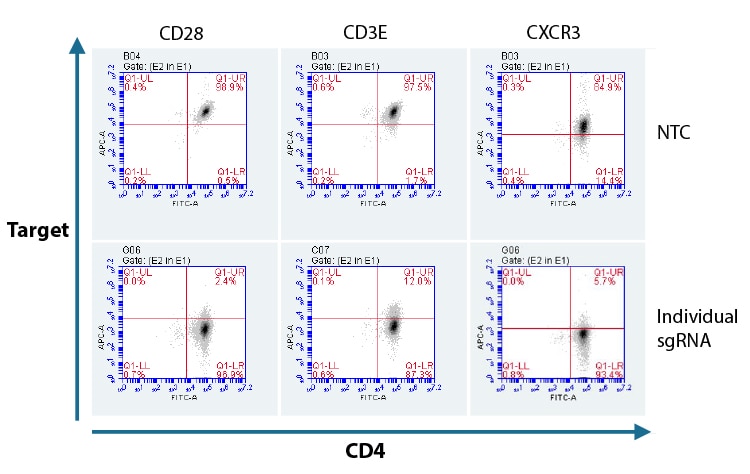 |
Lonza 96-well Shuttleシステムにより、初代ヒトCD4 + T細胞に、標的遺伝子ごとに設計した個別のデザイン済み化学合成sgRNAまたはNon-targetingコントロール(NTC)とCas9からなるRNPをヌクレオフェクションしました。72時間後、FACS分析により、CD28、CD3E、およびCXCR3の機能的ノックアウトを、標的遺伝子を発現していない細胞の割合として評価しました。細胞のCD4発現をAlexa Fluor 488標識抗体で染色して正規化し、APC標識一次抗体を使用してターゲットであるCD28、CD3E、およびCXCR3を比較しました。
化学合成sgRNAの混合物(プール)によるFUS遺伝子の機能的ノックアウト
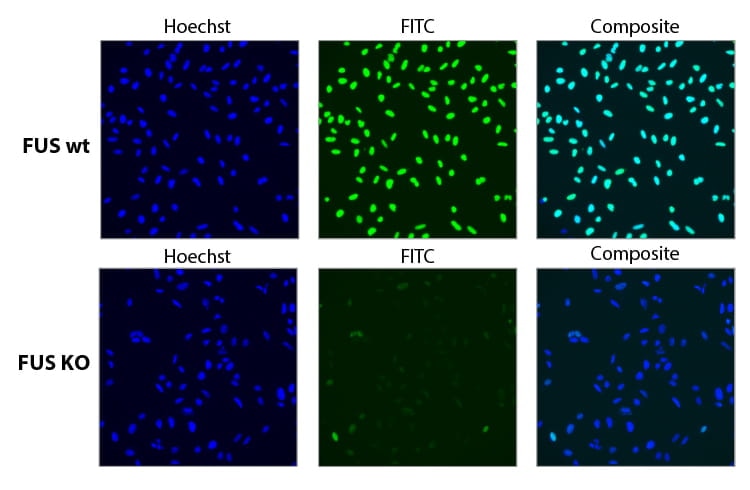
FUSの機能的ノックアウトを、CAGプロモーター下でCas9を恒常的に発現するU2OS細胞で評価しました。0.04 µL DharmaFECT 4を用いて、FUSをターゲットとする3つの異なるEdit-R デザイン済みsgRNAプール(25 nM)を細胞にトランスフェクトしました。トランスフェクションから96時間後に細胞固定後、FUSを標的とする一次抗体およびAlexa Fluor 488蛍光標識二次抗体で染色しました。核識別のためにHoechst染色を使用しました。
Nuclease stabilization modifications improve gene editing efficiency
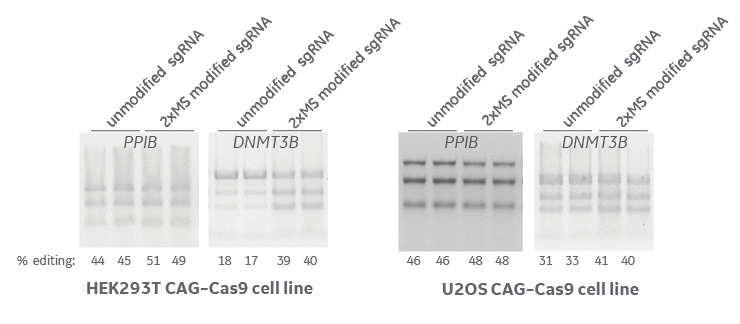
HEK293T and U2OS integrated Cas9 (under the CAG promoter) cell lines were transfected with unmodified and 5’ and 3’ 2xMS modified synthetic sgRNA targeting PPIB and DNMT3B genes. At 72 hours editing efficiency was assessed with a mismatch detection assay, T7 Endonuclease I (T7EI). In all cases the stabilizing modifications improve gene editing efficiency. MS = 2’-O-methyl nucleotides and phosphorothioate backbone linkage.
Edit-R synthetic guide RNAs cause virtually no innate immune response or toxicity compared to in vitro transcribed guide RNA

A HEK293T Cas9 nuclease expressing cell line was transfected with different synthetic guide RNA formats including unmodified crRNA:tracrRNA, crRNA:tracrRNA modified with 2xMS on 5’ crRNA and 3’ tracrRNA, crRNA:tracrRNA modified with 3xMS on both 5’ and 3’ crRNA and tracrRNA, unmodified synthetic sgRNA, modified synthetic sgRNA with 5’ and 3’ 2xMS or 3xMS, and in vitro transcribed (IVT) sgRNA targeting PPIB and DNMT3B genes. At 72 hours viability was assessed with the Resazurin reduction assay (red dots) and the levels of five immune response genes were quantified by RT-qPCR. MS = 2’-O-methyl nucleotides and phosphorothioate backbone linkages.
Structure of modifications for improved nuclease resistance on Edit-R synthetic sgRNA
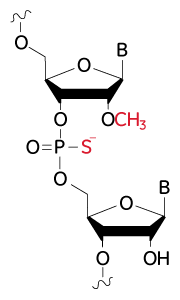
Modifications for improved nuclease resistance: 2’-O-methyl modified nucleotides and phosphorothioate backbone linkages.
Sequence structure of Edit-R synthetic single guide RNA
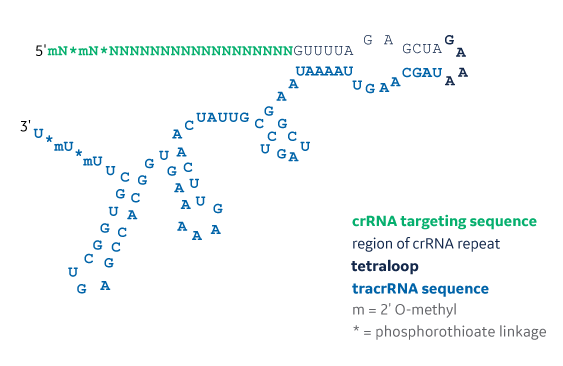
The structure of a Edit-R synthetic single guide RNA showing the 2xMS nuclease stability modifications on both 5’ and 3’ ends of the molecule. The structure contains a 20 nt targeting sequence (shown in green as poly Ns), 12 nt crRNA repeat sequence (shown in light gray), 4 nt tetraloop sequence (shown in bold black), and a 64 nt tracrRNA sequence (shown in blue). MS = 2’O-methyl nucleotides and phosphorothioate linkages (MS).
How much sgRNA do I need?
This table provides the approximate number of experiments that can be carried out for lipid transfection methods at the recommended sgRNA working concentration (25nM) in various plate/well formats. Calculations do not account for pipetting errors.
| sgRNA nmol | 96-well plate 100 µL reaction volume | 24-well plate 500 µL reaction volume | 12-well plate 1000 µL reaction volume | 6-well plate 2500 µL reaction volume |
|---|---|---|---|---|
| 2 | 800 | 160 | 80 | 32 |
| 5 | 2000 | 400 | 200 | 80 |
| 10 | 4000 | 800 | 400 | 160 |
- R. Barrangou, A. Birmingham, et. al. Advances in CRISPR-Cas9 genome engineering: lessons learned from RNA interference. Nucleic Acids Research, 43(7) 3407-3419 (2015)
- M.L. Kelley, Ž. Strezoska, et al. Versatility of chemically synthesized guide RNAs for CRISPR-Cas9 genome editing. J. Biotechnol. 233, 74–83 (2016). doi:10.1016/j.jbiotec.2016.06.011
- Basila, M., M. L. Kelley, et al.. Minimal 2’-O-methyl phosphorothioate linkage modification pattern of synthetic guide RNAs for increased stability and efficient CRISPR-Cas9 gene editing avoiding cellular toxicity. PLoS One. 12, e0188593 (2017). doi: 10.1371/journal.pone.0188593
- Anderson, E.M., A. Haupt, et al. Systematic analysis of CRISPR-Cas9 mismatch tolerance reveals low levels of off-target activity. J. Biotechnol. 211, 56-65 (2015)
- He, K., E. Chou, et al. Conjugation and evaluation of triazole-linked single guide RNA for CRISPR-Cas9 gene editing. ChemBioChem. DOI: 10.1002/cbic.201600320 (2016)
Application notes
Product data
Protocols
Safety data sheets
Selection guides
Technical manuals
-
CRISPR-Cas9 genome engineering with Cas9 nuclease expression plasmids and Edit-R synthetic RNA - Technical Manual
-
CRISPR-Cas9 genome engineering with Cas9 nuclease mRNA and Edit-R synthetic RNA - Technical Manual
-
CRISPR-Cas9 genome engineering with Cas9 nuclease protein and Edit-R synthetic guide RNA - Technical Manual
-
CRISPR-Cas9 genome engineering with lentiviral Cas9 particles and Edit-R synthetic guide RNA - Technical Manual
Related Products
A DNA-free option for Cas9 nuclease expression. Purified Cas9 nuclease protein with an enhanced hybrid NLS composition enabling more efficient nuclear delivery and gene editing. Suitable for a wide range of RNP workflows including nucleofection or co-transfection with Edit-R synthetic guide RNA for a completely DNA-free genome engineering system.
精製済みCas9 nuclease mRNAは、化学合成ガイドRNAとのコトランスフェクションで使用することにより完全にDNA-freeのゲノム編集を行えます。
ポジティブコントロールsgRNAを用いて、ゲノム編集の効率を最大化するための実験条件の検討、および最適化の確認のために使用します。ミスマッチ検出アッセイプライマーとのキット製品もご用意しています。
