相同組換え修復によるゲノム工学
相同組換えは、配列の相同性を利用したDNA配列情報の交換です。相同性指向性修復(Homology-directed repair: HDR)は相同組換えのプロセスであり、二本鎖切断(double-strand break: DSB)の正確な修復に必要な相同性を提供するためにDNAテンプレートが使われます。このテンプレートは、有糸分裂が完了する前に姉妹染色分体が利用可能になる細胞周期のS期後期やG2期に細胞内から得られます。さらに、外来性の修復テンプレートを細胞内に導入することも可能で、多くの場合、化学合成一本鎖DNAドナーオリゴまたはDNAドナープラスミドの形で、ゲノムに正確な変化を生じさせます。
HDRにおける一本鎖DNAドナーオリゴの使用
一本鎖DNAドナーオリゴは、内在性のゲノム標的領域にDNAの短い配列(SNP、アミノ酸置換、エピトープタグなど)を挿入または置換させるために細胞内に送達されます。化学合成DNAドナーオリゴを使用する利点は、ドナーテンプレートを作成するためのクローニングが不要であることと、ヌクレアーゼに対する耐性を高めるなどの様々な用途のために合成中にDNA修飾を加えることができることです。伝統的に、DNAドナーオリゴの使用に推奨される最大インサート長は50ヌクレオチド以下です。
HDR用DNAドナーオリゴの設計に関する考察
HDRを用いて正確なゲノム編集を行うためのDNAドナーオリゴを設計するためには、Cas9切断部位から目的とするHDR修飾部位(または挿入部位)の距離、特定の挿入部位に対するcrRNA、HDR修飾のタイプ、相同性アームの長さ、DNAドナーオリゴにCRISPR標的部位が存在する場合にはその破壊を考慮する必要があります。ここでは、CRISPR-Cas9システムで使用するDNAドナーオリゴの設計について説明します。一般的な原則は、どの部位特異的ヌクレアーゼにも適用できます。
挿入部位と切断部位の距離
哺乳動物細胞では、挿入部位とDSBの距離が10塩基以内のときにHDRの効率が最も高く、この距離が長くなるとHDRの効率は急速に低下します1)。
crRNA候補の同定
目的の挿入部位から20 nt上流と下流のDNA領域を、両鎖のPAM部位について評価し、3-5個の候補crRNAをDNAミスマッチ検出アッセイで切断効率についてテストする必要があります。
特定の挿入部位で最大のHDR効率を得るためのcrRNAの選択
HDR効率はDSBの非相同末端接合(non-homologous end joining: NHEJ)修復に比べて比較的低いため、可能な限り高いDNA切断効率を持つcrRNAを選択することが重要です。Cas9が生成するDSBの数が多ければ多いほど、DNAドナーオリゴによる修復のためにHDRメカニズムが利用できる切断部位が多くなります。
すべての挿入部位が周囲の20 nt以内にPAM配列を持つとは限らず、利用可能なPAM部位でのcrRNA効率評価が非常に低い場合もあります。このような状況では、ゲノムカット部位でHDRによる遺伝子改変を検出する可能性が最も高くなるように、挿入予定部位にできるだけ近い、遺伝子の標的領域に対して最も活性の高いcrRNAを選択します。繰り返しになりますが、HDRは挿入部位とDSBが10ヌクレオチド以内にある場合に最も効率が高くなります。
化学合成DNAドナーオリゴを設計するためのゲノム配列の入手
DNAドナーオリゴの設計を開始するには、アノテーションされたゲノム配列が必要です。ゲノム配列を選択する際には、DNAテンプレートが目的のHDR修飾部位を正しく修復するように、ゲノム配列(イントロンとエクソン)を検索することが不可欠です。.
化学合成DNAドナーオリゴのホモロジーアームの長さ
ヌクレオチドの置換やエピトープタグの挿入のためにDNAドナーオリゴを設計する場合2)、相同性アームは目的のHDR修飾を挟み、HDR修飾が行われるべき部位に特異的である必要があります(Figure 1)。ドナーオリゴの各相同性アームの長さは、短いもの(30-40 nt)から長いもの(70-90 nt)まで様々であり、通常は目的の挿入部位を中心に対称的な長さです3,4)。一本鎖DNAドナーオリゴを使用する場合、5'および3' 相同性アームともに30-40ヌクレオチドを使用することを推奨します5)。DNAオリゴの両端に2つのホスホロチオエートを付加することで、ホスホロチオエートを付加していないDNAドナーオリゴよりもHDR効率が向上することが実証されています3,5)。
HDR用DNAドナーオリゴの設計
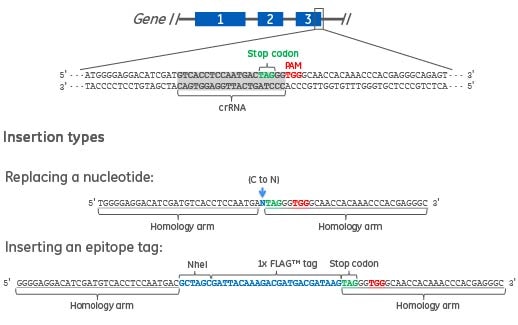
Figure 1. 図に2種類のDNAドナーオリゴを示します。1つ目は、標的遺伝子の単一ヌクレオチドを置換する61ヌクレオチドのDNAドナーオリゴです。標的遺伝子に特異的な相同性アーム(下線)が、変化させるヌクレオチド(青字)を挟んでいます。もう一つは、C末端エピトープタグ(例えばFLAG)とRFLP検出部位(例えばNheI)を作るための90ヌクレオチドDNAドナーオリゴです。遺伝子の標的に特異的な相同性アーム(下線)は、エピトープタグとNheI制限部位(青文字)を挟んでいます。
HDR遺伝子座のCRISPR-Cas9切断の回避
ドナーテンプレートを設計する際、CRISPR標的部位がドナーテンプレート内に存在する場合、CRISPR認識部位と切断部位(20塩基以下の標的配列とPAM)を破壊することが不可欠です。CRISPR標的部位を破壊しないと、修復後にCas9がHDR編集遺伝子座を切断してしまいます。これは、ドナーテンプレートに以下の変更を加えることで回避できます(Figure 2)。 (1) ドナーオリゴを、希望するDNA改変部位が20ヌクレオチドの標的配列を分割するようにデザインします。ドナーオリゴの2つのホモロジーアームが意図したDNA改変部位を取り囲みます。または(2)PAMまたは標的領域にヌクレオチド変化を作り、HDR編集部位でのCas9標的切断を防ぎます。HDR遺伝子座のCas9切断を回避するには、設計オプション(1)または(2)のどちらか一方が必要であり、両方は必要ありません。
HDRを介した編集後のCRISPR-Cas9切断を回避するためのドナーオリゴ設計戦略
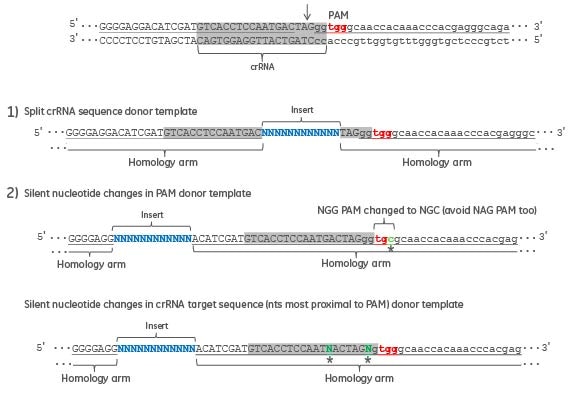
Figure 2. 図は、ゲノム配列のエクソン配列を大文字で、イントロン配列を小文字で、crRNA標的配列を灰色で示しています。HDRによって目的の配列を挿入した後、CRISPR-Cas9切断とNHEJ修復が再び起こることを回避するドナーオリゴの設計戦略を示しています。
制限酵素によるHDRの検出
制限酵素認識配列を挿入する、または挿入部近傍のヌクレオチドを変化させて制限酵素部位を作ることにより、制限酵素部位を含めることが望ましいとされています。これにより、制限断片長多型(restriction fragment length polymorphism: RFLP)のようなPCRベースの検出アッセイによるHDR成功の検出が容易になり、新たに導入された制限酵素部位でPCR産物が切断されます。
References
- B. Elliott, C. Richardson, et al., Gene conversion tracts from double-strand break repair in mammalian cells. Mol. Cell Biol. 18(1), 93-101 (1998)
- Bill Brizzard., Epitope tagging Biotechniques. 44(5), 693-5 (2008)
- JB. Renaud, C. Boix, et al., Improved genome editing efficiency and flexibility using modified oligonucleotides with TALEN and CRISPR-Cas9 nucleases. Cell Rep. 14(9), 2263-72 (2016)
- FA. Ran, PD. Hsu, et al., Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity.Cell. 154(6), 1380-9 (2013)
- J. Schiel, E. Chou, et al., Homology-directed repair with Dharmacon™ Edit-R™ CRISPR-Cas9 reagents and single-strand DNA oligos.Dharmacon 2015
Order Products
カスタムDNA合成
便利なオンラインツールを使って、実験デザインに合ったカスタムDNAオリゴを設計し、ご注文ください。
CRISPR-Cas9ゲノム編集
信頼性の高いゲノムエンジニアリングのための最適化された試薬
CRISPR Design Tool
ゲノム編集アプリケーションのためのご研究に合うカスタムのcrRNAおよびsgRNAをデザインできます。
HDRドナーデザイナーワークフロー
30種以上の生物種におけるHDRを介したゲノム編集のためのカスタムドナーオリゴまたはプラスミドを設計し、注文することができます。
Helpful Resources
Read protocol
Edit-R Cas9 mRNA、crRNA:tracrRNAおよびHDRドナーテンプレートのトランスフェクションプロトコール
Read app note
Dharmacon Edit-R CRISPR-Cas9試薬と一本鎖DNAオリゴによる相同組換え修復
View poster
一本鎖DNAオリゴと化学合成crRNAおよびtracrRNAを用いた相同組換え修復
Read app note
CRISPR-Cas9遺伝子工学ワークフロー:Edit-R Cas9と化学合成crRNAおよびtracrRNAを用いた機能的ノックアウトの作製
Read app note
ゲノムエンジニアリングのためのDharmacon Edit-R Cas9 Nuclease mRNA、化学合成crRNAおよびtracrRNAを用いたゼブラフィッシュ胚のマイクロインジェクション
