HDRによるゲノム工学
相同組換え修復(Homology-directed repair: HDR)とは、DNA二本鎖切断(double-strand break: DSB)がDNAテンプレートを用いて相同組換えにより修復されるプロセスです。このテンプレートは、細胞周期のS期後期あるいはG2期、すなわち有糸分裂が完了する前に姉妹染色分体が利用可能な時期に細胞内から得られます。さらに、外来性の修復テンプレートを細胞内に導入することも可能で、多くの場合、化学合成一本鎖DNAドナーオリゴまたはドナープラスミドの形で、ゲノムに正確な変化を生じさせます。
HDRにおけるプラスミドドナーの使用
プラスミドドナー修復テンプレートは、内在性ゲノム標的領域にDNAの大きな配列(蛍光レポーターのノックインまたは遺伝子全体の置換)を挿入または変更するために細胞内に送達されます。
プラスミドドナーを用いたHDRのためのデザイン上の考慮点
HDRを用いた正確なゲノム編集のためにDNAドナープラスミドを設計するには、以下の点を考慮してください。
- 目的とするHDR挿入部位(または改変部位)のCas9切断部位からの距離
- 特定の挿入部位付近のgRNA効率
- 相同性アームの長さ
- CRISPR標的部位がプラスミドドナー修復テンプレートに存在する場合の破壊
ここでは、CRISPR-Cas9システムで使用するHDRプラスミドドナーの設計について説明します。一般的な原理は、どの部位特異的ヌクレアーゼにも適用できます。
挿入部位と切断部位の距離
哺乳動物細胞では、挿入部位とDSBが10塩基以内の距離にあるとき、HDRの効率が最も高くなります。この距離が長くなると、HDR効率は急速に低下します1)。
特定の挿入部位で最大のHDR効率を得るためのgRNAの選択
HDR効率はDSBの非相同末端接合(non-homologous end joining: NHEJ)修復に比べて比較的低いため、可能な限り高いDNA切断効率を持つgRNAを選択することが重要です。NHEJを介する効率は少なくとも25%が推奨されます。これは、正確なHDRを介した修復は、利用可能なDSBのほんの一部でしか起こらないためです。
さらに、全ての挿入部位が周囲 20 nt に PAM 配列を持つとは限らず、利用可能な PAM 部位で評価された gRNA 効率は非常に低いかもしれません。このような状況では、挿入予定部位にできるだけ近い、最も活性の高いgRNAを選択します。繰り返しになりますが、HDRは挿入部位とDSBが10塩基以内にある場合に最も効率が高くなることにご注意ください1)。
プラスミドドナーの相同性アームの長さ
プラスミドドナーのホモロジーアームの長さは、通常500-1000 ntの範囲です2-4)。
プラスミドドナーのデザイン
以下は、HDR Donor Designerを使用する際に自動的に組み込まれる基本的な設計上の考慮事項です。
ゲノム配列の取得
プラスミドドナーの設計を開始するには、アノテーションされたゲノム配列が必要です。ゲノム配列を選択する際には、DNAテンプレートが目的のHDR修飾部位を正しく修復するように、例えばmRNA配列ではなく、ゲノム配列(イントロンとエクソン)を取得することが重要です。
挿入位置の決定
挿入位置は標的タンパク質の内在性遺伝子内にあるべきです。理想的な挿入部位は、N末端挿入の場合は開始コドンの正確な後、逆にC末端挿入の場合は停止コドンの直前です。
相同性アームを増幅するためのプライマー設計
目的とする遺伝子標的の相同性アームを増幅するPCRプライマーを設計する場合、選択された挿入部位が挿入隣接プライマーの開始位置を示します(Figure 1)。対応するプライマーペアが挿入部位から離れる距離によって、それぞれの相同性アームの長さが決まります。さらに、プラスミドドナーへの適切なライゲーションフリーアセンブリーのために、アダプター配列がプライマーの遺伝子特異的部分に付加されます(Figure 1、赤またはオレンジ長方形)。
標的遺伝子とインフレームのmKate2ドナープラスミドを作製するための相同性アームプライマーデザイン
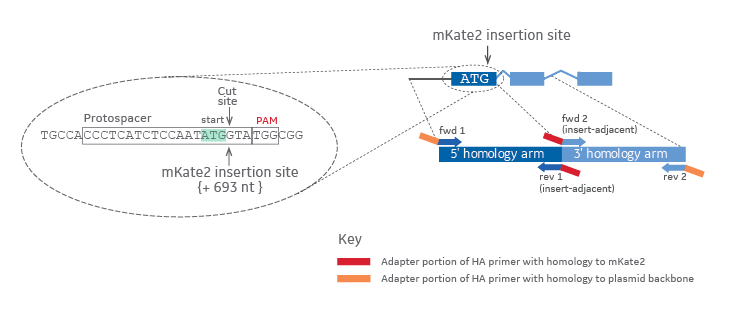
Figure 1. gRNAターゲットまたはプロトスペーサーとPAM配列は、左の黒い点線楕円内に拡大しました。切断部位と挿入部位を矢印で、開始コドン(ATG)は薄緑色でハイライトされています。右側の色はDNAのオリジンを示しています(濃青: 5'相同性アーム、薄青:3'相同性アーム、赤:mKate2蛍光レポーター配列、オレンジ:プラスミドバックボーン)。
HDR後のCRISPR-Cas9による遺伝子座切断の回避
ドナープラスミドを設計する際、CRISPR標的部位がドナーテンプレート内に存在する場合には、それを破壊することが不可欠です。CRISPR標的部位を破壊しないと、修復後にCas9がHDRで編集した遺伝子座を切断してしまいます。これは、ドナーテンプレートに以下の変更を加えることで回避できます(Figure 2)。 (1) DNA挿入が20ヌクレオチドのCRISPR標的配列を分割するようにドナープラスミドを設計します。または(2) 適切な遺伝子特異的挿入隣接相同性アームプライマーに1つ以上のヌクレオチド変化を導入することにより、PAMまたはCRISPR標的領域にサイレント変異を導入します。
ドナープラスミドを用いたHDRによる挿入後のCRISPR-Cas9切断を回避するデザイン戦略
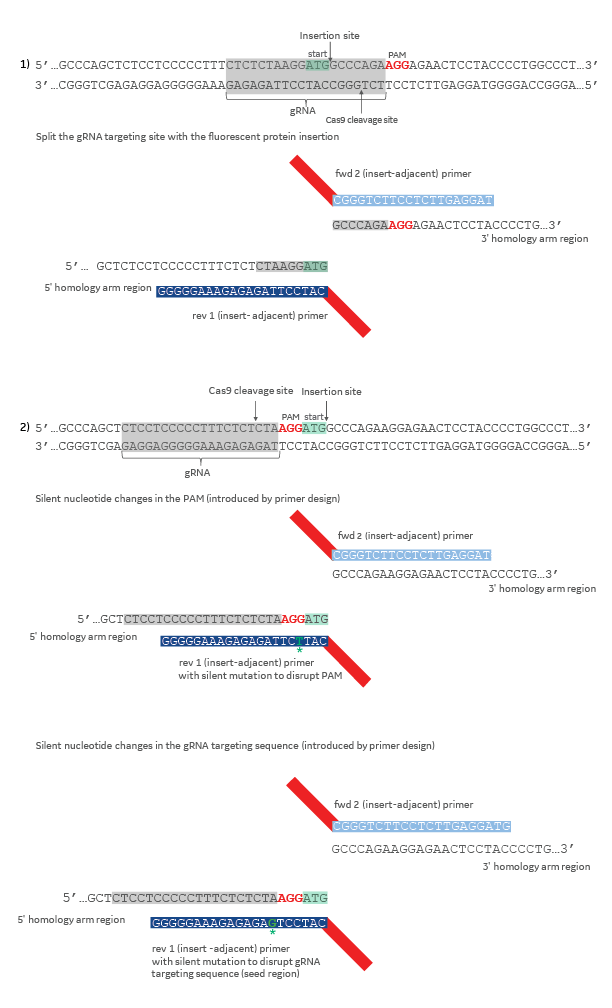
Figure 2. gRNA標的配列は灰色で、PAM配列は赤色で、開始コドン(ATG)は薄緑色でハイライトされています。プライマー配列のサイレント変異は濃緑色で着色し、アスタリスクで示しています。図中の色はDNAの起源を示します(濃い青:5'相同性、薄青:3'相同性、赤:mKate2蛍光レポーター配列)。
References
- B. Elliott, C. Richardson, et al. Gene conversion tracts from double-strand break repair in mammalian cells. Mol. Cell. Biol. 18(1), 93-101 (1998)
- R. Mahen, B. Koch, et al. Comparative assessment of fluorescent transgene methods for quantitative imaging in human cells. Mol. Biol. Cell. 25(22), 3610–3618 (2014)
- F. Merkle, W. Neuhausser, et al. Efficient CRISPR-Cas9-Mediated Generation of Knockin Human Pluripotent Stem Cells Lacking Undesired Mutations at the Targeted Locus. Cell Reports. 11(6), 875-883 (2015)
- M. Ratz, I. Testa, et al. CRISPR /Cas9-mediated endogenous protein tagging for RESOLFT super-resolution microscopy of living human cells. Scientific Reports. 5(9592),1-6 (2015)
Order Products
HDR Donor Designer
正確なCRISPR-Cas9ゲノム編集のためのドナーテンプレートを迅速かつ容易に設計し、注文することができます。
Edit-R HDRプラスミドドナーキット
HDR用プラスミドドナーを迅速かつ容易に組み立てることができます。
Edit-R HDRプラスミドドナーコンポーネント
HDRドナープラスミドを迅速かつ効率的に構築します。
Edit-R HDRプラスミドドナープライマー
Edit-R Plasmidドナーキット用PCRコンポーネント
CRISPR Design Tool
ゲノム編集アプリケーション用のカスタムcrRNAおよびsgRNAをデザインできます。
CRISPR-Cas9ゲノム編集
信頼性の高いゲノムエンジニアリングのための最適化された試薬
$name
Read app note
Dharmacon Edit-R CRISPR-Cas9試薬を用いた相同組換え修復による内在性遺伝子の蛍光のタグ付け。Dharmacon Edit-R Cas9 mRNA、化学合成crRNA:tracrRNA、およびEGFPドナープラスミドを用いた内在性遺伝子タギングの実行を説明したアプリケーションノート。
Read app note
エレクトロポレーション法を用いた、トランスフェクションが困難なK-652細胞におけるHDR試薬の送達を最適化する推奨プロトコール。
